

Herausgeber | Wissenschaftler haben nach effizienten Möglichkeiten gesucht, die Passung zwischen diesen „Schlüsseln“ und „Schlössern“ oder Protein-Ligand-Wechselwirkungen vorherzusagen.
Herkömmliche datengesteuerte Methoden verfallen jedoch oft in das „Auswendiglernen“, bei dem sie sich Liganden- und Protein-Trainingsdaten merken, anstatt die Wechselwirkungen zwischen ihnen wirklich zu lernen.
Kürzlich hat ein Forschungsteam der Zhejiang-Universität und der Chinesischen Akademie der Wissenschaften eine neue Bewertungsmethode namens EquiScore vorgeschlagen, die heterogene graphische neuronale Netze verwendet, um physikalisches Vorwissen zu integrieren und Protein-Ligand-Wechselwirkungen im Gleichungstransformationsraum zu charakterisieren.
EquiScore wird anhand eines neuen Datensatzes trainiert, der mithilfe mehrerer Datenerweiterungsstrategien und eines strengen Redundanzbeseitigungsschemas erstellt wurde.
Bei zwei großen externen Testreihen konnte sich EquiScore im Vergleich zu 21 anderen Methoden durchsetzen. Wenn EquiScore mit verschiedenen Docking-Methoden verwendet wird, können die Screening-Funktionen dieser Docking-Methoden effektiv verbessert werden. EquiScore schnitt auch bei der Bewertung der Aktivität einer Reihe strukturell ähnlicher Substanzen gut ab und demonstrierte damit sein Potenzial als Leitfaden für die Optimierung von Leitverbindungen.
Abschließend wurden verschiedene Interpretierbarkeitsstufen von EquiScore untersucht, die möglicherweise weitere Erkenntnisse für das strukturbasierte Arzneimitteldesign liefern.
Die Studie trug den Titel „
Generische Protein-Ligand-Interaktionsbewertung durch Integration von physikalischem Vorwissen und Datenerweiterungsmodellierung“ und wurde am 6. Juni 2024 in „Nature Machine Intelligence“ veröffentlicht.
 Link zum Papier:
Link zum Papier:
Mit der Explosion experimenteller Daten zur Protein-Ligand-Interaktion haben auf maschinellem Lernen basierende Bewertungsmethoden erhebliche Fortschritte gemacht.
Die zunehmende Kapazität maschineller Lernmodelle ermöglicht es ihnen, sich den gesamten Trainingsdatensatz zu merken. Gleichzeitig führen Probleme mit Datenlecks zwischen Trainingsdaten und Testdaten zu einer zu optimistischen Bewertung der Fähigkeiten dieser Modelle ist die effektive Integration relevanter physikalischer Vorinformationen zu Ligand-Protein-Wechselwirkungen.
Die Architektur von EquiScoreZweitens wird durch die Einführung neuer Arten von Knoten und Kanten und eines informationsbewussten Aufmerksamkeitsmechanismus ein heterogener Graph vorgeschlagen, der frühere Informationen über physikalische intermolekulare Wechselwirkungen integrieren kann.
Abbildung: Pipeline zum Erstellen eines PDBscreen-Datensatzes. (Quelle: Papier) EquiScore ist ein binäres Klassifizierungsmodell, das das Bindungspotenzial zwischen Proteinen und Liganden durch Eingabe einer isomorphen Karte bewertet, die aus Proteintaschenregionen und Liganden erstellt wurde.
EquiScore ist ein binäres Klassifizierungsmodell, das das Bindungspotenzial zwischen Proteinen und Liganden durch Eingabe einer isomorphen Karte bewertet, die aus Proteintaschenregionen und Liganden erstellt wurde.
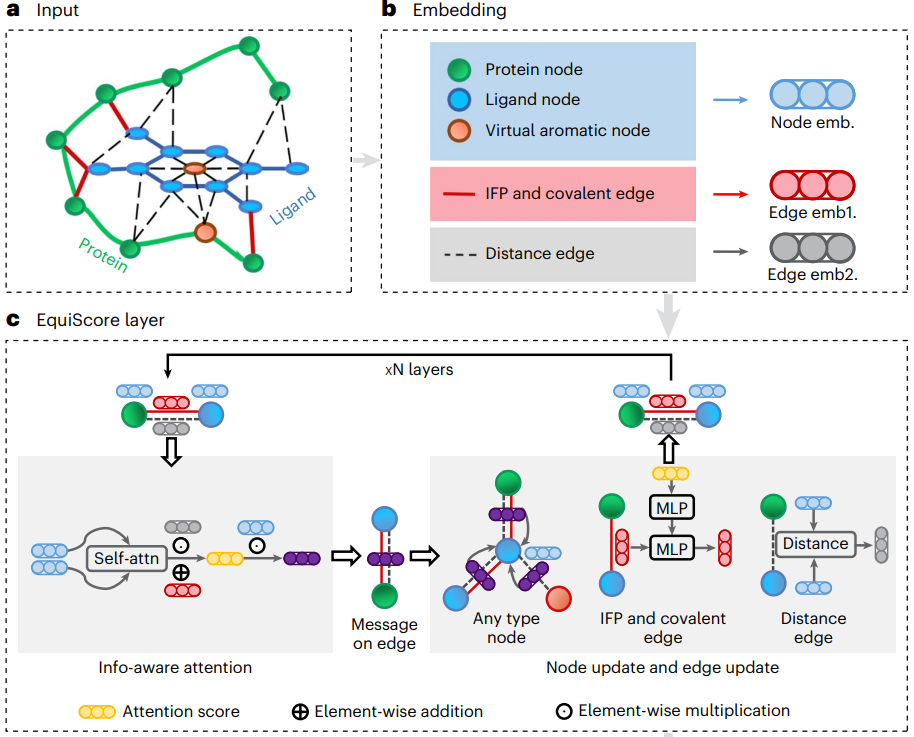
 Im ersten Schritt entwarfen die Forscher ein heterogenes Diagrammkonstruktionsschema. Zusätzlich zur Abstraktion bestehender Atome in Knoten wird für jeden aromatischen Ring ein virtueller Knoten hinzugefügt, der auf dem Vorwissen von Experten basiert, um das aromatische System besser darzustellen. Um Kanten zu bilden, werden zwischen Knoten geometrische abstandsbasierte Kanten (
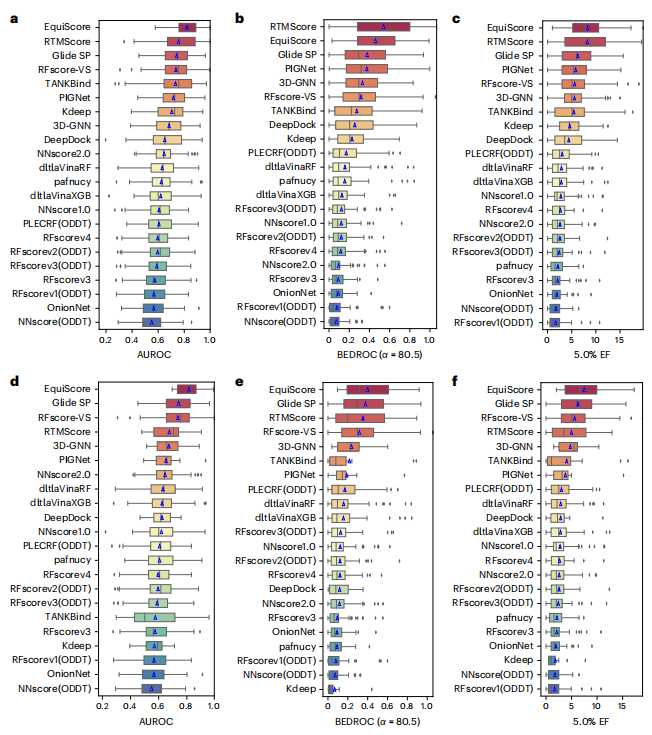
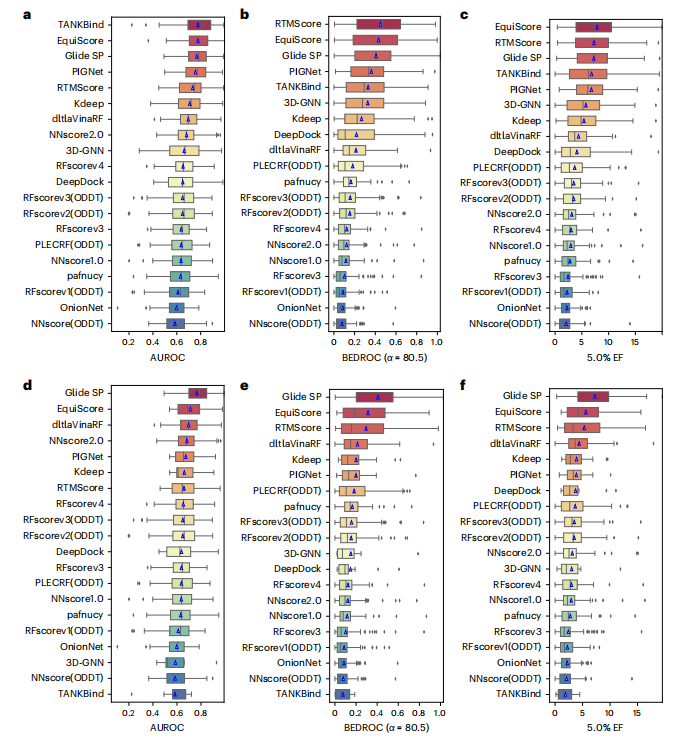
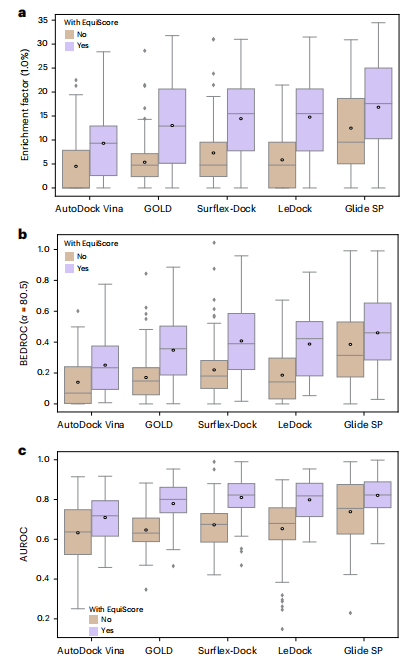
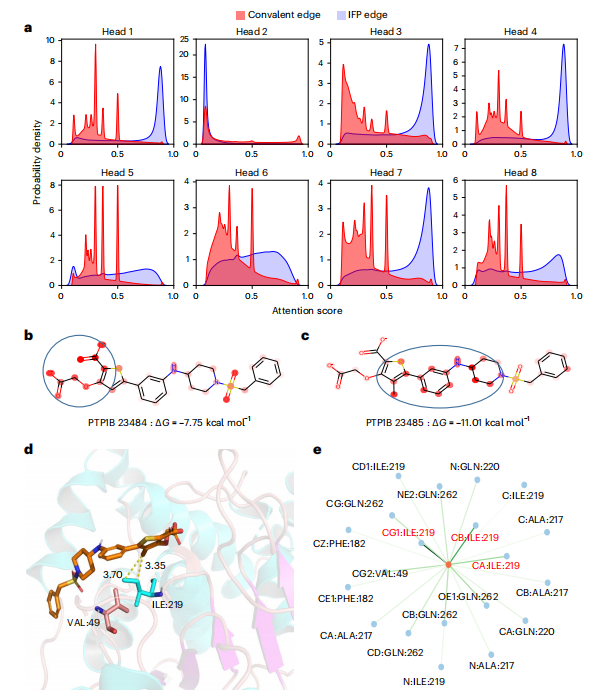
Im ersten Schritt entwarfen die Forscher ein heterogenes Diagrammkonstruktionsschema. Zusätzlich zur Abstraktion bestehender Atome in Knoten wird für jeden aromatischen Ring ein virtueller Knoten hinzugefügt, der auf dem Vorwissen von Experten basiert, um das aromatische System besser darzustellen. Um Kanten zu bilden, werden zwischen Knoten geometrische abstandsbasierte Kanten (strukturell) erstellt. Die Forscher fügten EStrukturen auch eine Klasse von Kanten hinzu, die auf ProLIF-berechneten empirischen Protein-Ligand-Interaktionskomponenten (IFPs) basieren, um a priori physikalisches Wissen über intermolekulare Wechselwirkungen einzubeziehen. Im zweiten Schritt wird eine Einbettungsschicht verwendet, um eine latente Darstellung jedes Kanten- und Knotentyps im heterogenen Diagramm zu erhalten. Dieses Schema kann weitere neue Knoten und Kanten mit klarer physikalischer Bedeutung einführen und kann nahtlos in nachfolgende Darstellungslernmodule integriert werden. Um die induktive Verzerrung der Informationen von verschiedenen Knoten und Kanten voll auszunutzen und gleichzeitig die gleiche Varianz des Modells sicherzustellen, besteht die EquiScore-Schicht aus drei Untermodulen: Informationsbewusstes Aufmerksamkeitsmodul, Knotenaktualisierungsmodul und Kantenaktualisierungsmodul. Das informationsbewusste Aufmerksamkeitsmodul kann Interaktionen aus verschiedenen Informationen interpretieren, darunter (1) äquivariante geometrische Informationen, (2) chemische Strukturinformationen und (3) empirische Protein-Ligand-Interaktionskomponenten. Die Forscher bewerteten die Leistung des generierten EquiScore-Modells. Im Szenario des virtuellen Screenings (VS) erzielte EquiScore im Vergleich zu 21 bestehenden Bewertungsmethoden für unsichtbare Proteine in zwei externen Datensätzen, DEKOIS2.0 und DUD-E, durchweg Spitzenplätze. Im Lead-Optimierungsszenario zeigte EquiScore nur bei acht verschiedenen Methoden eine geringere Rankingfähigkeit im Vergleich zu FEP+. Wenn man bedenkt, dass FEP+-Berechnungen deutlich höhere Rechenkosten erfordern, zeigt EquiScore einen ausgeglicheneren Vorteil zwischen Geschwindigkeit und Genauigkeit. Darüber hinaus wurde festgestellt, dass EquiScore starke Neubewertungsfunktionen aufweist, wenn es auf Posen angewendet wird, die durch verschiedene Docking-Methoden generiert wurden, und dass die Verwendung der EquiScore-Neubewertung die VS-Leistung für alle Bewertungsmethoden verbessern kann. Abschließend analysierten die Forscher die Interpretierbarkeit des Modells und stellten fest, dass das Modell wichtige intermolekulare Wechselwirkungen erfassen konnte, was die Rationalität des Modells bewies und nützliche Hinweise für ein rationales Arzneimitteldesign lieferte. Robuste Vorhersagen von Protein-Ligand-Wechselwirkungen werden wertvolle Möglichkeiten bieten, die Biologie von Proteinen zu verstehen und ihren Einfluss auf zukünftige Arzneimitteltherapien zu bestimmen. EquiScore wird zu einem besseren Verständnis der menschlichen Gesundheit und Krankheit beitragen und die Entdeckung neuer Medikamente erleichtern. Bewertung der Modellleistung
Das obige ist der detaillierte Inhalt von„KI+Physik-Vorkenntnisse', allgemeine Bewertungsmethode für Protein-Ligand-Interaktionen der Zhejiang-Universität und der Chinesischen Akademie der Wissenschaften, veröffentlicht im Nature-Unterjournal. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
 So verwenden Sie Etikettenetiketten
So verwenden Sie Etikettenetiketten
 Was bedeutet WLAN deaktiviert?
Was bedeutet WLAN deaktiviert?
 So verbinden Sie ASP mit dem Zugriff auf die Datenbank
So verbinden Sie ASP mit dem Zugriff auf die Datenbank
 Maßnahmen zur Speicherung der Datenverschlüsselung
Maßnahmen zur Speicherung der Datenverschlüsselung
 In Word gibt es eine zusätzliche leere Seite, die ich nicht löschen kann.
In Word gibt es eine zusätzliche leere Seite, die ich nicht löschen kann.
 So verwenden Sie die Achsenfunktion in Matlab
So verwenden Sie die Achsenfunktion in Matlab
 Was sind die gängigen Linux-Systeme?
Was sind die gängigen Linux-Systeme?
 Was ist ein Festplattenkontingent?
Was ist ein Festplattenkontingent?




