

Deep-Learning-Methoden haben zu erheblichen Fortschritten bei der Einzelzustandsvorhersage biomolekularer Strukturen geführt. Die Funktionalität von Biomolekülen hängt jedoch von der Bandbreite der Konformationen ab, die sie annehmen können. Dies gilt insbesondere für Peptide, eine Klasse hochflexibler Moleküle, die an einer Vielzahl biologischer Prozesse beteiligt sind und als Therapeutika von großem Interesse sind.
Philip M. Kim und Osama Abdin von der University of Toronto haben PepFlow entwickelt, ein übertragbares generatives Modell, das die Probennahme aller Atome direkt aus dem zulässigen Konformationsraum eines Eingabepeptids ermöglicht. Die Forscher trainierten das Modell in einem Diffusionsrahmen und verwendeten dann einen äquivalenten Fluss für die Konformationsprobenahme.
Um die unerschwinglichen Kosten einer verallgemeinerten Gesamtatommodellierung zu überwinden, modularisierten sie den Generierungsprozess und integrierten Supernetzwerke, um sequenzspezifische Netzwerkparameter vorherzusagen. PepFlow sagt Peptidstrukturen genau voraus und reproduziert effizient experimentelle Peptidsammlungen in einem Bruchteil der Laufzeit herkömmlicher Methoden. PepFlow kann auch zur Probenahme von Konformationen verwendet werden, die Einschränkungen wie Makrocyclisierung erfüllen.
„Bisher konnten wir die vollständige Konformation eines Peptids nicht simulieren.“ Osama Abdin, der Erstautor der Studie, sagte: „PepFlow nutzt Deep Learning, um die genaue Konformation des Peptids in wenigen Minuten zu erfassen Das Modell hat das Potenzial, vom Design „Peptide als Bindemittel zur Steuerung der Arzneimittelentwicklung“ verwendet zu werden. 2024.
 Protein-Peptid-Wechselwirkungen
Protein-Peptid-Wechselwirkungen
Peptide haben eine hohe Spezifität und ein geringes Toxizitätsrisiko.
Philip M. Kim erklärte, dass sich das PepFlow-Modell auf Peptide konzentriert, da es sich um wichtige Biomoleküle mit natürlichen Aktivitäten handelt und ihre Konformationen simuliert werden müssen, um die Funktion zu verstehen.
Abbildung: Schematische Darstellung der PepFlow-Architektur. (Quelle: Papier) 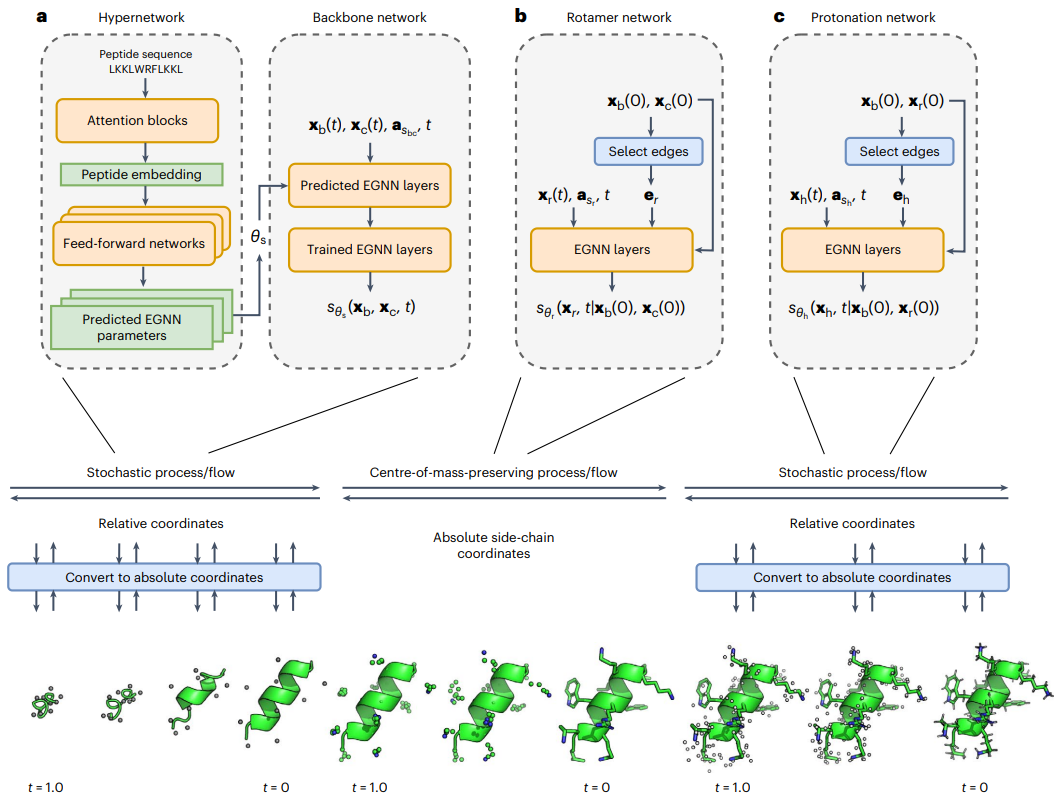
PepFlow verfügt über leistungsstarke Funktionen zur Vorhersage von Singulett-Peptidstrukturen und Sammlungen kurzer linearer Motive (SLiM) und kann Peptidstrukturen unter Einschränkungen wie Makrozyklisierung durch Konformationssuchen im latenten Raum modellieren.
Dieses Modell erweitert die Fähigkeit von AlphaFold, dem führenden Google Deepmind AI-System, Proteinstrukturen vorherzusagen. PepFlow übertrifft AlphaFold2, indem es eine Reihe von Konformationen für ein bestimmtes Peptid generiert. AlphaFold2 wurde nicht zur Lösung dieses Problems entwickelt.
Was PepFlow auszeichnet, ist die technologische Innovation dahinter. Es handelt sich beispielsweise um ein verallgemeinertes Modell, das vom Boltzmann-Generator inspiriert ist, einem sehr fortschrittlichen physikbasierten Modell für maschinelles Lernen.
„Die Verwendung der PepFlow-Modellierung kann Einblick in den wahren Energiestatus von Peptiden geben.“ Abdin sagte: „Die Entwicklung von PepFlow hat nur einen Monat gedauert, aber es lohnt sich, auf die nächste Grenze zu gehen.“ Darüber hinaus können Modelle, die nur eine Struktur eines Peptids vorhersagen, das Andocken und Design von Peptiden verbessern. Peptid-Docking-Methoden beginnen typischerweise mit einer Bibliothek von Peptidkonformationen, die an das Protein von Interesse angedockt sind. Eine präzisere Erzeugung von Peptidensembles könnte diesen Prozess verbessern.
PepFlow kann auch verwendet werden, um die Neigung verschiedener Sequenzen zur Annahme von Konformationen an Zielprotein-Protein-Schnittstellen zu bewerten, was wiederum zur Entwicklung hemmender Peptide verwendet werden kann.
Abbildung: Vergleich von Ensembles, die durch PepFlow- und Molekulardynamiksimulationen erzeugt wurden. (Quelle: Papier)Obwohl PepFlow AlphaFold2 verbessert, weist es auch Einschränkungen auf, da dies nur die erste Version des Modells ist. 
PepFlow hat einen erheblichen Nachteil: Im Gegensatz zum Boltzmann-Generator fehlt PepFlow die Möglichkeit, die generierten Stichproben neu zu gewichten, um eine genaue Boltzmann-Verteilung zu erreichen.
Während PepFlow in der Lage ist, Wahrscheinlichkeitsberechnungen für generierte Stichproben durchzuführen, erfordern nachvollziehbare Berechnungen die Verwendung stochastischer Schätzer, die den berechneten Werten Rauschen hinzufügen. Darüber hinaus generiert PepFlow gelegentlich hochenergetische Proben, ist jedoch nicht in der Lage, die gesamte Energielandschaft zu erfassen, die in Molekulardynamiksimulationen beobachtet wird.
Eine mögliche Möglichkeit zur Verbesserung von PepFlow besteht darin, das entwickelte Modell auf andere Sampling-Frameworks zu übertragen. In den bedingten Einstellungen wurde ein normalisierter Fluss verwendet und es wurden verschiedene Stichprobenmethoden verwendet, um die Stichprobe aus der Boltzmann-Verteilung zu erleichtern.
Das kürzlich von der akademischen Gemeinschaft entwickelte Flow-Matching-Paradigma dient darüber hinaus als Alternative zum simulierungsfreien Training kontinuierlicher normalisierter Strömungsmodelle. Flow Matching wurde effektiv für die strukturelle Probenahme verschiedener Moleküle, einschließlich kleiner Moleküle und Proteine, eingesetzt und kann möglicherweise zur Erweiterung der Wirksamkeit des PepFlow-Frameworks verwendet werden.
Zusammenfassend ist PepFlow so konzipiert, dass es leicht erweiterbar ist, um andere Faktoren, neue Informationen und potenzielle Verwendungsmöglichkeiten zu berücksichtigen.
Bereits in der ersten Version ist PepFlow ein umfassendes und wirksames Modell mit Potenzial für die Weiterentwicklung von Therapeutika, die auf der Bindung von Peptiden beruhen, um biologische Prozesse zu aktivieren oder zu hemmen.
Link zum Papier: https://www.nature.com/articles/s42256-024-00860-4
Verwandte Berichte: https://phys.org/news/2024-06-deep-outperforms-google-ai -peptide.html
Das obige ist der detaillierte Inhalt vonNature-Unterjournal, besser als AlphaFold, All-Atom-Sampling, eine KI-Methode zur Vorhersage der Peptidstruktur. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
 So erhalten Sie die Eingabenummer in Java
So erhalten Sie die Eingabenummer in Java
 So öffnen Sie eine NRG-Datei
So öffnen Sie eine NRG-Datei
 Historisches Bitcoin-Preisdiagramm
Historisches Bitcoin-Preisdiagramm
 So deduplizieren Sie eine Datenbank in MySQL
So deduplizieren Sie eine Datenbank in MySQL
 In der Sprache C gibt es mehrere Ausgabe- und Eingabefunktionen
In der Sprache C gibt es mehrere Ausgabe- und Eingabefunktionen
 Google Earth kann keine Verbindung zur Serverlösung herstellen
Google Earth kann keine Verbindung zur Serverlösung herstellen
 So reparieren Sie eine SQL-Datenbank
So reparieren Sie eine SQL-Datenbank
 Was sind die sieben Prinzipien der PHP-Codespezifikationen?
Was sind die sieben Prinzipien der PHP-Codespezifikationen?
 So führen Sie ein Shell-Skript aus
So führen Sie ein Shell-Skript aus




