
Bei der Arzneimittelentwicklung ist es entscheidend, die Bindungsaffinität und funktionelle Wirkung von kleinen Molekülliganden auf Proteine zu bestimmen. Aktuelle Computermethoden können diese Protein-Ligand-Wechselwirkungseigenschaften vorhersagen, aber ohne hochauflösende Proteinstrukturen geht häufig die Genauigkeit verloren und funktionelle Auswirkungen können nicht vorhergesagt werden.
Forscher der Monash University und der Griffith University haben PSICHIC (PhySIcoCHhemICal Graph Neural Network) entwickelt, ein Framework, das physikochemische Einschränkungen direkt aus Sequenzen von Daten kombiniert, die Interaktionsfingerabdrücke dekodieren. Dies ermöglicht es PSICHIC, die Mechanismen hinter Protein-Ligand-Wechselwirkungen zu entschlüsseln und so Genauigkeit und Interpretierbarkeit auf dem neuesten Stand der Technik zu erreichen.
An den gleichen Protein-Ligand-Paaren ohne Strukturdaten trainiert, erbrachte PSICHIC bei der Vorhersage der Bindungsaffinität eine gleichwertige Leistung wie die führenden strukturbasierten Methoden oder übertraf diese sogar. Der interpretierbare Fingerabdruck von
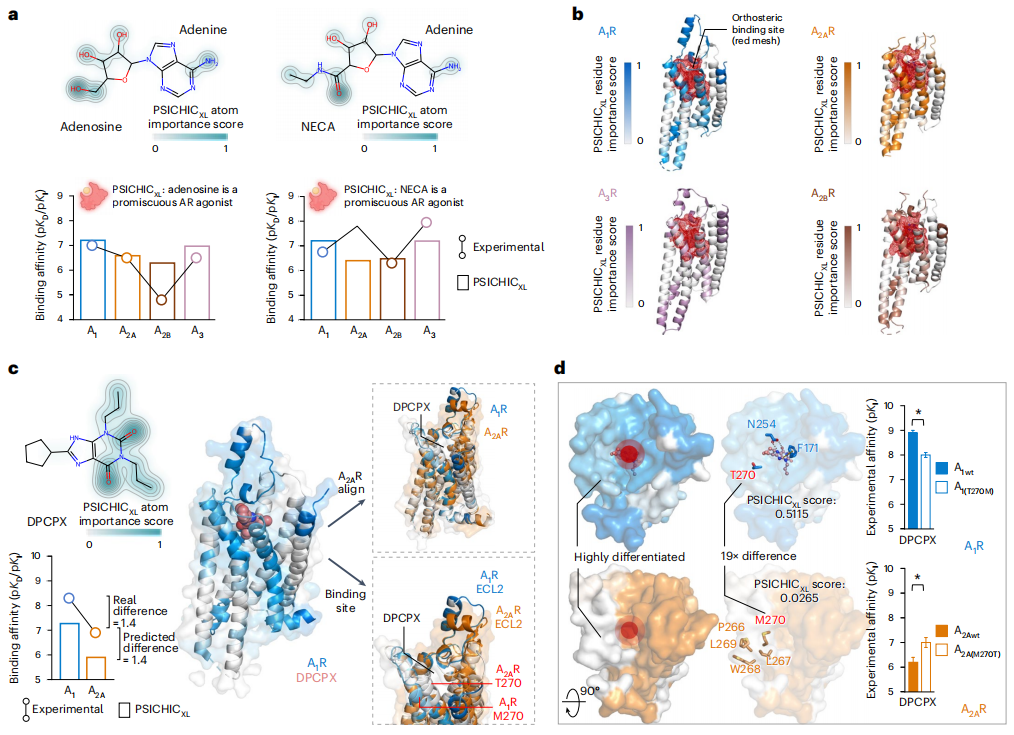
PSICHIC identifiziert Proteinreste und Ligandenatome, die an der Wechselwirkung beteiligt sind, und hilft dabei, die Selektivitätsdeterminanten von Protein-Ligand-Wechselwirkungen aufzudecken.
Die Studie trug den Titel „Physikochemisches graphisches neuronales Netzwerk zum Erlernen von Fingerabdrücken der Protein-Ligand-Interaktion aus Sequenzdaten“ und wurde am 17. Juni 2024 in „Nature Machine Intelligence“ veröffentlicht.

Im Prozess der Arzneimittelentdeckung ist das Verständnis der Bindungsaffinität und der funktionellen Wirkungen kleiner Molekülliganden auf Proteine von entscheidender Bedeutung, da die selektive Wechselwirkung des Liganden mit einem spezifischen Protein die erwartete Wirkung bestimmt der Droge.
Obwohl aktuelle Computermethoden in der Lage sind, Protein-Ligand-Wechselwirkungseigenschaften vorherzusagen, nimmt die Vorhersagegenauigkeit oft ab, wenn keine hochauflösenden Proteinstrukturen vorliegen, und es gibt auch Schwierigkeiten bei der Vorhersage funktioneller Effekte.
Obwohl sequenzbasierte Methoden größere Kosten- und Ressourcenvorteile bieten (z. B. erfordern sie keine teuren experimentellen Strukturbestimmungsprozesse), stehen diese Methoden häufig vor dem Problem übermäßiger Freiheitsgrade beim Mustervergleich, was leicht zu einer Überanpassung führen kann und begrenzte Generalisierungsmöglichkeiten, wodurch eine Leistungslücke im Vergleich zu struktur- oder zusammengesetzten Methoden entsteht.
Physical Chemistry Graph Neural Network
Ein Forschungsteam der Monash University und der Griffith University hat PSICHIC (Physical Chemistry Graph Neural Network) entwickelt, eine Methode zur direkten Dekodierung von Proteinliganden aus Sequenzdaten nach physikalischen und chemischen Prinzipien der Körperinteraktion Methode. Im Gegensatz zu früheren sequenzbasierten Modellen berücksichtigt PSICHIC auf einzigartige Weise physikalisch-chemische Einschränkungen, um eine Genauigkeit und Interpretierbarkeit auf dem neuesten Stand der Technik zu erreichen.
Als 2D-sequenzbasierte Methode generiert PSICHIC diese Einschränkungen und legt sie einem 2D-Diagramm durch die Anwendung eines Clustering-Algorithmus auf, wodurch PSICHIC sich in erster Linie an die rationalen zugrunde liegenden Muster anpassen kann, die Protein-Ligand-Wechselwirkungen während des Trainings bestimmen. Abbildung: PSICHIC-Übersicht (Quelle: Papier) Strukturbasierte und kompositbasierte Methoden der Kunst konkurrieren mit ihnen oder übertreffen sie sogar.
 Experimentelle Ergebnisse zu PDBBind v2016- und PDBBind v2020-Datensätzen zeigen, dass PSICHIC andere sequenzbasierte Methoden wie TransCPI, MolTrans und DrugBAN bei mehreren Indikatoren übertrifft.
Experimentelle Ergebnisse zu PDBBind v2016- und PDBBind v2020-Datensätzen zeigen, dass PSICHIC andere sequenzbasierte Methoden wie TransCPI, MolTrans und DrugBAN bei mehreren Indikatoren übertrifft.
Leistungsstatistische Zusammenfassung der Protein-Ligand-Bindungsaffinitätsvorhersagen für die Benchmarks PDBBind v2016 und PDBBind v2020. (Quelle: Papier)
Im Einzelnen:PSICHIC weist einen geringeren Vorhersagefehler und einen höheren Korrelationsindex auf, insbesondere im Hinblick auf Vorhersagegenauigkeit und Generalisierungsfähigkeit. 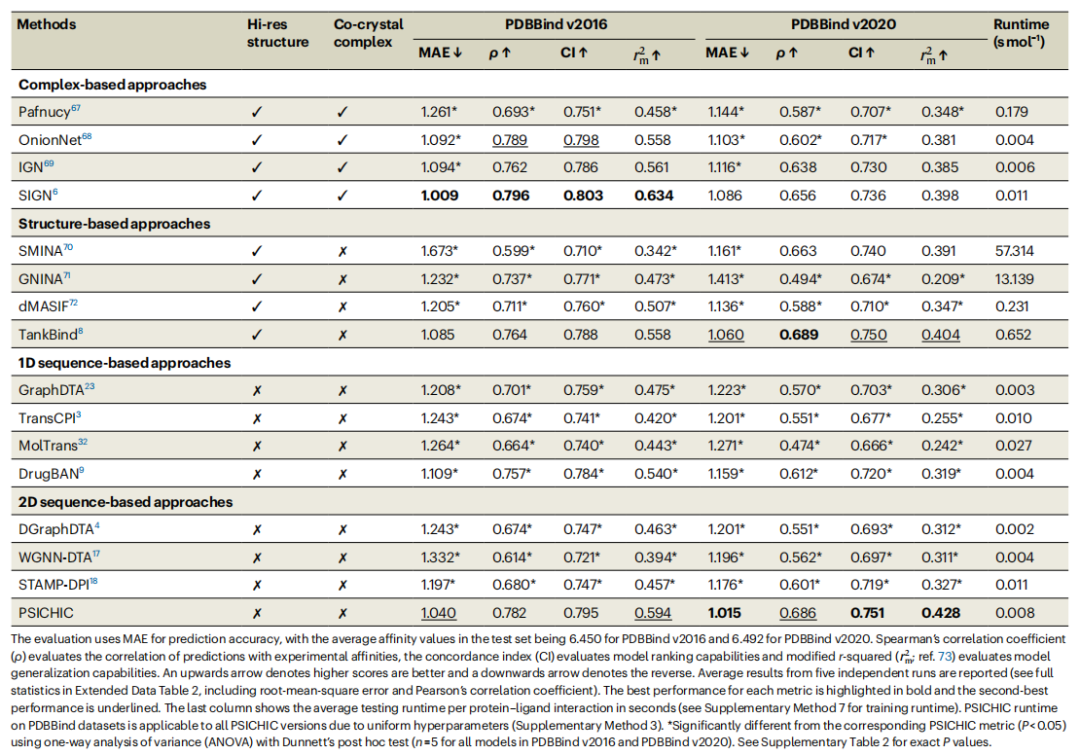
Außerdem:
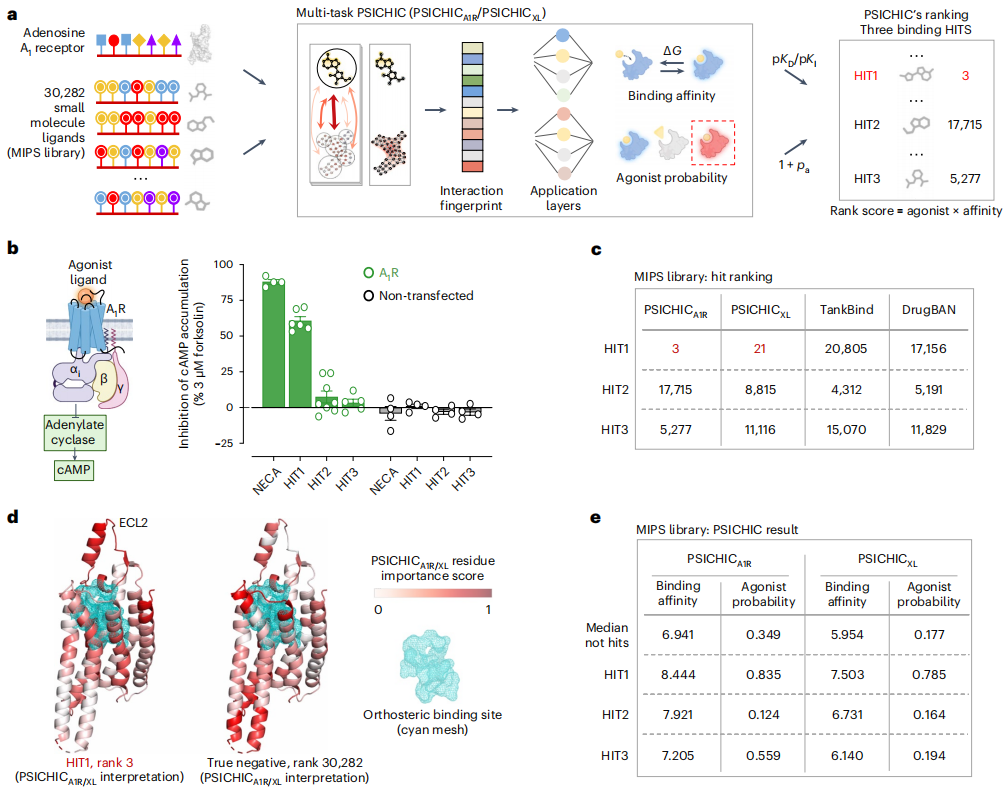
Interessanterweise zeigen die interpretierbaren Fingerabdrücke von PSICHIC, dass es die Fähigkeit erhält, den zugrunde liegenden Mechanismus von Protein-Ligand-Wechselwirkungen allein aus Sequenzdaten zu entschlüsseln, indem es Proteinreste an Bindungsstellen und die atomaren Fähigkeiten der beteiligten Liganden identifiziert, selbst wenn es nur darauf trainiert wird Sequenzdaten mit Bindungsaffinitätsmarkierungen und ohne Interaktionsinformationen.
Zukunftsausblick
Das obige ist der detaillierte Inhalt vonMit einer Genauigkeitsrate von 96 % prognostiziert KI Protein-Ligand-Wechselwirkungen anhand von Sequenzen. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!




