Technologie-Peripheriegeräte
Technologie-Peripheriegeräte
 KI
KI
 Das Molekül ist zu 100 % wirksam, die Liganden wurden von Grund auf neu entwickelt und die Hunan-Universität schlägt ein fragmentbasiertes molekulares Charakterisierungsgerüst vor
Das Molekül ist zu 100 % wirksam, die Liganden wurden von Grund auf neu entwickelt und die Hunan-Universität schlägt ein fragmentbasiertes molekulares Charakterisierungsgerüst vor
Das Molekül ist zu 100 % wirksam, die Liganden wurden von Grund auf neu entwickelt und die Hunan-Universität schlägt ein fragmentbasiertes molekulares Charakterisierungsgerüst vor
Anwendungen und Herausforderungen molekularer Deskriptoren
Molekulare Deskriptoren werden häufig in der molekularen Modellierung verwendet. Allerdings mangelt es im Bereich der KI-gestützten molekularen Entdeckung an natürlich anwendbaren, vollständigen und originellen molekularen Darstellungen, was sich auf die Modellleistung und Interpretierbarkeit auswirkt.
Vorschlag des t-SMILES-Frameworks
Das fragmentbasierte, mehrskalige molekulare Charakterisierungsframework t-SMILES löst das Problem der molekularen Charakterisierung. Das Framework verwendet Zeichenfolgen vom Typ SMILES zur Beschreibung von Molekülen und unterstützt Sequenzmodelle als generative Modelle.
t-SMILES‘ Code-Algorithmen
t-SMILES verfügt über drei Code-Algorithmen: TSSA, TSDY und TSID.
Experimentelle Ergebnisse
Experimente zeigen, dass die vom t-SMILES-Modell erzeugten Moleküle 100 % theoretische Gültigkeit und hohe Neuheit aufweisen, was besser ist als das auf SOTA SMILES basierende Modell.
Darüber hinaus vermeidet das t-SMILES-Modell eine Überanpassung und behält die Ähnlichkeit bei gekennzeichneten Datensätzen mit geringen Ressourcen bei, während gleichzeitig eine höhere Neuheit erreicht wird.
Veröffentlichte Informationen
Die Studie mit dem Titel „t-SMILES: ein fragmentbasiertes molekulares Darstellungsgerüst für das De-novo-Ligandendesign“ wurde am 11. Juni in „Nature Communications“ veröffentlicht.

Forschung zu molekularen Darstellungsmethoden basierend auf SMILES
Eine effektive Charakterisierung von Molekülen ist ein Schlüsselfaktor für die Leistung künstlicher Intelligenzmodelle.
Graph Neural Networks (GNN) sind bekannt für ihre Fähigkeit, 100 % effiziente Moleküle zu erzeugen, ihre Ausdrucksmöglichkeiten sind jedoch begrenzt.
Simplified Molecular Linear Input Specification (SMILES) als lineare Darstellung neigt dazu, chemisch ungültige Zeichenfolgen zu erzeugen. DeepSMILES und SELFIES sind Verbesserungen als Alternativen, weisen aber immer noch Probleme auf.
Darüber hinaus zeigen Untersuchungen, dass Sprachmodelle (LM) die meisten GNNs beim Lernen großer, komplexer Moleküle übertreffen können. Kürzlich haben auf Transformers basierende LMs ihre Fähigkeit unter Beweis gestellt, Texte zu erzeugen, die der menschlichen Schrift stark ähneln.
Inspiriert von diesen Ideen wählten die Forscher SMILES als Ausgangspunkt für die Fragmentbeschreibung und kombinierten es mit fortschrittlicher Technologie zur Verarbeitung natürlicher Sprache, um fragmentbasierte molekulare Modellierungsaufgaben zu bewältigen, die das Diagrammmodell zusammenführen können, um der molekularen Topologie mehr Aufmerksamkeit zu schenken LM Der Vorteil einer starken Lernfähigkeit.
Generieren Sie 100 % wirksame neue Moleküle, besser als SOTA
Daher schlug das Team der Hunan-Universität ein neues molekulares Beschreibungsgerüst vor, das auf fragmentierten Molekülen basiert, t-SMILES (baumbasiertes SMILES). Das Framework enthält drei t-SMILES-Kodierungsalgorithmen: TSSA (t-SMILES mit gemeinsamen Atomen), TSDY (t-SMILES mit virtuellen Atomen, aber ohne IDs) und TSID (t-SMILES mit IDs und virtuellen Atomen).

Das neu vorgeschlagene t-SMILES-Framework
- generiert azyklische Molekülbäume (AMT), die fragmentierte Moleküle darstellen.
- Konvertieren Sie AMT in einen vollständigen Binärbaum (FBT).
- Führen Sie eine Breitendurchquerung auf FBT durch, um die t-SMILES-Zeichenfolge zu erhalten.
Im Vergleich zu SMILES
t-SMILES führt nur zwei neue Symbole „&“ und „^“ ein, um eine mehrskalige und hierarchische molekulare Topologie zu kodieren. Der
t-SMILES-Algorithmus
bietet ein skalierbares und anpassbares Framework, das theoretisch eine breite Palette von Unterstrukturschemata unterstützen kann.
Das auf t-SMILES basierende Modell
ist in der Lage, topologische Strukturinformationen auf hoher Ebene zu erlernen und gleichzeitig detaillierte Unterstrukturinformationen zu verarbeiten.
Multicode-System
Der t-SMILES-Algorithmus kann ein Multicode-System zur molekularen Beschreibung erstellen, wobei:
- Klassisches SMILES als Sonderfall von t-SMILES (TS_Vanilla) integriert werden kann.
- Mehrere Beschreibungen können zusammenarbeiten, um die Gesamtleistung zu verbessern.
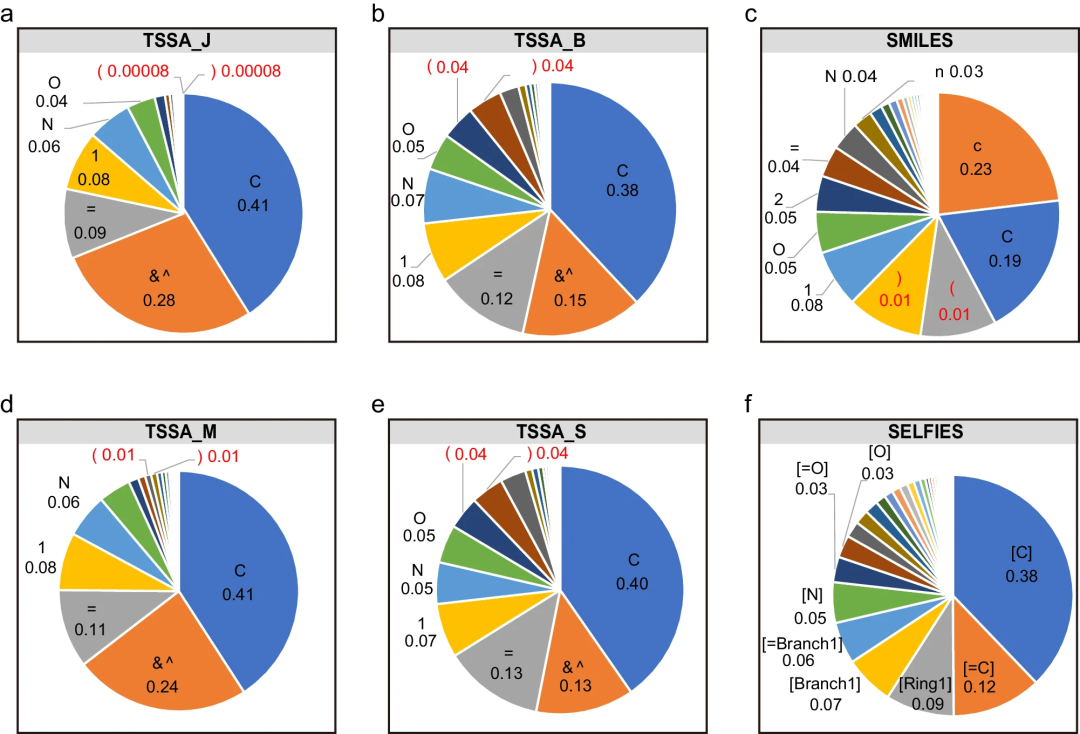
Illustration: Verteilung von Tokens für TSSA-Code, SMILES und SELFIES. (Quelle: Papier)
Zunächst bewerteten die Forscher t-SMILES systematisch, indem sie sich mit seinen einzigartigen Eigenschaften befassten. Anschließend wurden Experimente mit TSSA und TSDY an zwei markierten Datensätzen mit geringen Ressourcen durchgeführt, JNK332 und AID170633.
Die Forschung konzentriert sich auf die Einschränkungen von t-SMILES und seinen Alternativen, die durch die Nutzung von Standard-, Datenerweiterungs- und vorab trainierten, fein abgestimmten Modellen erreicht werden. Zwanzig zielgerichtete Aufgaben zu ChEMBL wurden parallel mithilfe von TSDY, TSSA und TSID bewertet. Es wurden auch gründliche Experimente mit ChEMBL, Zinc und QM9 durchgeführt, um t-SMILES und seine Alternativen unter Verwendung ähnlicher Aufbauten zu vergleichen. Darüber hinaus werden verschiedene fragmentbasierte Basismodelle und SOTA-GNN-Modelle verglichen.
Abschließend wird eine Ablationsstudie durchgeführt, um die Wirksamkeit des generativen Modells basierend auf SMILES mit Rekonstruktion zu bestätigen. Um die Anpassungsfähigkeit und Flexibilität des t-SMILES-Algorithmus zu bewerten, wurden vier zuvor veröffentlichte Fragmentierungsalgorithmen zur Zerlegung von Molekülen verwendet, darunter JTVAE, BRICS, MMPA und Scaffold. In verschiedenen Experimenten wurden drei Metriken verwendet: ein Benchmark für verteiltes Lernen, ein zielgerichteter Benchmark und die Wasserstein-Distanzmetrik für physikalisch-chemische Eigenschaften.
Detaillierte Vergleichsexperimente zeigen, dass die durch das t-SMILES-Modell erzeugten neuen Moleküle zu 100 % theoretisch gültig und besser sind als das auf SOTA SMILES basierende Modell. Im Vergleich zu SMILES, DSMILES und SELFIES kann die Gesamtlösung von t-SMILES Überanpassungsprobleme vermeiden und die ausgewogene Leistung bei Datensätzen mit geringen Ressourcen erheblich verbessern, unabhängig davon, ob Datenerweiterung oder ein vorab trainiertes und dann fein abgestimmtes Modell verwendet wird.
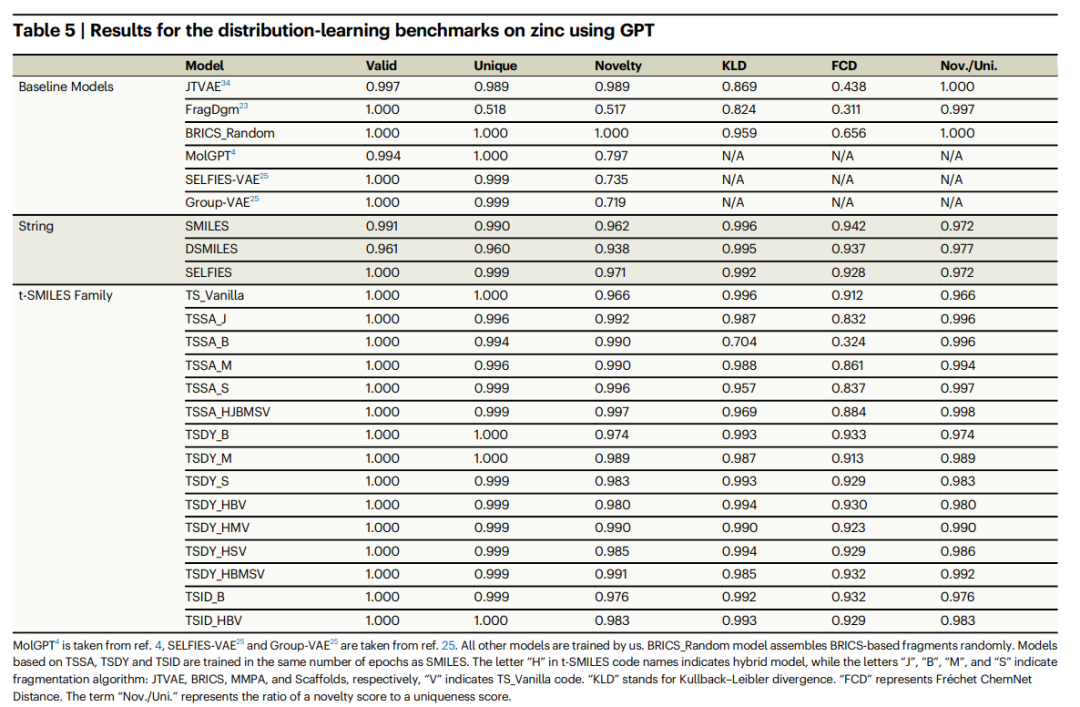
Darüber hinaus ist das t-SMILES-Modell in der Lage, die physikalisch-chemischen Eigenschaften von Molekülen geschickt zu erfassen und sicherzustellen, dass die erzeugten Moleküle Ähnlichkeit mit der Trainingsmolekülverteilung beibehalten. Dies verbessert die Leistung im Vergleich zu bestehenden fragmentbasierten und graphbasierten Basismodellen erheblich. Insbesondere das t-SMILES-Modell mit zielorientiertem Rekonstruktionsalgorithmus zeigt bei zielorientierten Aufgaben deutliche Vorteile gegenüber SMILES, DSMILES, SELFIES und SOTA CReM.
Einschränkungen und Verbesserungsmöglichkeiten
- LLM kann gut formatierte englische Grammatik verstehen. Ob die Baumstruktur von t-SMILES erlernt werden kann und wie LM über statistische Korrelationen von Oberflächen hinausgehen kann, um chemische Kenntnisse über Moleküle zu erlernen, muss daher noch eingehend untersucht werden.
- Diese Forschung konzentriert sich auf die Codierung fragmentierter Moleküle in Sequenzen, daher werden nur veröffentlichte Fragmentierungsalgorithmen als Beispiele für die Erstellung „chemischer Wörter“ verwendet. Zukünftige Forschungen können t-SMILES nutzen, um andere Fragmentierungsalgorithmen zu untersuchen, um chemische Sätze und Bedeutungen tiefer zu interpretieren, was tatsächlich anspruchsvoller ist als NLP.
- Obwohl t-SMILES darauf ausgelegt war, die Leistung molekularer Beschreibungen zu verbessern und die Einschränkungen von SMILES zu umgehen, wurde in der Studie nicht mit komplexeren Molekülen experimentiert. Dies wird Gegenstand zukünftiger Forschung sein.
- Schließlich ist dies ein vielversprechender Anfang für die Codierung fragmentierter Moleküle in Strings vom Typ SMILES. Weitere Forschung könnte fortschrittliche Algorithmen für die molekulare Rekonstruktion und Optimierung, verbesserte generative Modelle und evolutionäre Techniken untersuchen. Darüber hinaus kann sich die Forschung auf Aufgaben der Eigenschafts-, Retrosynthese- und Reaktionsvorhersage konzentrieren.
Hinweis: Das Cover stammt aus dem Internet
Das obige ist der detaillierte Inhalt vonDas Molekül ist zu 100 % wirksam, die Liganden wurden von Grund auf neu entwickelt und die Hunan-Universität schlägt ein fragmentbasiertes molekulares Charakterisierungsgerüst vor. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!

Heiße KI -Werkzeuge

Undresser.AI Undress
KI-gestützte App zum Erstellen realistischer Aktfotos

AI Clothes Remover
Online-KI-Tool zum Entfernen von Kleidung aus Fotos.

Undress AI Tool
Ausziehbilder kostenlos

Clothoff.io
KI-Kleiderentferner

Video Face Swap
Tauschen Sie Gesichter in jedem Video mühelos mit unserem völlig kostenlosen KI-Gesichtstausch-Tool aus!

Heißer Artikel

Heiße Werkzeuge

Notepad++7.3.1
Einfach zu bedienender und kostenloser Code-Editor

SublimeText3 chinesische Version
Chinesische Version, sehr einfach zu bedienen

Senden Sie Studio 13.0.1
Leistungsstarke integrierte PHP-Entwicklungsumgebung

Dreamweaver CS6
Visuelle Webentwicklungstools

SublimeText3 Mac-Version
Codebearbeitungssoftware auf Gottesniveau (SublimeText3)

Heiße Themen
 1667
1667
 14
14
 1426
1426
 52
52
 1328
1328
 25
25
 1273
1273
 29
29
 1255
1255
 24
24
 Der DeepMind-Roboter spielt Tischtennis und seine Vor- und Rückhand rutschen in die Luft, wodurch menschliche Anfänger völlig besiegt werden
Aug 09, 2024 pm 04:01 PM
Der DeepMind-Roboter spielt Tischtennis und seine Vor- und Rückhand rutschen in die Luft, wodurch menschliche Anfänger völlig besiegt werden
Aug 09, 2024 pm 04:01 PM
Aber vielleicht kann er den alten Mann im Park nicht besiegen? Die Olympischen Spiele in Paris sind in vollem Gange und Tischtennis hat viel Aufmerksamkeit erregt. Gleichzeitig haben Roboter auch beim Tischtennisspielen neue Durchbrüche erzielt. Gerade hat DeepMind den ersten lernenden Roboteragenten vorgeschlagen, der das Niveau menschlicher Amateurspieler im Tischtennis-Wettkampf erreichen kann. Papieradresse: https://arxiv.org/pdf/2408.03906 Wie gut ist der DeepMind-Roboter beim Tischtennisspielen? Vermutlich auf Augenhöhe mit menschlichen Amateurspielern: Sowohl Vorhand als auch Rückhand: Der Gegner nutzt unterschiedliche Spielstile, und auch der Roboter hält aus: Aufschlagannahme mit unterschiedlichem Spin: Allerdings scheint die Intensität des Spiels nicht so intensiv zu sein wie Der alte Mann im Park. Für Roboter, Tischtennis
 Die erste mechanische Klaue! Yuanluobao trat auf der Weltroboterkonferenz 2024 auf und stellte den ersten Schachroboter vor, der das Haus betreten kann
Aug 21, 2024 pm 07:33 PM
Die erste mechanische Klaue! Yuanluobao trat auf der Weltroboterkonferenz 2024 auf und stellte den ersten Schachroboter vor, der das Haus betreten kann
Aug 21, 2024 pm 07:33 PM
Am 21. August fand in Peking die Weltroboterkonferenz 2024 im großen Stil statt. Die Heimrobotermarke „Yuanluobot SenseRobot“ von SenseTime hat ihre gesamte Produktfamilie vorgestellt und kürzlich den Yuanluobot AI-Schachspielroboter – Chess Professional Edition (im Folgenden als „Yuanluobot SenseRobot“ bezeichnet) herausgebracht und ist damit der weltweit erste A-Schachroboter für heim. Als drittes schachspielendes Roboterprodukt von Yuanluobo hat der neue Guoxiang-Roboter eine Vielzahl spezieller technischer Verbesserungen und Innovationen in den Bereichen KI und Maschinenbau erfahren und erstmals die Fähigkeit erkannt, dreidimensionale Schachfiguren aufzunehmen B. durch mechanische Klauen an einem Heimroboter, und führen Sie Mensch-Maschine-Funktionen aus, z. B. Schach spielen, jeder spielt Schach, Überprüfung der Notation usw.
 Claude ist auch faul geworden! Netizen: Lernen Sie, sich einen Urlaub zu gönnen
Sep 02, 2024 pm 01:56 PM
Claude ist auch faul geworden! Netizen: Lernen Sie, sich einen Urlaub zu gönnen
Sep 02, 2024 pm 01:56 PM
Der Schulstart steht vor der Tür und nicht nur die Schüler, die bald ins neue Semester starten, sollten auf sich selbst aufpassen, sondern auch die großen KI-Modelle. Vor einiger Zeit war Reddit voller Internetnutzer, die sich darüber beschwerten, dass Claude faul werde. „Sein Niveau ist stark gesunken, es kommt oft zu Pausen und sogar die Ausgabe wird sehr kurz. In der ersten Woche der Veröffentlichung konnte es ein komplettes 4-seitiges Dokument auf einmal übersetzen, aber jetzt kann es nicht einmal eine halbe Seite ausgeben.“ !
 Auf der Weltroboterkonferenz wurde dieser Haushaltsroboter, der „die Hoffnung auf eine zukünftige Altenpflege' in sich trägt, umzingelt
Aug 22, 2024 pm 10:35 PM
Auf der Weltroboterkonferenz wurde dieser Haushaltsroboter, der „die Hoffnung auf eine zukünftige Altenpflege' in sich trägt, umzingelt
Aug 22, 2024 pm 10:35 PM
Auf der World Robot Conference in Peking ist die Präsentation humanoider Roboter zum absoluten Mittelpunkt der Szene geworden. Am Stand von Stardust Intelligent führte der KI-Roboterassistent S1 drei große Darbietungen mit Hackbrett, Kampfkunst und Kalligraphie auf Ein Ausstellungsbereich, der sowohl Literatur als auch Kampfkunst umfasst, zog eine große Anzahl von Fachpublikum und Medien an. Durch das elegante Spiel auf den elastischen Saiten demonstriert der S1 eine feine Bedienung und absolute Kontrolle mit Geschwindigkeit, Kraft und Präzision. CCTV News führte einen Sonderbericht über das Nachahmungslernen und die intelligente Steuerung hinter „Kalligraphie“ durch. Firmengründer Lai Jie erklärte, dass hinter den seidenweichen Bewegungen die Hardware-Seite die beste Kraftkontrolle und die menschenähnlichsten Körperindikatoren (Geschwindigkeit, Belastung) anstrebt. usw.), aber auf der KI-Seite werden die realen Bewegungsdaten von Menschen gesammelt, sodass der Roboter stärker werden kann, wenn er auf eine schwierige Situation stößt, und lernen kann, sich schnell weiterzuentwickeln. Und agil
 Bekanntgabe der ACL 2024 Awards: Eines der besten Papers zum Thema Oracle Deciphering von HuaTech, GloVe Time Test Award
Aug 15, 2024 pm 04:37 PM
Bekanntgabe der ACL 2024 Awards: Eines der besten Papers zum Thema Oracle Deciphering von HuaTech, GloVe Time Test Award
Aug 15, 2024 pm 04:37 PM
Bei dieser ACL-Konferenz haben die Teilnehmer viel gewonnen. Die sechstägige ACL2024 findet in Bangkok, Thailand, statt. ACL ist die führende internationale Konferenz im Bereich Computerlinguistik und Verarbeitung natürlicher Sprache. Sie wird von der International Association for Computational Linguistics organisiert und findet jährlich statt. ACL steht seit jeher an erster Stelle, wenn es um akademischen Einfluss im Bereich NLP geht, und ist außerdem eine von der CCF-A empfohlene Konferenz. Die diesjährige ACL-Konferenz ist die 62. und hat mehr als 400 innovative Arbeiten im Bereich NLP eingereicht. Gestern Nachmittag gab die Konferenz den besten Vortrag und weitere Auszeichnungen bekannt. Diesmal gibt es 7 Best Paper Awards (zwei davon unveröffentlicht), 1 Best Theme Paper Award und 35 Outstanding Paper Awards. Die Konferenz verlieh außerdem drei Resource Paper Awards (ResourceAward) und einen Social Impact Award (
 Das Team von Li Feifei schlug ReKep vor, um Robotern räumliche Intelligenz zu verleihen und GPT-4o zu integrieren
Sep 03, 2024 pm 05:18 PM
Das Team von Li Feifei schlug ReKep vor, um Robotern räumliche Intelligenz zu verleihen und GPT-4o zu integrieren
Sep 03, 2024 pm 05:18 PM
Tiefe Integration von Vision und Roboterlernen. Wenn zwei Roboterhände reibungslos zusammenarbeiten, um Kleidung zu falten, Tee einzuschenken und Schuhe zu packen, gepaart mit dem humanoiden 1X-Roboter NEO, der in letzter Zeit für Schlagzeilen gesorgt hat, haben Sie vielleicht das Gefühl: Wir scheinen in das Zeitalter der Roboter einzutreten. Tatsächlich sind diese seidigen Bewegungen das Produkt fortschrittlicher Robotertechnologie + exquisitem Rahmendesign + multimodaler großer Modelle. Wir wissen, dass nützliche Roboter oft komplexe und exquisite Interaktionen mit der Umgebung erfordern und die Umgebung als Einschränkungen im räumlichen und zeitlichen Bereich dargestellt werden kann. Wenn Sie beispielsweise möchten, dass ein Roboter Tee einschenkt, muss der Roboter zunächst den Griff der Teekanne ergreifen und sie aufrecht halten, ohne den Tee zu verschütten, und ihn dann sanft bewegen, bis die Öffnung der Kanne mit der Öffnung der Tasse übereinstimmt , und neigen Sie dann die Teekanne in einem bestimmten Winkel. Das
 Distributed Artificial Intelligence Conference DAI 2024 Call for Papers: Agent Day, Richard Sutton, der Vater des Reinforcement Learning, wird teilnehmen! Yan Shuicheng, Sergey Levine und DeepMind-Wissenschaftler werden Grundsatzreden halten
Aug 22, 2024 pm 08:02 PM
Distributed Artificial Intelligence Conference DAI 2024 Call for Papers: Agent Day, Richard Sutton, der Vater des Reinforcement Learning, wird teilnehmen! Yan Shuicheng, Sergey Levine und DeepMind-Wissenschaftler werden Grundsatzreden halten
Aug 22, 2024 pm 08:02 PM
Einleitung zur Konferenz Mit der rasanten Entwicklung von Wissenschaft und Technologie ist künstliche Intelligenz zu einer wichtigen Kraft bei der Förderung des sozialen Fortschritts geworden. In dieser Zeit haben wir das Glück, die Innovation und Anwendung der verteilten künstlichen Intelligenz (DAI) mitzuerleben und daran teilzuhaben. Verteilte Künstliche Intelligenz ist ein wichtiger Zweig des Gebiets der Künstlichen Intelligenz, der in den letzten Jahren immer mehr Aufmerksamkeit erregt hat. Durch die Kombination des leistungsstarken Sprachverständnisses und der Generierungsfähigkeiten großer Modelle sind plötzlich Agenten aufgetaucht, die auf natürlichen Sprachinteraktionen, Wissensbegründung, Aufgabenplanung usw. basieren. AIAgent übernimmt das große Sprachmodell und ist zu einem heißen Thema im aktuellen KI-Kreis geworden. Au
 Hongmeng Smart Travel S9 und die umfassende Einführungskonferenz für neue Produkte wurden gemeinsam mit einer Reihe neuer Blockbuster-Produkte veröffentlicht
Aug 08, 2024 am 07:02 AM
Hongmeng Smart Travel S9 und die umfassende Einführungskonferenz für neue Produkte wurden gemeinsam mit einer Reihe neuer Blockbuster-Produkte veröffentlicht
Aug 08, 2024 am 07:02 AM
Heute Nachmittag begrüßte Hongmeng Zhixing offiziell neue Marken und neue Autos. Am 6. August veranstaltete Huawei die Hongmeng Smart Xingxing S9 und die Huawei-Konferenz zur Einführung neuer Produkte mit umfassendem Szenario und brachte die Panorama-Smart-Flaggschiff-Limousine Xiangjie S9, das neue M7Pro und Huawei novaFlip, MatePad Pro 12,2 Zoll, das neue MatePad Air und Huawei Bisheng mit Mit vielen neuen Smart-Produkten für alle Szenarien, darunter die Laserdrucker der X1-Serie, FreeBuds6i, WATCHFIT3 und der Smart Screen S5Pro, von Smart Travel über Smart Office bis hin zu Smart Wear baut Huawei weiterhin ein Smart-Ökosystem für alle Szenarien auf, um Verbrauchern ein Smart-Erlebnis zu bieten Internet von allem. Hongmeng Zhixing: Huawei arbeitet mit chinesischen Partnern aus der Automobilindustrie zusammen, um die Modernisierung der Smart-Car-Industrie voranzutreiben