

Proteine sind an vielen biologischen Funktionen wie der Zellzusammensetzung, der Muskelkontraktion, der Verdauung von Nahrungsmitteln und der Identifizierung von Viren beteiligt.
Um bessere Proteine (einschließlich Antikörper) zu entwickeln, mutieren Wissenschaftler häufig wiederholt Aminosäuren (indem sie die Einheiten, aus denen Proteine bestehen, in einer bestimmten Reihenfolge anordnen) an verschiedenen Positionen, bis das Protein die erforderliche Funktion erhält.
Aber es gibt mehr Aminosäuresequenzen als Sandkörner auf der Welt, daher ist es oft schwierig, das beste Protein und damit das beste potenzielle Medikament zu finden. Angesichts dieser Herausforderung geben Wissenschaftler oft Millionen von Dollar aus und testen miniaturisierte, vereinfachte Versionen biologischer Systeme.
„Dies erfordert viel Rätselraten und Verifizierung.“ Brian L. Hie, Assistenzprofessor für Chemieingenieurwesen an der Stanford University und Innovationsstipendiat am Arc Institute, sagte: „Das Ziel vieler intelligenter Algorithmen besteht darin, das Rätselraten zu beseitigen.“
Wissenschaftler der Stanford University haben eine neue Methode entwickelt, die auf maschinellem Lernen basiert und molekulare Veränderungen, die zu besseren Antikörpermedikamenten führen, schneller und genauer vorhersagen kann. Durch die Kombination der 3D-Struktur des Proteinrückgrats mit einem großen Sprachmodell basierend auf der Aminosäuresequenz konnten die Forscher innerhalb von Minuten seltene und wünschenswerte Mutationen finden.
Die Studie trug den Titel „Unüberwachte Evolution von Protein- und Antikörperkomplexen mit einem strukturinformierten Sprachmodell“ und wurde am 4. Juli 2024 in „Science“ veröffentlicht.

Große Sprachmodelle, die ausschließlich auf Sequenzinformationen trainiert werden, können hochrangige Prinzipien des Proteindesigns erlernen. Neben der Sequenz bestimmt jedoch auch die dreidimensionale Struktur von Proteinen ihre spezifische Funktion, Aktivität und Evolvierbarkeit.
Für Probleme bei der Antikörpertechnik verwendeten Forscher der Stanford University strukturell informierte Proteinsprachmodelle, um Sequenzen mit hoher Fitness vorherzusagen, die durch bekannte Antikörper- oder Antikörper-Antigen-Komplexstrukturen eingeschränkt sind.
Untersuchungen zeigen, dass ein universelles Protein-Sprachmodell, ergänzt um Protein-Strukturkoordinaten, die Entwicklung verschiedener Proteine steuern kann, ohne dass einzelne Funktionsaufgaben modelliert werden müssen.
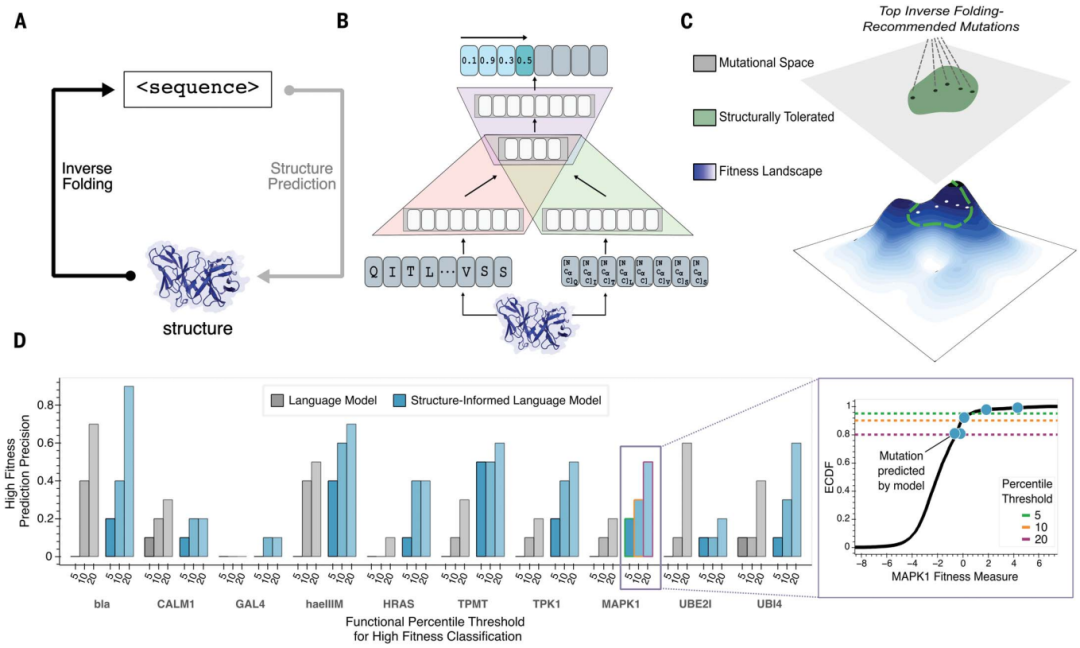
Strukturgesteuertes Paradigma:
Breite Anwendung:
Proteinkomplexdesign:
Entwicklung menschlicher Antikörper:
Große Datenmengen ersetzen:
Gerichtete Evolution:

Mit dieser Methode untersuchte das Team etwa 30 Kandidaten auf zwei therapeutische klinische Antikörper zur Behandlung schwerer Varianten des akuten respiratorischen Syndroms Coronavirus 2 (SARS-CoV-2). Gleichzeitig erreichten die Forscher eine 25-fache Steigerung der Neutralisierung und eine 37-fache Steigerung der Affinität gegen die Antikörper-Escape-Virusvarianten BQ.1.1 und XBB.1.5.
Zusammenfassend lässt sich sagen: Dieses Tool hilft dabei, schnell auf neue oder sich entwickelnde Krankheiten zu reagieren. Es senkt auch die Hürden für die Herstellung wirksamerer Medikamente. Stärkere Medikamente bedeuten, dass geringere Dosen erforderlich sind, sodass mehr Patienten von einer bestimmten Dosis profitieren können.
Link zum Papier: https://www.science.org/doi/10.1126/science.adk8946
Verwandte Berichte: https://phys.org/news/2024-07-ai-approach-optimizes- Antikörper-Medikamente.html
Das obige ist der detaillierte Inhalt vonMelden Sie sich bei Science an, die Arzneimittelaffinität ist um das 37-fache gestiegen, die KI führt eine unbeaufsichtigte Optimierung von Protein- und Antikörperkomplexen durch. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
 Anwendung künstlicher Intelligenz im Leben
Anwendung künstlicher Intelligenz im Leben
 Was ist das Grundkonzept der künstlichen Intelligenz?
Was ist das Grundkonzept der künstlichen Intelligenz?
 Was sind Python-Bezeichner?
Was sind Python-Bezeichner?
 Software zur Verschlüsselung von Mobiltelefonen
Software zur Verschlüsselung von Mobiltelefonen
 So verwenden Sie die Durchschnittsfunktion
So verwenden Sie die Durchschnittsfunktion
 So können Sie den Gasverbrauch nach der Zahlung wieder aufnehmen
So können Sie den Gasverbrauch nach der Zahlung wieder aufnehmen
 Was ist eine NFC-Zugangskontrollkarte?
Was ist eine NFC-Zugangskontrollkarte?
 Samsung s5830-Upgrade
Samsung s5830-Upgrade




