 Technologie-Peripheriegeräte
Technologie-Peripheriegeräte
 KI
KI
 Die Teams von Tencent und der Peking-Universität entwickelten Antikörper von Grund auf, trainierten vorab große Sprachmodelle und veröffentlichten sie im Nature-Unterjournal
Die Teams von Tencent und der Peking-Universität entwickelten Antikörper von Grund auf, trainierten vorab große Sprachmodelle und veröffentlichten sie im Nature-Unterjournal
Die Teams von Tencent und der Peking-Universität entwickelten Antikörper von Grund auf, trainierten vorab große Sprachmodelle und veröffentlichten sie im Nature-Unterjournal
 Herausgeber |. KX
Herausgeber |. KX
Die KI-Technologie hat große Fortschritte bei der Unterstützung des Antikörperdesigns gemacht. Allerdings ist das Antikörperdesign immer noch stark auf die Isolierung antigenspezifischer Antikörper aus Serum angewiesen, was ein ressourcenintensiver und zeitaufwändiger Prozess ist.
Um dieses Problem zu lösen, schlug das Forschungsteam des Tencent AI Lab, der Peking University Shenzhen Graduate School und des Xijing Digestive Disease Hospital ein vorab trainiertes großes Sprachmodell zur Antikörpererzeugung (PALM-H3) für de vor Novo Generation Künstliche Antikörper CDRH3 mit der erforderlichen Antigenbindungsspezifität, wodurch die Abhängigkeit von natürlichen Antikörpern verringert wird.
Darüber hinaus wurde ein hochpräzises Antigen-Antikörper-Bindungsvorhersagemodell A2binder entwickelt, um die Antigen-Epitopsequenz mit der Antikörpersequenz abzugleichen und so die Bindungsspezifität und -affinität vorherzusagen.
Zusammenfassend lässt sich sagen, dass diese Studie ein künstliches Intelligenz-Framework für die Antikörpergenerierung und -bewertung etabliert, das das Potenzial hat, die Entwicklung von Antikörpermedikamenten erheblich zu beschleunigen.
Verwandte Forschung mit dem Titel „De-novo-Generierung des SARS-CoV-2-Antikörpers CDRH3 mit einem vorab trainierten generativen großen Sprachmodell“ wurde am 10. August veröffentlicht 🎜#Nature Communications".

Das Framework von PALM-H3 und A2Binder
Der Workflow und das Modell-Framework von PALM-H3 und A2Binder sind in der folgenden Abbildung dargestellt.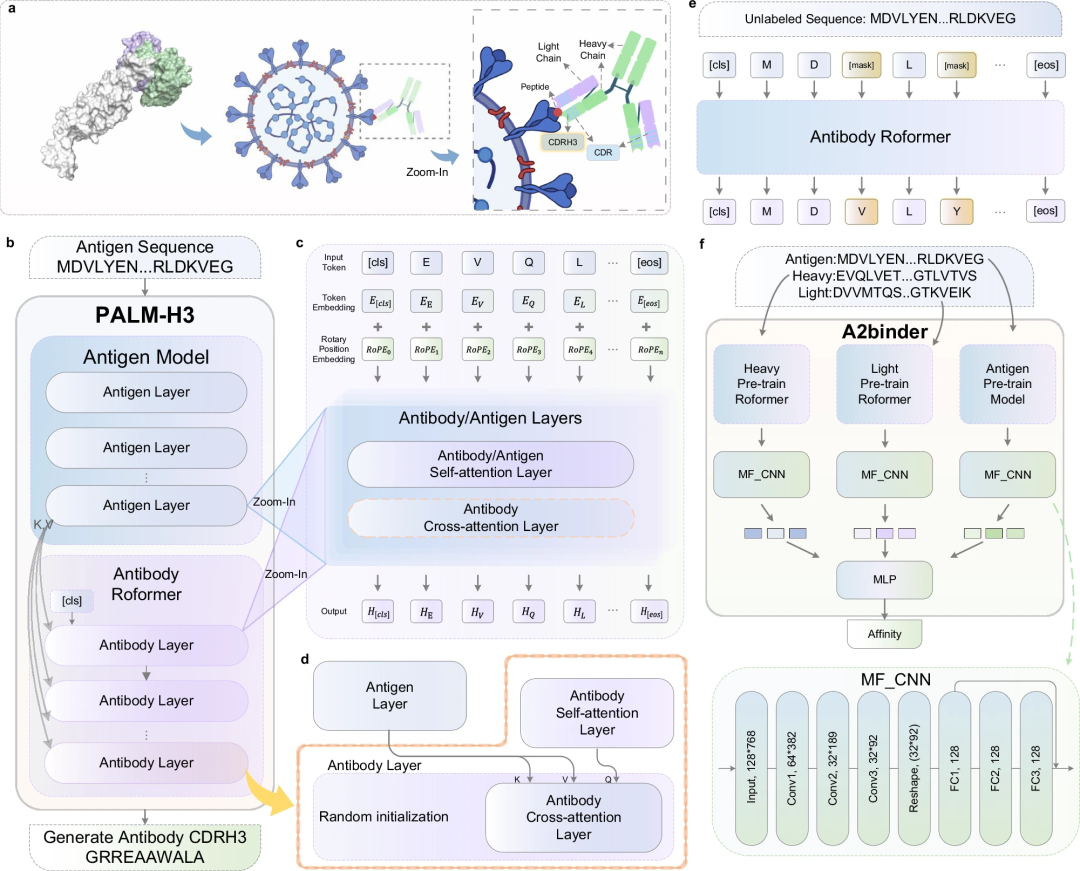
Die Konstruktion von PALM-H3 und A2binder umfasst drei Schritte: Zunächst trainierten die Forscher zwei Roformer auf ungepaarten Antikörper-Schwerketten- bzw. Leichtkettensequenzen im Modell. Anschließend wurde A2binder auf der Grundlage von vorab trainiertem ESM2, Antikörper-Schwerkette-Roformer und Antikörper-Leichtkette-Roformer erstellt und mithilfe gepaarter Affinitätsdaten trainiert. Schließlich wurde PALM-H3 unter Verwendung von vortrainiertem ESM2 und Antikörper-Schwerkette-Roformer erstellt und auf gepaarten Antigen-CDRH3-Daten trainiert, um CDRH3 de novo zu erzeugen. A2binder kann die Wahrscheinlichkeit der Antigen-Antikörper-Bindung und die Affinität seiner Leistung genau vorhersagen.
A2binder schneidet beim Affinitätsdatensatz gut ab, teilweise aufgrund des Vortrainings auf Antikörpersequenzen, das es A2binder ermöglicht, die in diesen Sequenzen vorhandenen einzigartigen Muster zu lernen.結果表明,在所有抗原抗體親和力預測資料集上,A2binder 的表現均優於基線模型ESM-F(後者俱有相同的框架,但預訓練模型被ESM2 取代),這表明使用抗體序列進行預訓練可能對相關的下游任務有益。
為了評估模型在預測親和力值方面的表現,研究人員也利用了兩個包含親和力值標籤的資料集 14H 和 14L。
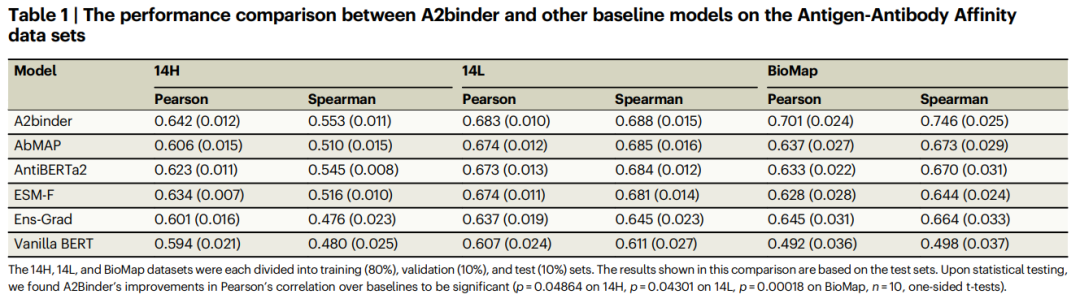
A2binder 在 Pearson 相關性和 Spearman 相關性指標上均優於所有基線模型。 A2binder 在 14H 資料集上實現了 0.642 的 Pearson 相關性(提高了 3%),在 14L 資料集上實現了 0.683(提高了 1%)。
然而,與其他資料集相比,A2binder 和其他基準模型在 14H 和 14L 資料集上的表現略有下降。這項觀察結果與先前的研究一致。
PALM-H3 在生成高結合機率抗體方面表現優異
研究人員探討了 PALM-H3 產生的抗體與天然抗體之間的差異。發現它們的序列有顯著差異,但產生的抗體的結合機率並沒有受到這些差異的顯著影響。同時,它們的結構差異確實導致結合親和力的下降。這些結果與先前關於抗體庫網絡分析和功能性蛋白質序列生成的研究一致。
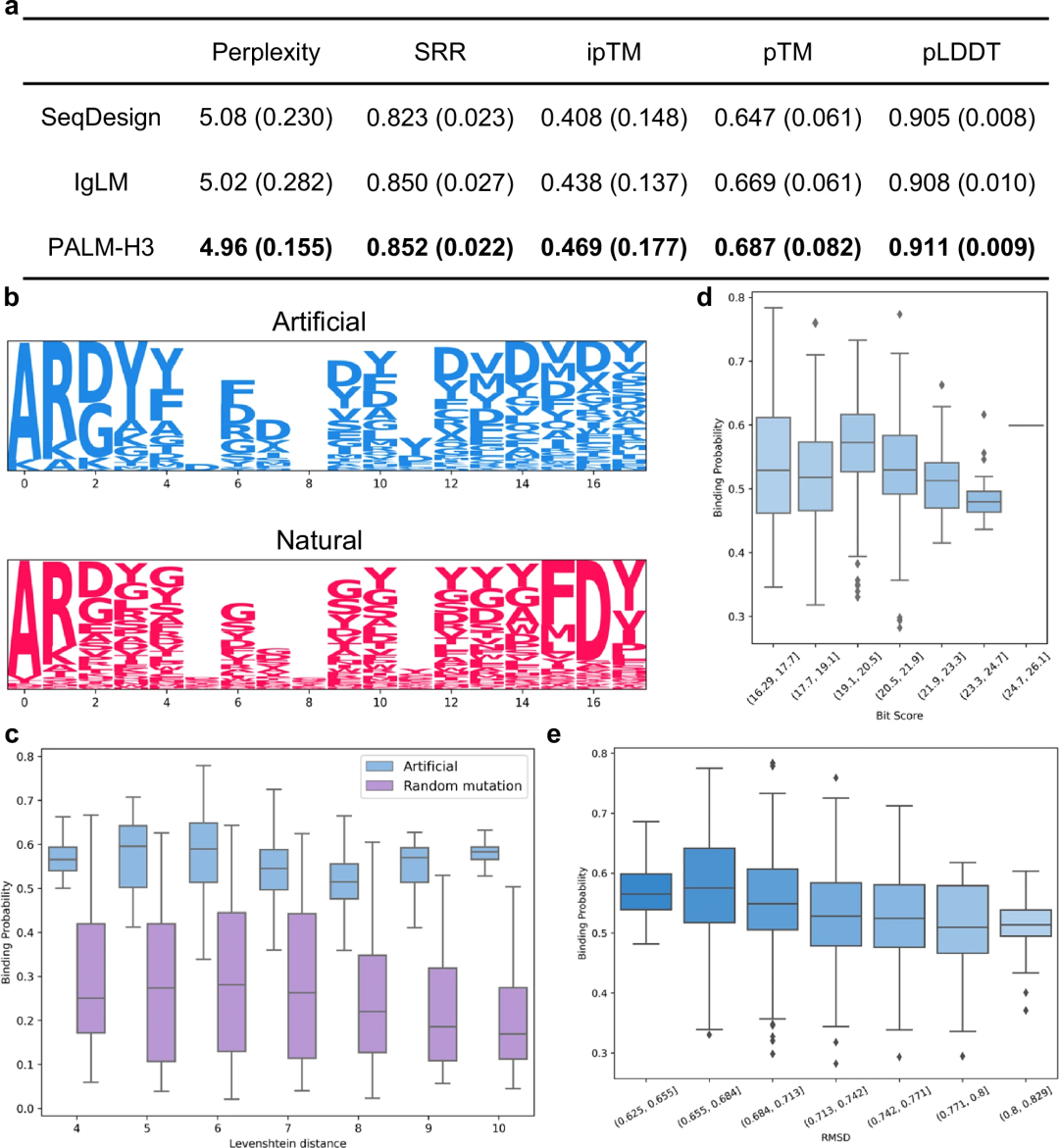
總體而言,結果表明,儘管與天然抗體不同,但 PALM-H3 能夠產生具有高結合親和力的多種抗體序列。
此外,研究人員透過 ClusPro 和 SnugDock 驗證了 PALM-H3 的性能。 PALM-H3 能夠產生針對 SARS-CoV-2 HR2 區穩定勝肽的抗體 CDRH3 序列。它產生了新的 CDRH3 序列,並且驗證了產生的序列 GRREAAWALA 與天然 CDHR3 序列 GKAAGTFDS 相比,對抗原穩定勝肽的標靶性有所改善。
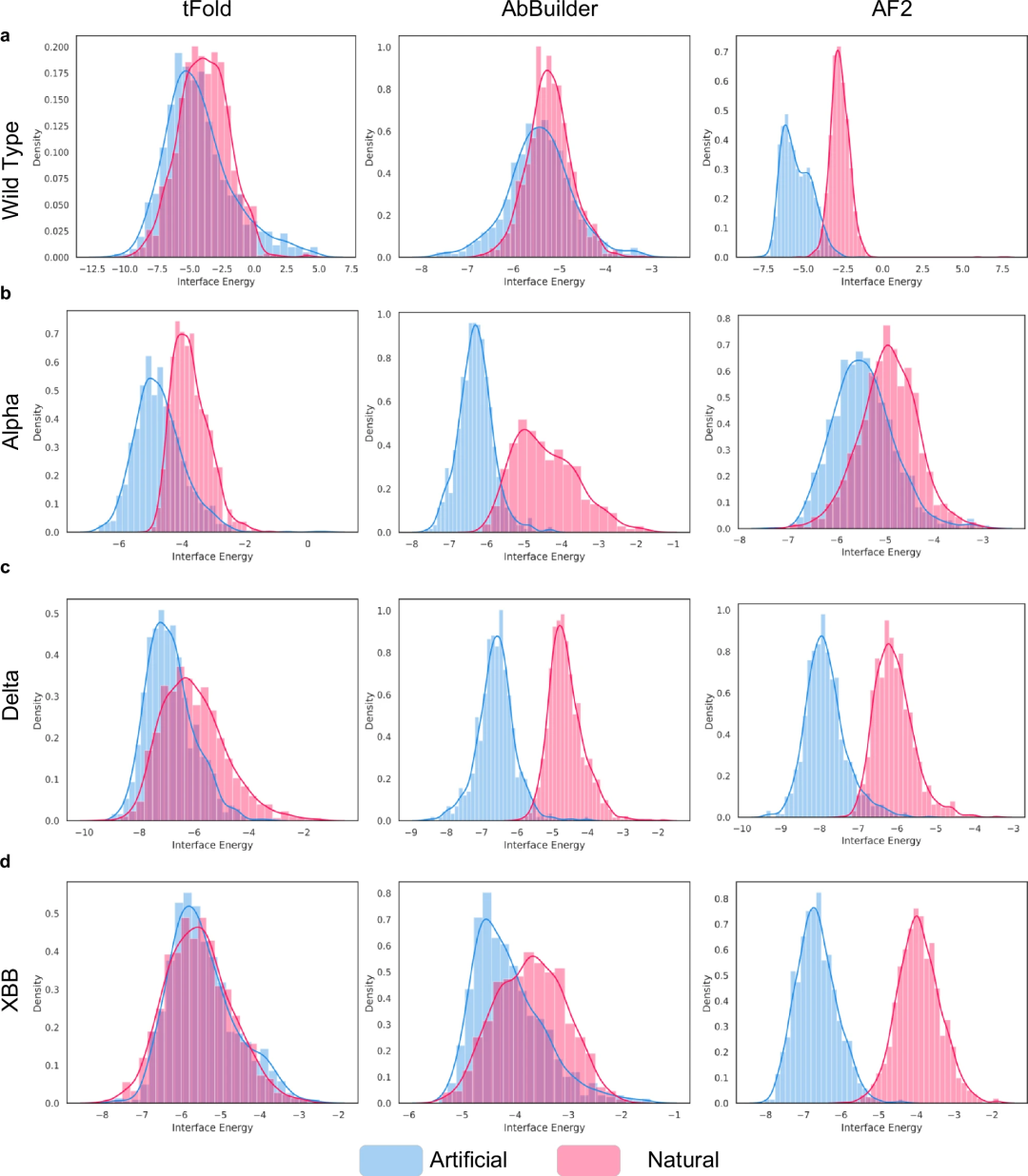
此外,PALM-H3 能夠產生對新出現的 SARS-CoV-2 變體 XBB 具有更高親和力的抗體 CDRH3 序列。產生的序列 AKDSRTSPLRLDYS 對 XBB 的親和力比其來源 ASEVLDNLRDGYNF 更強。
此外,PALM-H3 不僅克服了傳統順序突變策略面臨的局部最優陷阱,而且與 E-EVO 方法相比,它還能產生具有更高抗原結合親和力的抗體。這凸顯了 PALM-H3 在抗體設計方面的優勢,能夠更有效地探索序列空間並產生針對特定表位的高親和力結合物。
體外實驗
此外,研究人員還進行了體外試驗,包括蛋白質印跡、表面等離子體共振分析和假病毒中和試驗,為 PALM-H3 設計抗體的有效性提供了關鍵驗證。
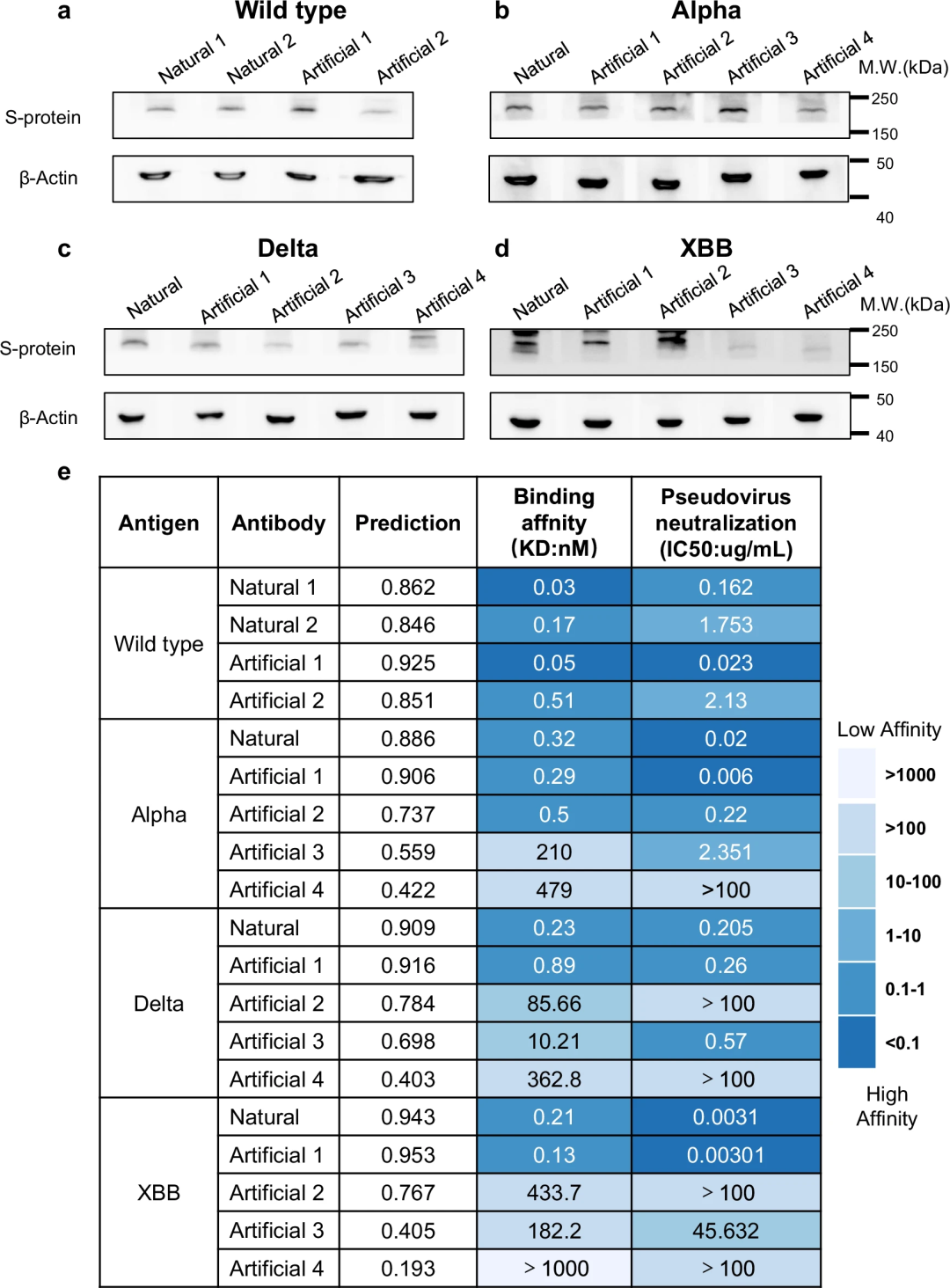
PALM-H3 產生的針對SARS-CoV-2 野生型、Alpha、Delta 和XBB 變體刺突蛋白的兩種抗體在這些試驗中都實現了比天然抗體更高的結合親和力和中和效力。這些濕實驗室實驗的有力經驗結果補充了計算預測和分析,驗證了 PALM-H3 和 A2binder 在生成和選擇對已知和新抗原具有高特異性和親和力的強效抗體方面的能力。
總之,提出的 PALM-H3 整合了大規模抗體預訓練的能力和全局特徵融合的有效性,從而具有卓越的親和力預測性能和設計高親和力抗體的能力。此外,直接序列產生和可解釋的權重視覺化使其成為設計高親和力抗體的有效且可解釋的工具。
Das obige ist der detaillierte Inhalt vonDie Teams von Tencent und der Peking-Universität entwickelten Antikörper von Grund auf, trainierten vorab große Sprachmodelle und veröffentlichten sie im Nature-Unterjournal. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!

Heiße KI -Werkzeuge

Undresser.AI Undress
KI-gestützte App zum Erstellen realistischer Aktfotos

AI Clothes Remover
Online-KI-Tool zum Entfernen von Kleidung aus Fotos.

Undress AI Tool
Ausziehbilder kostenlos

Clothoff.io
KI-Kleiderentferner

Video Face Swap
Tauschen Sie Gesichter in jedem Video mühelos mit unserem völlig kostenlosen KI-Gesichtstausch-Tool aus!

Heißer Artikel

Heiße Werkzeuge

Notepad++7.3.1
Einfach zu bedienender und kostenloser Code-Editor

SublimeText3 chinesische Version
Chinesische Version, sehr einfach zu bedienen

Senden Sie Studio 13.0.1
Leistungsstarke integrierte PHP-Entwicklungsumgebung

Dreamweaver CS6
Visuelle Webentwicklungstools

SublimeText3 Mac-Version
Codebearbeitungssoftware auf Gottesniveau (SublimeText3)

Heiße Themen
 1674
1674
 14
14
 1429
1429
 52
52
 1333
1333
 25
25
 1278
1278
 29
29
 1257
1257
 24
24
 „Defect Spectrum' durchbricht die Grenzen der herkömmlichen Fehlererkennung und erreicht erstmals eine hochpräzise und umfassende semantische Fehlererkennung in der Industrie.
Jul 26, 2024 pm 05:38 PM
„Defect Spectrum' durchbricht die Grenzen der herkömmlichen Fehlererkennung und erreicht erstmals eine hochpräzise und umfassende semantische Fehlererkennung in der Industrie.
Jul 26, 2024 pm 05:38 PM
In der modernen Fertigung ist die genaue Fehlererkennung nicht nur der Schlüssel zur Sicherstellung der Produktqualität, sondern auch der Kern für die Verbesserung der Produktionseffizienz. Allerdings mangelt es vorhandenen Datensätzen zur Fehlererkennung häufig an der Genauigkeit und dem semantischen Reichtum, die für praktische Anwendungen erforderlich sind, was dazu führt, dass Modelle bestimmte Fehlerkategorien oder -orte nicht identifizieren können. Um dieses Problem zu lösen, hat ein Spitzenforschungsteam bestehend aus der Hong Kong University of Science and Technology Guangzhou und Simou Technology innovativ den „DefectSpectrum“-Datensatz entwickelt, der eine detaillierte und semantisch reichhaltige groß angelegte Annotation von Industriedefekten ermöglicht. Wie in Tabelle 1 gezeigt, bietet der Datensatz „DefectSpectrum“ im Vergleich zu anderen Industriedatensätzen die meisten Fehleranmerkungen (5438 Fehlerproben) und die detaillierteste Fehlerklassifizierung (125 Fehlerkategorien).
 Training mit Millionen von Kristalldaten zur Lösung kristallographischer Phasenprobleme, die Deep-Learning-Methode PhAI wird in Science veröffentlicht
Aug 08, 2024 pm 09:22 PM
Training mit Millionen von Kristalldaten zur Lösung kristallographischer Phasenprobleme, die Deep-Learning-Methode PhAI wird in Science veröffentlicht
Aug 08, 2024 pm 09:22 PM
Herausgeber |KX Bis heute sind die durch die Kristallographie ermittelten Strukturdetails und Präzision, von einfachen Metallen bis hin zu großen Membranproteinen, mit keiner anderen Methode zu erreichen. Die größte Herausforderung, das sogenannte Phasenproblem, bleibt jedoch die Gewinnung von Phaseninformationen aus experimentell bestimmten Amplituden. Forscher der Universität Kopenhagen in Dänemark haben eine Deep-Learning-Methode namens PhAI entwickelt, um Kristallphasenprobleme zu lösen. Ein Deep-Learning-Neuronales Netzwerk, das mithilfe von Millionen künstlicher Kristallstrukturen und den entsprechenden synthetischen Beugungsdaten trainiert wird, kann genaue Elektronendichtekarten erstellen. Die Studie zeigt, dass diese Deep-Learning-basierte Ab-initio-Strukturlösungsmethode das Phasenproblem mit einer Auflösung von nur 2 Angström lösen kann, was nur 10 bis 20 % der bei atomarer Auflösung verfügbaren Daten im Vergleich zur herkömmlichen Ab-initio-Berechnung entspricht
 Das NVIDIA-Dialogmodell ChatQA wurde auf Version 2.0 weiterentwickelt, wobei die angegebene Kontextlänge 128 KB beträgt
Jul 26, 2024 am 08:40 AM
Das NVIDIA-Dialogmodell ChatQA wurde auf Version 2.0 weiterentwickelt, wobei die angegebene Kontextlänge 128 KB beträgt
Jul 26, 2024 am 08:40 AM
Die offene LLM-Community ist eine Ära, in der hundert Blumen blühen und konkurrieren. Sie können Llama-3-70B-Instruct, QWen2-72B-Instruct, Nemotron-4-340B-Instruct, Mixtral-8x22BInstruct-v0.1 und viele andere sehen hervorragende Darsteller. Allerdings weisen offene Modelle im Vergleich zu den proprietären Großmodellen GPT-4-Turbo in vielen Bereichen noch erhebliche Lücken auf. Zusätzlich zu allgemeinen Modellen wurden einige offene Modelle entwickelt, die sich auf Schlüsselbereiche spezialisieren, wie etwa DeepSeek-Coder-V2 für Programmierung und Mathematik und InternVL für visuelle Sprachaufgaben.
 Google AI gewann die Silbermedaille der IMO Mathematical Olympiad, das mathematische Argumentationsmodell AlphaProof wurde eingeführt und Reinforcement Learning ist zurück
Jul 26, 2024 pm 02:40 PM
Google AI gewann die Silbermedaille der IMO Mathematical Olympiad, das mathematische Argumentationsmodell AlphaProof wurde eingeführt und Reinforcement Learning ist zurück
Jul 26, 2024 pm 02:40 PM
Für KI ist die Mathematikolympiade kein Problem mehr. Am Donnerstag hat die künstliche Intelligenz von Google DeepMind eine Meisterleistung vollbracht: Sie nutzte KI, um meiner Meinung nach die eigentliche Frage der diesjährigen Internationalen Mathematikolympiade zu lösen, und war nur einen Schritt davon entfernt, die Goldmedaille zu gewinnen. Der IMO-Wettbewerb, der gerade letzte Woche zu Ende ging, hatte sechs Fragen zu Algebra, Kombinatorik, Geometrie und Zahlentheorie. Das von Google vorgeschlagene hybride KI-System beantwortete vier Fragen richtig und erzielte 28 Punkte und erreichte damit die Silbermedaillenstufe. Anfang dieses Monats hatte der UCLA-Professor Terence Tao gerade die KI-Mathematische Olympiade (AIMO Progress Award) mit einem Millionenpreis gefördert. Unerwarteterweise hatte sich das Niveau der KI-Problemlösung vor Juli auf dieses Niveau verbessert. Beantworten Sie die Fragen meiner Meinung nach gleichzeitig. Am schwierigsten ist es meiner Meinung nach, da sie die längste Geschichte, den größten Umfang und die negativsten Fragen haben
 PRO |. Warum verdienen große Modelle, die auf MoE basieren, mehr Aufmerksamkeit?
Aug 07, 2024 pm 07:08 PM
PRO |. Warum verdienen große Modelle, die auf MoE basieren, mehr Aufmerksamkeit?
Aug 07, 2024 pm 07:08 PM
Im Jahr 2023 entwickeln sich fast alle Bereiche der KI in beispielloser Geschwindigkeit weiter. Gleichzeitig verschiebt die KI ständig die technologischen Grenzen wichtiger Bereiche wie der verkörperten Intelligenz und des autonomen Fahrens. Wird der Status von Transformer als Mainstream-Architektur großer KI-Modelle durch den multimodalen Trend erschüttert? Warum ist die Erforschung großer Modelle auf Basis der MoE-Architektur (Mixture of Experts) zu einem neuen Trend in der Branche geworden? Können Large Vision Models (LVM) ein neuer Durchbruch im allgemeinen Sehvermögen sein? ...Aus dem PRO-Mitglieder-Newsletter 2023 dieser Website, der in den letzten sechs Monaten veröffentlicht wurde, haben wir 10 spezielle Interpretationen ausgewählt, die eine detaillierte Analyse der technologischen Trends und industriellen Veränderungen in den oben genannten Bereichen bieten, um Ihnen dabei zu helfen, Ihre Ziele in der Zukunft zu erreichen Jahr vorbereitet sein. Diese Interpretation stammt aus Week50 2023
 Um ein neues wissenschaftliches und komplexes Frage-Antwort-Benchmark- und Bewertungssystem für große Modelle bereitzustellen, haben UNSW, Argonne, die University of Chicago und andere Institutionen gemeinsam das SciQAG-Framework eingeführt
Jul 25, 2024 am 06:42 AM
Um ein neues wissenschaftliches und komplexes Frage-Antwort-Benchmark- und Bewertungssystem für große Modelle bereitzustellen, haben UNSW, Argonne, die University of Chicago und andere Institutionen gemeinsam das SciQAG-Framework eingeführt
Jul 25, 2024 am 06:42 AM
Herausgeber | Der Frage-Antwort-Datensatz (QA) von ScienceAI spielt eine entscheidende Rolle bei der Förderung der Forschung zur Verarbeitung natürlicher Sprache (NLP). Hochwertige QS-Datensätze können nicht nur zur Feinabstimmung von Modellen verwendet werden, sondern auch effektiv die Fähigkeiten großer Sprachmodelle (LLMs) bewerten, insbesondere die Fähigkeit, wissenschaftliche Erkenntnisse zu verstehen und zu begründen. Obwohl es derzeit viele wissenschaftliche QS-Datensätze aus den Bereichen Medizin, Chemie, Biologie und anderen Bereichen gibt, weisen diese Datensätze immer noch einige Mängel auf. Erstens ist das Datenformular relativ einfach, die meisten davon sind Multiple-Choice-Fragen. Sie sind leicht auszuwerten, schränken jedoch den Antwortauswahlbereich des Modells ein und können die Fähigkeit des Modells zur Beantwortung wissenschaftlicher Fragen nicht vollständig testen. Im Gegensatz dazu offene Fragen und Antworten
 Die Genauigkeitsrate erreicht 60,8 %. Das auf Transformer basierende Modell zur Vorhersage der chemischen Retrosynthese wurde in der Unterzeitschrift „Nature' veröffentlicht
Aug 06, 2024 pm 07:34 PM
Die Genauigkeitsrate erreicht 60,8 %. Das auf Transformer basierende Modell zur Vorhersage der chemischen Retrosynthese wurde in der Unterzeitschrift „Nature' veröffentlicht
Aug 06, 2024 pm 07:34 PM
Herausgeber | KX-Retrosynthese ist eine entscheidende Aufgabe in der Arzneimittelforschung und organischen Synthese, und KI wird zunehmend eingesetzt, um den Prozess zu beschleunigen. Bestehende KI-Methoden weisen eine unbefriedigende Leistung und eine begrenzte Vielfalt auf. In der Praxis verursachen chemische Reaktionen häufig lokale molekulare Veränderungen mit erheblichen Überschneidungen zwischen Reaktanten und Produkten. Davon inspiriert schlug das Team von Hou Tingjun an der Zhejiang-Universität vor, die einstufige retrosynthetische Vorhersage als eine Aufgabe zur Bearbeitung molekularer Ketten neu zu definieren und dabei die Zielmolekülkette iterativ zu verfeinern, um Vorläuferverbindungen zu erzeugen. Außerdem wird ein bearbeitungsbasiertes retrosynthetisches Modell EditRetro vorgeschlagen, mit dem qualitativ hochwertige und vielfältige Vorhersagen erzielt werden können. Umfangreiche Experimente zeigen, dass das Modell beim Standard-Benchmark-Datensatz USPTO-50 K eine hervorragende Leistung mit einer Top-1-Genauigkeit von 60,8 % erzielt.
 Der Standpunkt der Natur: Die Erprobung künstlicher Intelligenz in der Medizin ist im Chaos. Was ist zu tun?
Aug 22, 2024 pm 04:37 PM
Der Standpunkt der Natur: Die Erprobung künstlicher Intelligenz in der Medizin ist im Chaos. Was ist zu tun?
Aug 22, 2024 pm 04:37 PM
Herausgeber | ScienceAI Basierend auf begrenzten klinischen Daten wurden Hunderte medizinischer Algorithmen genehmigt. Wissenschaftler diskutieren darüber, wer die Werkzeuge testen soll und wie dies am besten geschieht. Devin Singh wurde Zeuge, wie ein pädiatrischer Patient in der Notaufnahme einen Herzstillstand erlitt, während er lange auf eine Behandlung wartete, was ihn dazu veranlasste, den Einsatz von KI zu erforschen, um Wartezeiten zu verkürzen. Mithilfe von Triage-Daten aus den Notaufnahmen von SickKids erstellten Singh und Kollegen eine Reihe von KI-Modellen, um mögliche Diagnosen zu stellen und Tests zu empfehlen. Eine Studie zeigte, dass diese Modelle die Zahl der Arztbesuche um 22,3 % verkürzen können und die Verarbeitung der Ergebnisse pro Patient, der einen medizinischen Test benötigt, um fast drei Stunden beschleunigt. Der Erfolg von Algorithmen der künstlichen Intelligenz in der Forschung bestätigt dies jedoch nur



