
Bildsegmentierung, eines der grundlegendsten Verfahren in der Bildverarbeitung, ermöglicht es einem System, verschiedene Bereiche innerhalb eines Bildes zu zerlegen und zu analysieren. Ob es um Objekterkennung, medizinische Bildgebung oder autonomes Fahren geht, die Segmentierung ist es, die Bilder in sinnvolle Teile zerlegt.
Obwohl Deep-Learning-Modelle bei dieser Aufgabe immer beliebter werden, sind traditionelle Techniken in der digitalen Bildverarbeitung immer noch leistungsstark und praktisch. Zu den in diesem Beitrag besprochenen Ansätzen gehören Schwellenwertbildung, Kantenerkennung, bereichsbasiertes Clustering und die Implementierung eines bekannten Datensatzes für die Analyse von Zellbildern, des MIVIA HEp-2-Bilddatensatzes.
Der MIVIA HEp-2-Bilddatensatz besteht aus einer Reihe von Bildern der Zellen, die zur Analyse des Musters antinukleärer Antikörper (ANA) durch HEp-2-Zellen verwendet werden. Es besteht aus 2D-Bildern, die mittels Fluoreszenzmikroskopie aufgenommen wurden. Dadurch eignet es sich sehr gut für Segmentierungsaufgaben, vor allem für die medizinische Bildanalyse, bei der die Erkennung zellulärer Regionen am wichtigsten ist.
Jetzt kommen wir zu den Segmentierungstechniken, die zur Verarbeitung dieser Bilder verwendet werden, und vergleichen ihre Leistung basierend auf den F1-Ergebnissen.
Schwellenwert ist der Prozess, bei dem Graustufenbilder basierend auf Pixelintensitäten in Binärbilder umgewandelt werden. Im MIVIA HEp-2-Datensatz ist dieser Prozess bei der Zellextraktion aus dem Hintergrund nützlich. Es ist relativ einfach und effektiv, insbesondere mit der Otsu-Methode, da sie den optimalen Schwellenwert selbst berechnet.
Otsus Methode ist eine automatische Schwellenwertmethode, bei der versucht wird, den besten Schwellenwert zu finden, um die minimale Varianz innerhalb der Klasse zu erzielen und dadurch die beiden Klassen zu trennen: Vordergrund (Zellen) und Hintergrund. Die Methode untersucht das Bildhistogramm und berechnet den perfekten Schwellenwert, bei dem die Summe der Pixelintensitätsvarianzen in jeder Klasse minimiert wird.
# Thresholding Segmentation
def thresholding(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Otsu's thresholding
_, thresh = cv.threshold(gray, 0, 255, cv.THRESH_BINARY + cv.THRESH_OTSU)
return thresh
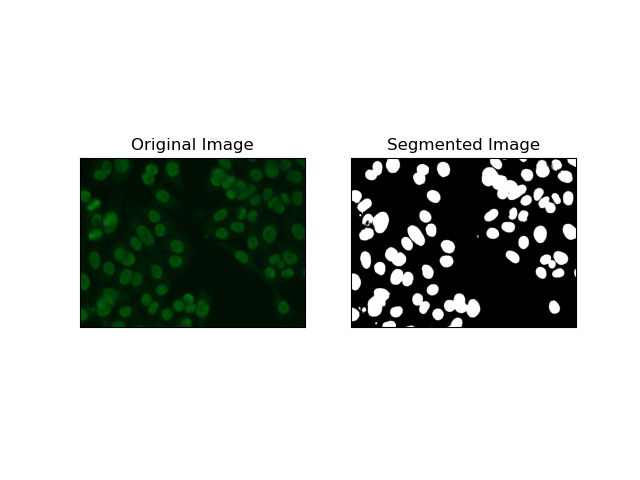
Kantenerkennung bezieht sich auf die Identifizierung von Grenzen von Objekten oder Regionen, wie z. B. Zellkanten im MIVIA HEp-2-Datensatz. Von den vielen verfügbaren Methoden zur Erkennung abrupter Intensitätsänderungen ist der Canny Edge Detector die beste und daher am besten geeignete Methode zur Erkennung von Zellgrenzen.
Canny Edge Detector ist ein mehrstufiger Algorithmus, der die Kanten erkennen kann, indem er Bereiche mit starken Intensitätsgradienten erkennt. Der Prozess umfasst die Glättung mit einem Gaußschen Filter, die Berechnung von Intensitätsgradienten, die Anwendung einer nicht-maximalen Unterdrückung zur Eliminierung von Störreaktionen und eine abschließende doppelte Schwellenwertoperation zur Beibehaltung nur hervorstechender Kanten.
# Edge Detection Segmentation
def edge_detection(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Gaussian blur
gray = cv.GaussianBlur(gray, (3, 3), 0)
# Calculate lower and upper thresholds for Canny edge detection
sigma = 0.33
v = np.median(gray)
lower = int(max(0, (1.0 - sigma) * v))
upper = int(min(255, (1.0 + sigma) * v))
# Apply Canny edge detection
edges = cv.Canny(gray, lower, upper)
# Dilate the edges to fill gaps
kernel = np.ones((5, 5), np.uint8)
dilated_edges = cv.dilate(edges, kernel, iterations=2)
# Clean the edges using morphological opening
cleaned_edges = cv.morphologyEx(dilated_edges, cv.MORPH_OPEN, kernel, iterations=1)
# Find connected components and filter out small components
num_labels, labels, stats, _ = cv.connectedComponentsWithStats(
cleaned_edges, connectivity=8
)
min_size = 500
filtered_mask = np.zeros_like(cleaned_edges)
for i in range(1, num_labels):
if stats[i, cv.CC_STAT_AREA] >= min_size:
filtered_mask[labels == i] = 255
# Find contours of the filtered mask
contours, _ = cv.findContours(
filtered_mask, cv.RETR_EXTERNAL, cv.CHAIN_APPROX_SIMPLE
)
# Create a filled mask using the contours
filled_mask = np.zeros_like(gray)
cv.drawContours(filled_mask, contours, -1, (255), thickness=cv.FILLED)
# Perform morphological closing to fill holes
final_filled_image = cv.morphologyEx(
filled_mask, cv.MORPH_CLOSE, kernel, iterations=2
)
# Dilate the final filled image to smooth the edges
final_filled_image = cv.dilate(final_filled_image, kernel, iterations=1)
return final_filled_image
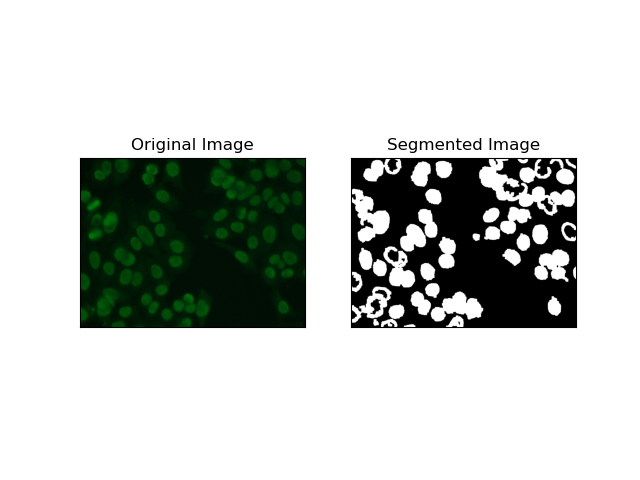
Regionale Segmentierung gruppiert ähnliche Pixel in Regionen, abhängig von bestimmten Kriterien wie Intensität oder Farbe. Die Watershed-Segmentierung-Technik kann zur Segmentierung von HEp-2-Zellbildern verwendet werden, um die Regionen erkennen zu können, die Zellen darstellen; Es betrachtet Pixelintensitäten als topografische Oberfläche und umreißt unterscheidende Regionen.
Wassereinzugsgebietssegmentierung behandelt die Intensitäten von Pixeln als topografische Oberfläche. Der Algorithmus identifiziert „Becken“, in denen er lokale Minima identifiziert, und überflutet diese Becken dann schrittweise, um bestimmte Regionen zu vergrößern. Diese Technik ist sehr nützlich, wenn man sich berührende Objekte trennen möchte, etwa im Fall von Zellen in mikroskopischen Bildern, sie kann jedoch rauschempfindlich sein. Der Prozess kann durch Marker gesteuert werden und eine Übersegmentierung kann oft reduziert werden.
# Region-Based Segmentation
def region_based(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Otsu's thresholding
_, thresh = cv.threshold(gray, 0, 255, cv.THRESH_BINARY_INV + cv.THRESH_OTSU)
# Apply morphological opening to remove noise
kernel = np.ones((3, 3), np.uint8)
opening = cv.morphologyEx(thresh, cv.MORPH_OPEN, kernel, iterations=2)
# Dilate the opening to get the background
sure_bg = cv.dilate(opening, kernel, iterations=3)
# Calculate the distance transform
dist_transform = cv.distanceTransform(opening, cv.DIST_L2, 5)
# Threshold the distance transform to get the foreground
_, sure_fg = cv.threshold(dist_transform, 0.2 * dist_transform.max(), 255, 0)
sure_fg = np.uint8(sure_fg)
# Find the unknown region
unknown = cv.subtract(sure_bg, sure_fg)
# Label the markers for watershed algorithm
_, markers = cv.connectedComponents(sure_fg)
markers = markers + 1
markers[unknown == 255] = 0
# Apply watershed algorithm
markers = cv.watershed(img, markers)
# Create a mask for the segmented region
mask = np.zeros_like(gray, dtype=np.uint8)
mask[markers == 1] = 255
return mask
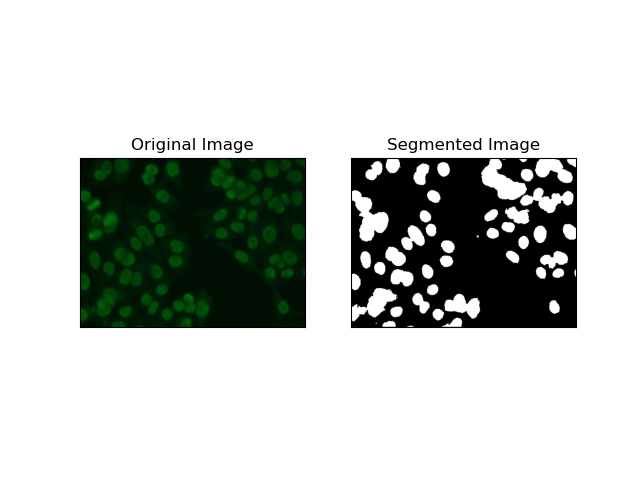
Clustering-Techniken wie K-Means neigen dazu, die Pixel in ähnlichen Clustern zu gruppieren, was gut funktioniert, wenn Zellen in mehrfarbigen oder komplexen Umgebungen segmentiert werden sollen, wie in HEp-2-Zellbildern zu sehen ist. Grundsätzlich könnte dies verschiedene Klassen darstellen, beispielsweise eine Zellregion gegenüber einem Hintergrund.
K-means is an unsupervised learning algorithm for clustering images based on the pixel similarity of color or intensity. The algorithm randomly selects K centroids, assigns each pixel to the nearest centroid, and updates the centroid iteratively until it converges. It is particularly effective in segmenting an image that has multiple regions of interest that are very different from one another.
# Clustering Segmentation
def clustering(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Reshape the image
Z = gray.reshape((-1, 3))
Z = np.float32(Z)
# Define the criteria for k-means clustering
criteria = (cv.TERM_CRITERIA_EPS + cv.TERM_CRITERIA_MAX_ITER, 10, 1.0)
# Set the number of clusters
K = 2
# Perform k-means clustering
_, label, center = cv.kmeans(Z, K, None, criteria, 10, cv.KMEANS_RANDOM_CENTERS)
# Convert the center values to uint8
center = np.uint8(center)
# Reshape the result
res = center[label.flatten()]
res = res.reshape((gray.shape))
# Apply thresholding to the result
_, res = cv.threshold(res, 0, 255, cv.THRESH_BINARY + cv.THRESH_OTSU)
return res

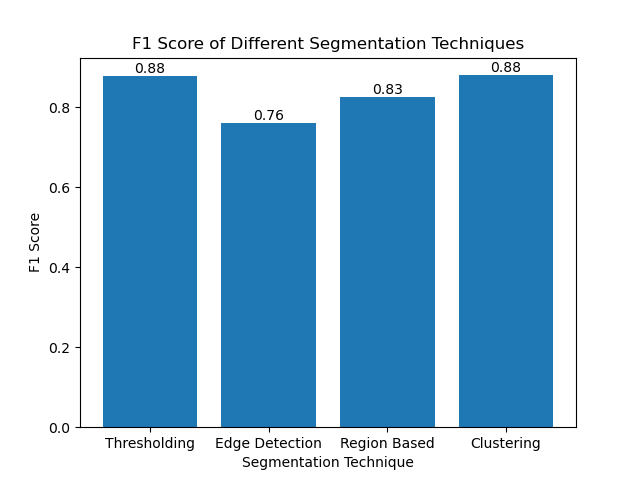
The F1 score is a measure that combines precision and recall together to compare the predicted segmentation image with the ground truth image. It is the harmonic mean of precision and recall, which is useful in cases of high data imbalance, such as in medical imaging datasets.
We calculated the F1 score for each segmentation method by flattening both the ground truth and the segmented image and calculating the weighted F1 score.
def calculate_f1_score(ground_image, segmented_image):
ground_image = ground_image.flatten()
segmented_image = segmented_image.flatten()
return f1_score(ground_image, segmented_image, average="weighted")
We then visualized the F1 scores of different methods using a simple bar chart:
Although many recent approaches for image segmentation are emerging, traditional segmentation techniques such as thresholding, edge detection, region-based methods, and clustering can be very useful when applied to datasets such as the MIVIA HEp-2 image dataset.
Each method has its strength:
By evaluating these methods using F1 scores, we understand the trade-offs each of these models has. These methods may not be as sophisticated as what is developed in the newest models of deep learning, but they are still fast, interpretable, and serviceable in a broad range of applications.
Thanks for reading! I hope this exploration of traditional image segmentation techniques inspires your next project. Feel free to share your thoughts and experiences in the comments below!
Das obige ist der detaillierte Inhalt vonBildsegmentierung beherrschen: Wie traditionelle Techniken auch im digitalen Zeitalter glänzen. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
 Unterbewertete Münzen, die man im Jahr 2024 horten sollte
Unterbewertete Münzen, die man im Jahr 2024 horten sollte
 Der heutige Marktpreis von Ethereum
Der heutige Marktpreis von Ethereum
 js-Methode zum Löschen des Knotens
js-Methode zum Löschen des Knotens
 Netzwerkrahmen
Netzwerkrahmen
 Was bedeutet Chrom?
Was bedeutet Chrom?
 So konfigurieren Sie die Datenbankverbindung in mybatis
So konfigurieren Sie die Datenbankverbindung in mybatis
 Was ist die Tastenkombination für die Pinselgröße?
Was ist die Tastenkombination für die Pinselgröße?
 So verwenden Sie Redis als Cache-Server
So verwenden Sie Redis als Cache-Server
 C-Sprachkonstantenverwendung
C-Sprachkonstantenverwendung




