 Technologie-Peripheriegeräte
Technologie-Peripheriegeräte
 KI
KI
 Open Source! Hong Kong Chinese, MIT und Fudan schlagen das erste RNA-Grundsteinmodell vor
Open Source! Hong Kong Chinese, MIT und Fudan schlagen das erste RNA-Grundsteinmodell vor
Open Source! Hong Kong Chinese, MIT und Fudan schlagen das erste RNA-Grundsteinmodell vor
Im Gegensatz zum Proteinbereich mangelt es der Forschung im RNA-Bereich häufig an ausreichenden Annotationsdaten. Beispielsweise umfassen 3D-Daten nur mehr als 1.000 RNAs. Dies schränkt die Entwicklung maschineller Lernmethoden für Aufgaben zur Vorhersage von RNA-Struktur-Funktionen erheblich ein.
Um den Mangel an annotierten Daten auszugleichen, zeigt dieser Artikel ein Eckpfeilermodell, das umfassendes strukturelles und funktionelles Wissen für verschiedene Arten der RNA-Forschung liefern kann – das RNA-Grundlagenmodell (RNA-FM). Als weltweit erstes RNA-Ecksteinmodell, das unbeaufsichtigt auf der Grundlage von 23 Millionen unbeschrifteten RNA-Sequenzen trainiert wurde, untersucht RNA-FM die in RNA-Sequenzen enthaltenen evolutionären und strukturellen Muster.
Es ist erwähnenswert, dass RNA-FM nur einem einfachen Downstream-Modell entsprechen oder nur Einbettung ermöglichen muss und bei vielen Downstream-Aufgaben eine weitaus bessere Leistung als SOTA erzielen kann, beispielsweise bei der Verbesserung der Sekundärstrukturvorhersage um 20 % Die Kartenvorhersage kann um 30 % verbessert werden. Groß angelegte Experimente haben gezeigt, dass das Modell hochgradig generalisierbar ist und sogar für COVID-19 und regulatorische Fragmente der mRNA verwendet werden kann. ?? FM

Server: https://proj.cse.cuhk.edu.hk/rnafm
- EinführungIn den letzten Jahren haben auf Deep Learning basierende biologische Computermethoden Durchbrüche auf diesem Gebiet erzielt Der bekannteste Meilenstein ist das vom Google DeepMind-Team entwickelte End-to-End-Framework zur Vorhersage der 3D-Struktur von Proteinen AlphaFold2. Allerdings ist Protein nur eine Art von vielen biologischen Molekülen. Gene (DNA/RNA) enthalten als Quelle der Proteinproduktion mehr grundlegende Informationen als letztere und haben einen größeren Forschungswert.
- Im Allgemeinen sind Proteine die Produkte der Translation von RNA, die zur Kodierung verwendet wird, d. h. mRNA. Eine feste mRNA kann in eine feste Proteinsequenz übersetzt werden. Tatsächlich macht dieser Teil der kodierenden RNA nur 2 % aller RNA-Sequenzen aus, und die restlichen 98 % sind nicht-kodierende RNA (ncRNA). Obwohl ncRNAs nicht direkt in Proteine „übersetzt“ werden, falten sie sich zu Tertiärstrukturen mit spezifischen Funktionen und spielen eine regulatorische Rolle im Translationsprozess von mRNA oder anderen biologischen Funktionen. Daher ist die Analyse der Struktur und Funktion von ncRNA eine grundlegendere und komplexere Forschung als die Proteinanalyse. Verglichen mit dem Proteinbereich, wo rechnerische Methoden ausgereifter sind, befindet sich die RNA-basierte Struktur- und Funktionsvorhersage jedoch noch in einem frühen Stadium, und rechnerische Methoden, die ursprünglich auf den Proteinbereich anwendbar waren, lassen sich nur schwer direkt auf den RNA-Bereich übertragen . Die Haupteinschränkung dieser Berechnungsmethoden besteht darin, dass die Annotation von RNA-Daten normalerweise schwierig zu erhalten ist und dass die Annotation einer kleinen Datenmenge viele experimentelle Ressourcen und Zeit erfordert. Die meisten Berechnungsmethoden erfordern eine große Menge an annotierter Daten für die Betreuung, um Höchstleistungen zu erbringen. Obwohl es nicht viele annotierte Daten gibt, hat das RNA-Feld tatsächlich viele nicht annotierte Sequenzdaten angesammelt. Die Methode dieses Artikels besteht darin, diese unbeschrifteten Daten zu verwenden, um zusätzliche effektive Informationen für verschiedene nachgelagerte Aufgaben bereitzustellen.
- Basierend auf dieser Überlegung schlugen Hongkong Chinese, MIT, Fudan und Shanghai Artificial Intelligence Laboratory-Teams
ein Grundlagenmodell vor, ein RNA-Grundlagenmodell, das unbeaufsichtigt auf 23 Millionen unbeschrifteten reinen RNA-Sequenzen trainiert wurde (RNA-FM). . Obwohl die Daten während des Trainingsprozesses keine Annotationsinformationen liefern, ermittelt RNA-FM dennoch unbeaufsichtigt die in diesen RNA-Sequenzen enthaltenen evolutionären und strukturellen Muster.
Wenn RNA-FM effektiv auf nachgelagerte Aufgaben zur Vorhersage von RNA-Strukturen und -Funktionen angewendet werden kann, werden diese Berechnungsmethoden sicherlich von dem durch RNA-FM zusammengefassten Wissen profitieren und dadurch die Leistung verbessern. Das Upstream-Pre-Training und Downstream-Migrations- und Anwendungs-Framework von RNA-FM ist in der folgenden Abbildung dargestellt.
Forschungsübersicht
Um zu bestätigen, ob der vorab trainierte RNA-FM „Wissen“ aus einer großen Menge unbeschrifteter Daten gelernt hat und welche Art von „Wissen“ er gelernt hat, der Artikelführte eine Reihe von Einbettungstests durch. Analyse .
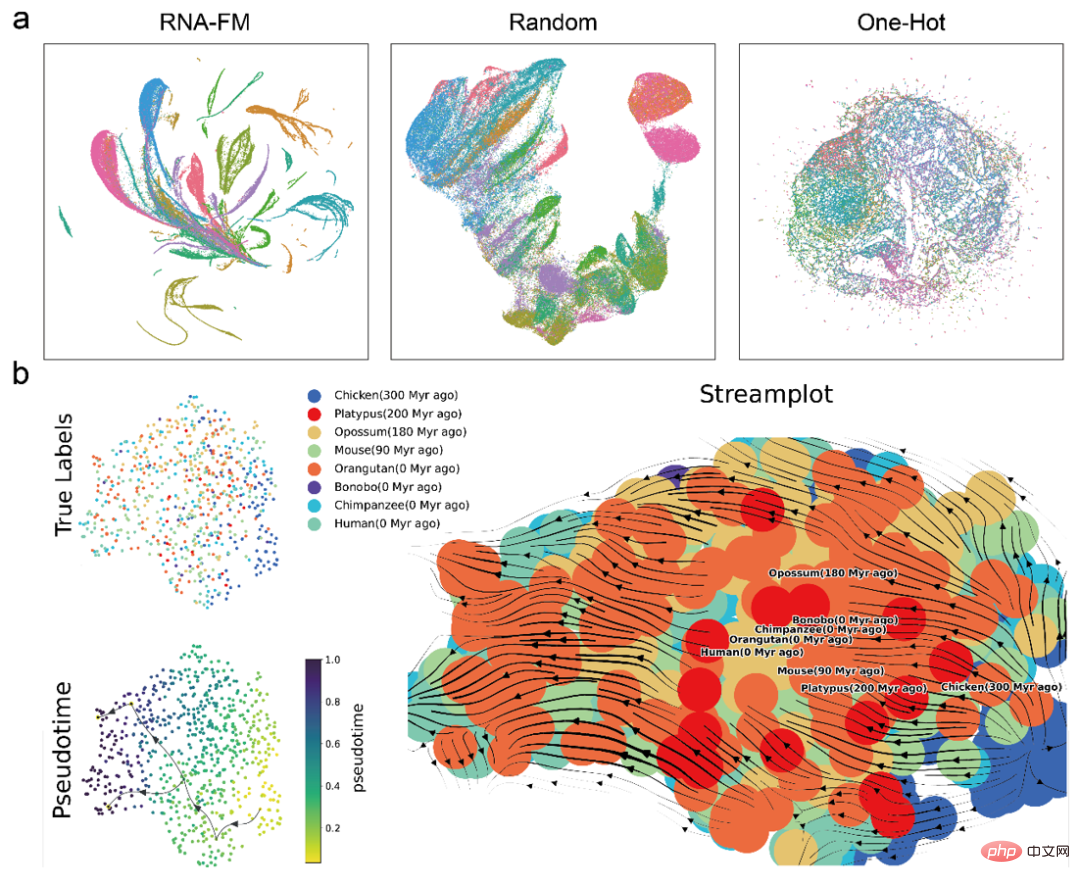
Zuerst wurde ein einfacher Clustering-Vergleich verschiedener Merkmale direkt über UMAP durchgeführt und es wurde festgestellt, dass die Einbettungen aus vorab trainiertem RNA-FM offensichtlichere RNA-Spezies-Cluster bildeten als andere Einbettungen. Dies bedeutet, dass die Einbettung von RNA-FM strukturelle oder funktionelle Informationen zur Unterscheidung von RNA-Arten enthält.
Dann der Artikel verwendet auch Trajektorieninferenz, um die Entwicklung von lncRNA aus verschiedenen Arten durch RNA-FM-Einbettung vorherzusagen. Aus dem folgenden Streamplot geht hervor, dass die vorhergesagte Pseudozeit der Evolution zwischen den Arten in etwa mit den tatsächlichen Evolutionsinformationen der Arten übereinstimmt, was darauf hindeutet, dass die RNA-FM-Einbettung auch einen Teil der Evolutionsinformationen enthält.
Es ist erwähnenswert, dass RNA-FM diesen Markierungen während des Trainings nicht direkt ausgesetzt war, unabhängig davon, ob es sich um Gemeinschaftsinformationen über RNA-Arten oder um evolutionäre Informationen über lncRNA handelt. RNA-FM entdeckt Muster im Zusammenhang mit Struktur, Funktion und Evolution aus reinen Sequenzen auf völlig selbstüberwachte Weise. Weitere experimentelle Ergebnisse Kontaktvorhersage, Abstandsvorhersage und Tertiärstrukturvorhersage haben alle erhebliche Verbesserungen erzielt
.
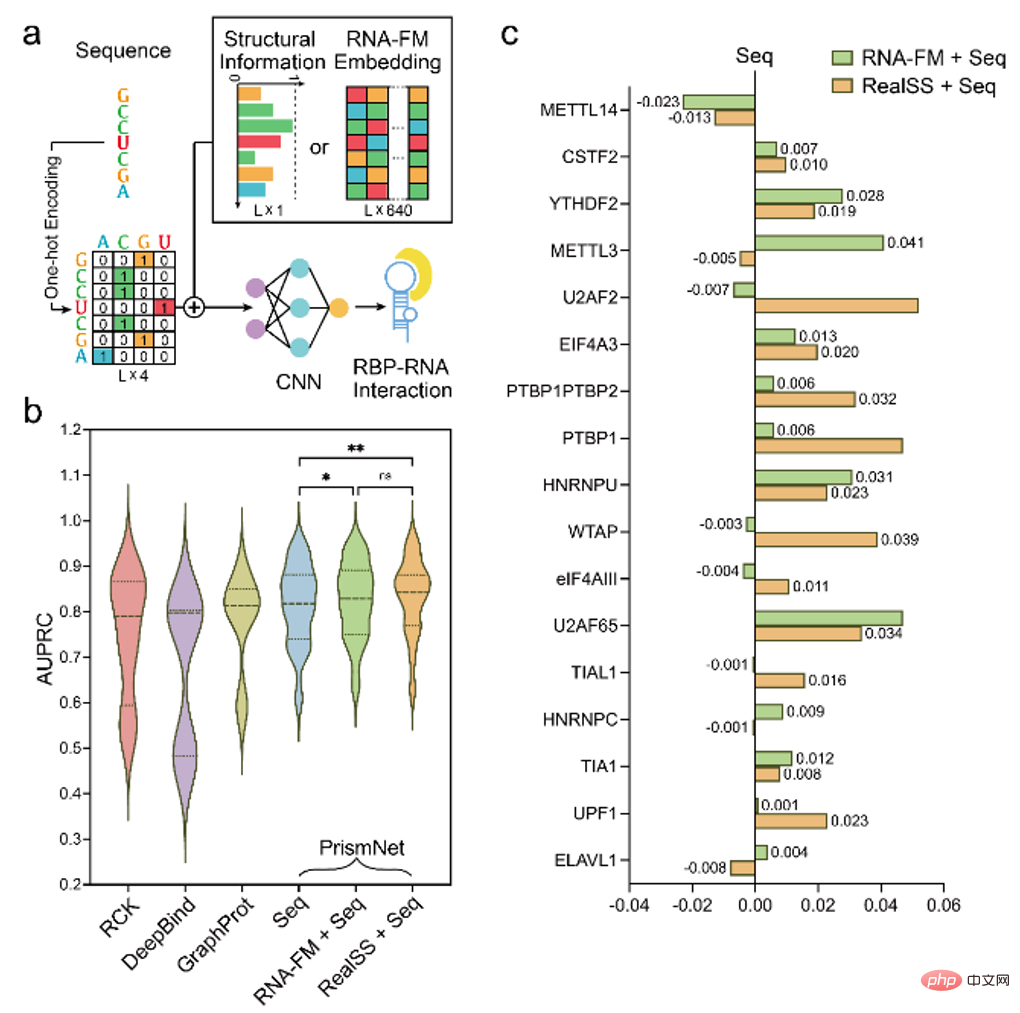
Insbesondere bei der Vorhersage der Sekundärstruktur verwendet der Artikel RNA-FM als Rückgrat und nur ein einfaches ResNet-Netzwerk als Downstream-Modell und übertrifft damit 12 andere State-of-the-State-Methoden für zwei öffentliche Datensätze ist 3-5 Prozentpunkte besser als das beste UFold im F1score. Im direkten Vergleich mit UFold übertrifft RNA-FM UFold in den meisten RNA-Kategorien. Wird RNA-FM mit E2Efold kombiniert, kann eine weitere Leistungssteigerung von 5 % erreicht werden.
Um den praktischen Anwendungswert des Modells zu überprüfen, verwendet der Artikel RNA-FM, um eine vollständige Analyse der COVID-19-Daten durchzuführen, einschließlich der Verwendung von RNA-FM zur genauen Vorhersage von COVID -19-Referenzgenom (29870 nt) wichtige regulatorische Elemente und nutzen die RNA-FM-Einbettung, um die Evolutionstrends der wichtigsten COVID-19-Varianten grob vorherzusagen.
Im Allgemeinen bestimmt die Struktur eines Moleküls seine Funktion. Da RNA-FM die Aufgabe der RNA-Strukturvorhersage hervorragend erfüllen kann, kann RNA-FM auch zur Verbesserung der Ergebnisse der Funktionsvorhersage verwendet werden?

Daher wird in dem Artikel weiterhin versucht, RNA-FM in nachgelagerte Aufgaben zur Vorhersage von RNA-Funktionen einzuführen, beispielsweise die Verwendung der RNA-FM-Einbettung zur Vorhersage von RNA-Protein-Rollen. 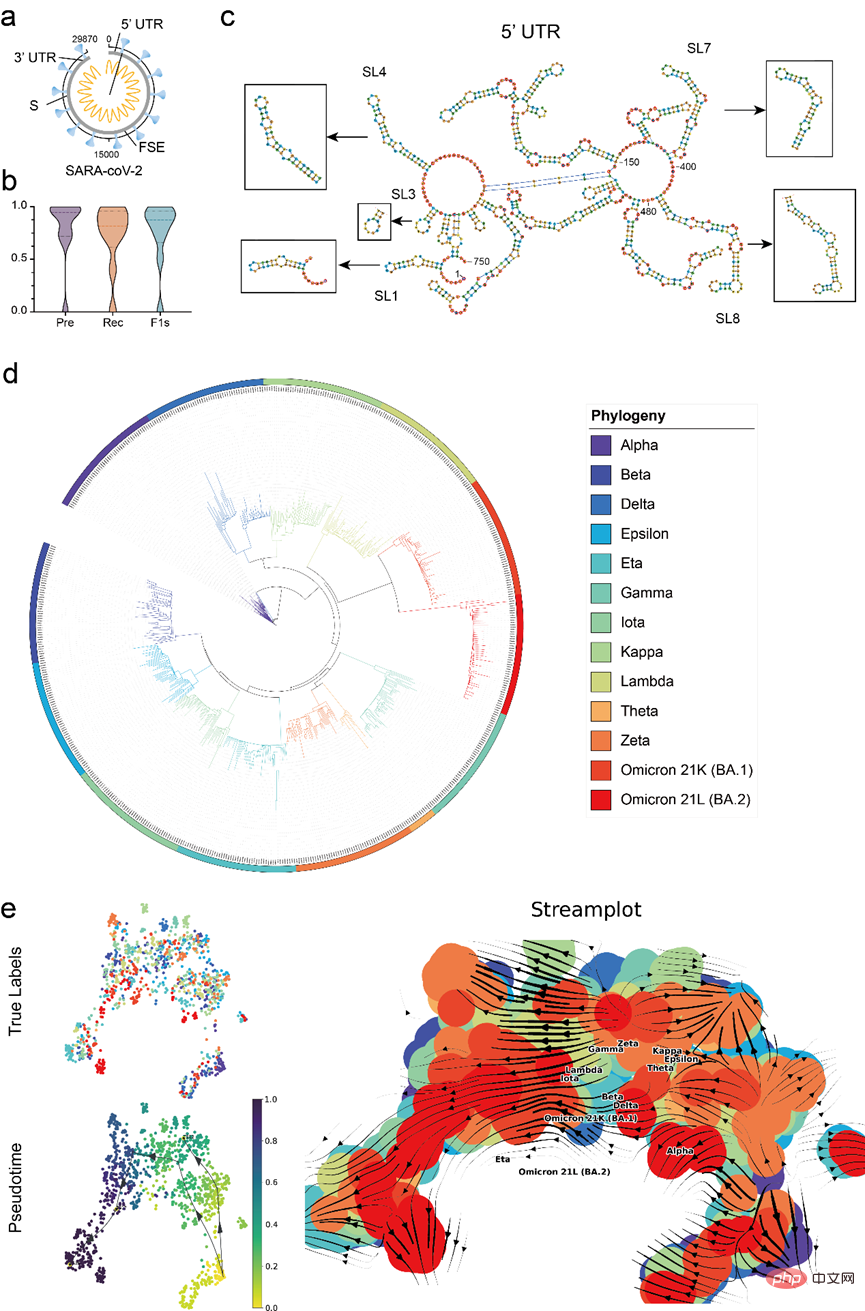
Um zu untersuchen, ob RNA-FM basierend auf ncRNA-Training auf andere RNAs übertragen werden kann, versucht der Artikelabschließend, RNA-FM zu verwenden, um die Funktion der Proteinexpression basierend auf der 5'UTR vorherzusagen auf mRNA. Obwohl mRNA nicht zu ncRNA gehört, ist die 5'UTR darauf eine Region, die nicht translatiert wird, aber regulatorische Funktionen hat, was mit den Eigenschaften von ncRNA übereinstimmt und nicht in den Trainingsdaten erscheint.
Wie Sie der Abbildung unten entnehmen können, sind Modelle mit RNA-FM-Einbettung immer besser als Modelle ohne. Obwohl die Leistungsverbesserung relativ begrenzt ist, zeigt sie teilweise, dass RNA-FM auch eine gewisse Generalisierungsfähigkeit für Nicht-ncRNA-Daten besitzt.
Fazit
Im Allgemeinen verwendet dieser Artikel unbeschriftete RNA-Sequenzdaten, um das Sprachmodell RNA-FM vorab zu trainieren, und zwar durch direkte oder indirekte Methoden in der Struktur oder Umfassend Die Verifizierung einer Reihe verschiedener Aufgaben wie Funktionen hat gezeigt, dass RNA-FM tatsächlich die Leistung von Rechenmethoden in nachgelagerten Aufgaben effektiv verbessern kann.
Das Aufkommen von RNA-FM hat die aktuelle Situation RNA-annotierter Daten in gewissem Maße gemildert und anderen Forschern einen bequemen Zugang zu großen Mengen unbeschrifteter Daten ermöglicht Die Schnittstelle wird als Basismodell im RNA-Bereich dienen und verschiedene Forschungsarbeiten auf diesem Gebiet umfassend unterstützen und unterstützen.
Über den Autor
Dieser Artikel hat zwei Co-Erstautoren. Chen Jiayang ist wissenschaftlicher Mitarbeiter an der Chinesischen Universität Hongkong. Hu Zhihang ist Doktorand an der Chinesischen Universität Hongkong.
Es gibt zwei korrespondierende Autoren dieses Artikels. Sun Siqi, junger Forscher am Intelligent Complex Systems Laboratory der Fudan University und am Shanghai Artificial Intelligence Laboratory, Homepage https://intersun.github.io.
Li Yu, Assistenzprofessorin, Chinesische Universität Hongkong, Visiting Assistant Professor, MIT James Collins Lab, Broad Institute of MIT und Harvard Research Scientist, Zu Besuch am Wyss Institute, Harvard University Scholar, Forbes 30 Under 30 Asia-Liste – Jahrgang 2022, Gesundheitswesen und Wissenschaft. Homepage: https://liyu95.com.
Das obige ist der detaillierte Inhalt vonOpen Source! Hong Kong Chinese, MIT und Fudan schlagen das erste RNA-Grundsteinmodell vor. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!

Heiße KI -Werkzeuge

Undresser.AI Undress
KI-gestützte App zum Erstellen realistischer Aktfotos

AI Clothes Remover
Online-KI-Tool zum Entfernen von Kleidung aus Fotos.

Undress AI Tool
Ausziehbilder kostenlos

Clothoff.io
KI-Kleiderentferner

AI Hentai Generator
Erstellen Sie kostenlos Ai Hentai.

Heißer Artikel

Heiße Werkzeuge

Notepad++7.3.1
Einfach zu bedienender und kostenloser Code-Editor

SublimeText3 chinesische Version
Chinesische Version, sehr einfach zu bedienen

Senden Sie Studio 13.0.1
Leistungsstarke integrierte PHP-Entwicklungsumgebung

Dreamweaver CS6
Visuelle Webentwicklungstools

SublimeText3 Mac-Version
Codebearbeitungssoftware auf Gottesniveau (SublimeText3)

Heiße Themen
 1378
1378
 52
52
 Das weltweit leistungsstärkste Open-Source-MoE-Modell ist da, mit chinesischen Fähigkeiten, die mit GPT-4 vergleichbar sind, und der Preis beträgt nur fast ein Prozent von GPT-4-Turbo
May 07, 2024 pm 04:13 PM
Das weltweit leistungsstärkste Open-Source-MoE-Modell ist da, mit chinesischen Fähigkeiten, die mit GPT-4 vergleichbar sind, und der Preis beträgt nur fast ein Prozent von GPT-4-Turbo
May 07, 2024 pm 04:13 PM
Stellen Sie sich ein Modell der künstlichen Intelligenz vor, das nicht nur die Fähigkeit besitzt, die traditionelle Datenverarbeitung zu übertreffen, sondern auch eine effizientere Leistung zu geringeren Kosten erzielt. Dies ist keine Science-Fiction, DeepSeek-V2[1], das weltweit leistungsstärkste Open-Source-MoE-Modell, ist da. DeepSeek-V2 ist ein leistungsstarkes MoE-Sprachmodell (Mix of Experts) mit den Merkmalen eines wirtschaftlichen Trainings und einer effizienten Inferenz. Es besteht aus 236B Parametern, von denen 21B zur Aktivierung jedes Markers verwendet werden. Im Vergleich zu DeepSeek67B bietet DeepSeek-V2 eine stärkere Leistung, spart gleichzeitig 42,5 % der Trainingskosten, reduziert den KV-Cache um 93,3 % und erhöht den maximalen Generierungsdurchsatz auf das 5,76-fache. DeepSeek ist ein Unternehmen, das sich mit allgemeiner künstlicher Intelligenz beschäftigt
 KI untergräbt die mathematische Forschung! Der Gewinner der Fields-Medaille und der chinesisch-amerikanische Mathematiker führten 11 hochrangige Arbeiten an | Gefällt mir bei Terence Tao
Apr 09, 2024 am 11:52 AM
KI untergräbt die mathematische Forschung! Der Gewinner der Fields-Medaille und der chinesisch-amerikanische Mathematiker führten 11 hochrangige Arbeiten an | Gefällt mir bei Terence Tao
Apr 09, 2024 am 11:52 AM
KI verändert tatsächlich die Mathematik. Vor kurzem hat Tao Zhexuan, der diesem Thema große Aufmerksamkeit gewidmet hat, die neueste Ausgabe des „Bulletin of the American Mathematical Society“ (Bulletin der American Mathematical Society) weitergeleitet. Zum Thema „Werden Maschinen die Mathematik verändern?“ äußerten viele Mathematiker ihre Meinung. Der gesamte Prozess war voller Funken, knallhart und aufregend. Der Autor verfügt über eine starke Besetzung, darunter der Fields-Medaillengewinner Akshay Venkatesh, der chinesische Mathematiker Zheng Lejun, der NYU-Informatiker Ernest Davis und viele andere bekannte Wissenschaftler der Branche. Die Welt der KI hat sich dramatisch verändert. Viele dieser Artikel wurden vor einem Jahr eingereicht.
 KAN, das MLP ersetzt, wurde durch Open-Source-Projekte auf Faltung erweitert
Jun 01, 2024 pm 10:03 PM
KAN, das MLP ersetzt, wurde durch Open-Source-Projekte auf Faltung erweitert
Jun 01, 2024 pm 10:03 PM
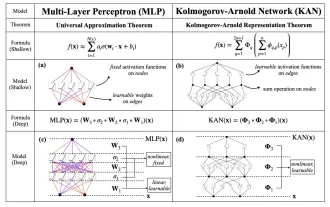
Anfang dieses Monats schlugen Forscher des MIT und anderer Institutionen eine vielversprechende Alternative zu MLP vor – KAN. KAN übertrifft MLP in Bezug auf Genauigkeit und Interpretierbarkeit. Und es kann MLP, das mit einer größeren Anzahl von Parametern ausgeführt wird, mit einer sehr kleinen Anzahl von Parametern übertreffen. Beispielsweise gaben die Autoren an, dass sie KAN nutzten, um die Ergebnisse von DeepMind mit einem kleineren Netzwerk und einem höheren Automatisierungsgrad zu reproduzieren. Konkret verfügt DeepMinds MLP über etwa 300.000 Parameter, während KAN nur etwa 200 Parameter hat. KAN hat eine starke mathematische Grundlage wie MLP und basiert auf dem universellen Approximationssatz, während KAN auf dem Kolmogorov-Arnold-Darstellungssatz basiert. Wie in der folgenden Abbildung gezeigt, hat KAN
 Google ist begeistert: JAX-Leistung übertrifft Pytorch und TensorFlow! Es könnte die schnellste Wahl für das GPU-Inferenztraining werden
Apr 01, 2024 pm 07:46 PM
Google ist begeistert: JAX-Leistung übertrifft Pytorch und TensorFlow! Es könnte die schnellste Wahl für das GPU-Inferenztraining werden
Apr 01, 2024 pm 07:46 PM
Die von Google geförderte Leistung von JAX hat in jüngsten Benchmark-Tests die von Pytorch und TensorFlow übertroffen und belegt bei 7 Indikatoren den ersten Platz. Und der Test wurde nicht auf der TPU mit der besten JAX-Leistung durchgeführt. Obwohl unter Entwicklern Pytorch immer noch beliebter ist als Tensorflow. Aber in Zukunft werden möglicherweise mehr große Modelle auf Basis der JAX-Plattform trainiert und ausgeführt. Modelle Kürzlich hat das Keras-Team drei Backends (TensorFlow, JAX, PyTorch) mit der nativen PyTorch-Implementierung und Keras2 mit TensorFlow verglichen. Zunächst wählen sie eine Reihe von Mainstream-Inhalten aus
 Hallo, elektrischer Atlas! Der Boston Dynamics-Roboter erwacht wieder zum Leben, seltsame 180-Grad-Bewegungen machen Musk Angst
Apr 18, 2024 pm 07:58 PM
Hallo, elektrischer Atlas! Der Boston Dynamics-Roboter erwacht wieder zum Leben, seltsame 180-Grad-Bewegungen machen Musk Angst
Apr 18, 2024 pm 07:58 PM
Boston Dynamics Atlas tritt offiziell in die Ära der Elektroroboter ein! Gestern hat sich der hydraulische Atlas einfach „unter Tränen“ von der Bühne der Geschichte zurückgezogen. Heute gab Boston Dynamics bekannt, dass der elektrische Atlas im Einsatz ist. Es scheint, dass Boston Dynamics im Bereich kommerzieller humanoider Roboter entschlossen ist, mit Tesla zu konkurrieren. Nach der Veröffentlichung des neuen Videos wurde es innerhalb von nur zehn Stunden bereits von mehr als einer Million Menschen angesehen. Die alten Leute gehen und neue Rollen entstehen. Das ist eine historische Notwendigkeit. Es besteht kein Zweifel, dass dieses Jahr das explosive Jahr der humanoiden Roboter ist. Netizens kommentierten: Die Weiterentwicklung der Roboter hat dazu geführt, dass die diesjährige Eröffnungsfeier wie Menschen aussieht, und der Freiheitsgrad ist weitaus größer als der von Menschen. Aber ist das wirklich kein Horrorfilm? Zu Beginn des Videos liegt Atlas ruhig auf dem Boden, scheinbar auf dem Rücken. Was folgt, ist atemberaubend
 Empfohlen: Ausgezeichnetes JS-Open-Source-Projekt zur Gesichtserkennung und -erkennung
Apr 03, 2024 am 11:55 AM
Empfohlen: Ausgezeichnetes JS-Open-Source-Projekt zur Gesichtserkennung und -erkennung
Apr 03, 2024 am 11:55 AM
Die Technologie zur Gesichtserkennung und -erkennung ist bereits eine relativ ausgereifte und weit verbreitete Technologie. Derzeit ist JS die am weitesten verbreitete Internetanwendungssprache. Die Implementierung der Gesichtserkennung und -erkennung im Web-Frontend hat im Vergleich zur Back-End-Gesichtserkennung Vor- und Nachteile. Zu den Vorteilen gehören die Reduzierung der Netzwerkinteraktion und die Echtzeiterkennung, was die Wartezeit des Benutzers erheblich verkürzt und das Benutzererlebnis verbessert. Die Nachteile sind: Es ist durch die Größe des Modells begrenzt und auch die Genauigkeit ist begrenzt. Wie implementiert man mit js die Gesichtserkennung im Web? Um die Gesichtserkennung im Web zu implementieren, müssen Sie mit verwandten Programmiersprachen und -technologien wie JavaScript, HTML, CSS, WebRTC usw. vertraut sein. Gleichzeitig müssen Sie auch relevante Technologien für Computer Vision und künstliche Intelligenz beherrschen. Dies ist aufgrund des Designs der Webseite erwähnenswert
 FisheyeDetNet: der erste Zielerkennungsalgorithmus basierend auf einer Fischaugenkamera
Apr 26, 2024 am 11:37 AM
FisheyeDetNet: der erste Zielerkennungsalgorithmus basierend auf einer Fischaugenkamera
Apr 26, 2024 am 11:37 AM
Die Zielerkennung ist ein relativ ausgereiftes Problem in autonomen Fahrsystemen, wobei die Fußgängererkennung einer der ersten Algorithmen ist, die eingesetzt werden. In den meisten Arbeiten wurde eine sehr umfassende Recherche durchgeführt. Die Entfernungswahrnehmung mithilfe von Fischaugenkameras für die Rundumsicht ist jedoch relativ wenig untersucht. Aufgrund der großen radialen Verzerrung ist es schwierig, die standardmäßige Bounding-Box-Darstellung in Fischaugenkameras zu implementieren. Um die obige Beschreibung zu vereinfachen, untersuchen wir erweiterte Begrenzungsrahmen-, Ellipsen- und allgemeine Polygondesigns in Polar-/Winkeldarstellungen und definieren eine mIOU-Metrik für die Instanzsegmentierung, um diese Darstellungen zu analysieren. Das vorgeschlagene Modell „fisheyeDetNet“ mit polygonaler Form übertrifft andere Modelle und erreicht gleichzeitig 49,5 % mAP auf dem Valeo-Fisheye-Kameradatensatz für autonomes Fahren
 Tesla-Roboter arbeiten in Fabriken, Musk: Der Freiheitsgrad der Hände wird dieses Jahr 22 erreichen!
May 06, 2024 pm 04:13 PM
Tesla-Roboter arbeiten in Fabriken, Musk: Der Freiheitsgrad der Hände wird dieses Jahr 22 erreichen!
May 06, 2024 pm 04:13 PM
Das neueste Video von Teslas Roboter Optimus ist veröffentlicht und er kann bereits in der Fabrik arbeiten. Bei normaler Geschwindigkeit sortiert es Batterien (Teslas 4680-Batterien) so: Der Beamte hat auch veröffentlicht, wie es bei 20-facher Geschwindigkeit aussieht – auf einer kleinen „Workstation“, pflücken und pflücken und pflücken: Dieses Mal wird es freigegeben. Eines der Highlights Der Vorteil des Videos besteht darin, dass Optimus diese Arbeit in der Fabrik völlig autonom und ohne menschliches Eingreifen während des gesamten Prozesses erledigt. Und aus Sicht von Optimus kann es auch die krumme Batterie aufnehmen und platzieren, wobei der Schwerpunkt auf der automatischen Fehlerkorrektur liegt: In Bezug auf die Hand von Optimus gab der NVIDIA-Wissenschaftler Jim Fan eine hohe Bewertung ab: Die Hand von Optimus ist der fünffingrige Roboter der Welt am geschicktesten. Seine Hände sind nicht nur taktil



