 Technologie-Peripheriegeräte
Technologie-Peripheriegeräte
 KI
KI
 Unendliche Möglichkeiten Das generative KI-Modell von NVIDIA beschleunigt die Proteinsynthese
Unendliche Möglichkeiten Das generative KI-Modell von NVIDIA beschleunigt die Proteinsynthese
Unendliche Möglichkeiten Das generative KI-Modell von NVIDIA beschleunigt die Proteinsynthese
In den letzten zwei Jahren hat maschinelles Lernen die Vorhersage der Proteinstruktur revolutioniert. Jetzt hat künstliche Intelligenz eine neue Revolution im Bereich des Proteindesigns ausgelöst.
Seit dem Aufkommen der KI haben sich viele Wissenschaftler dazu entschlossen, sie für die Proteinforschung zu nutzen. Biologen haben herausgefunden, dass mithilfe maschinellen Lernens Proteinmoleküle in Sekundenschnelle erzeugt werden können. Früher konnte diese Zeit mehrere Monate betragen.
Kürzlich nutzte das Start-up Evozyne vorab trainierte KI-Modelle von NVIDIA, um zwei Proteine mit erheblichem Potenzial in den Bereichen Medizin und saubere Energie zu entwickeln. Eines der Proteine wird zur Behandlung einer angeborenen Krankheit verwendet, ein anderes zum Verbrauch von Kohlendioxid, um die globale Erwärmung zu reduzieren.
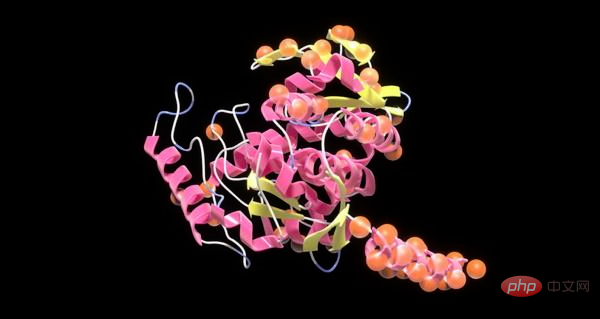
Auf diese Weise nutzen Wissenschaftler NVIDIA BioNeMo, um groß angelegte Sprachmodelle zu erstellen, die hochwertige Proteine erzeugen können, um die Arzneimittelentwicklung zu beschleunigen und zur Schaffung einer nachhaltigeren Umwelt beizutragen.
Ein neuer Weg zur Beschleunigung der Arzneimittelforschung
Andrew Ferguson, Mitbegründer von Evozyne und Mitautor des Papiers, sagte: „Erfreulich ist, dass die synthetischen Proteine, die dieses KI-Modell in der ersten Runde produziert hat, es sind.“ Genau wie natürlich vorkommende Proteine. Zeigt an, dass das Modell die Designregeln der Natur gelernt hat.“
Evozyne verwendet NVIDIAs ProtT5. ProtT5 ist ein Transformer-Modell, das Teil von NVIDIA BioNeMo ist, einem Software-Framework und Dienst zur Erstellung medizinischer KI-Modelle.
Der Molekularingenieur Ferguson, dessen Forschungsgebiete die Bereiche Chemie und maschinelles Lernen umfassen, sagte: „BioNeMo ist sehr leistungsstark und ermöglicht es uns, ein Modell zu trainieren und es dann zu sehr geringen Kosten zum Ausführen von Arbeitsaufgaben zu verwenden.“ „Generieren Sie Millionen von Sequenzen.“
Dieses Modell ist das Herzstück der Evovyne ProT-VAE-Pipeline. Die ProT-VAE-Pipeline von Evozyne kombiniert das leistungsstarke Transformer-Modell in NVIDIA BioNeMo mit Variational Autoencodern (VAEs).
Er sagte: „Vor ein paar Jahren ist niemandem aufgefallen, dass Proteine mithilfe großer Sprachmodelle in Kombination mit Variations-Autoencodern entworfen werden können.“
Im Gegensatz dazu durchläuft die Methode von Evozyne nur eine Runde. Sie kann die Hälfte oder mehr davon verändern Aminosäuren in einem Protein. Dies entspricht Hunderten von Mutationen.
Evozyne-Datenwissenschaftler Joshua Moller sagte: „Sie beschleunigen das Training, indem sie die Arbeit auf mehrere GPUs ausweiten.
Dadurch verkürzt sich die Zeit zum Trainieren eines großen KI-Modells von Monaten auf eine Woche. So können wir Modelle trainieren, die sonst unmöglich wären.“ zu trainieren, beispielsweise einige mit Milliarden trainierbarer Parameter. „
Revolutionäres KI-Modell
Die traditionelle Protein-Engineering-Designmethode, nämlich die gerichtete Evolution, verwendet einen langsamen, ungeplanten Ansatz, der normalerweise nur die Reihenfolge einiger weniger Aminosäuren gleichzeitig ändert. Maschinelles Lernen hilft dabei, massiv zu studieren Aminosäurekombinationen und identifizieren dann effizient die nützlichsten Sequenzen
BioNeMo ist ein KI-fähiger Cloud-Dienst und ein Framework für die Arzneimittelentwicklung, das auf dem KI-Modell „Biomolecule Transformer“ für das Training und die Bereitstellung groß angelegter Anwendungen basiert vorab trainiertes LLM, native Unterstützung für gängige Dateiformate für Proteine, DNA, RNA und Chemie und bietet auch Unterstützung für SMILES (für molekulare Strukturen) und FASTA (für Aminosäure- und Nukleotidsequenzen) mithilfe des Datenladers
Mit BioNeMo können Wissenschaftler ganz einfach vorab trainierte Modelle, automatische Downloader und Präprozessoren für UniRef50- und ZINC-Datenbanken mit verschiedenen Modellen, Einbettungen und Ausgaben verwenden. Durch das unbeaufsichtigte Vortraining sind auch gekennzeichnete Daten nicht mehr erforderlich, um schnell gelernte Einbettungen zu generieren, die Proteine vorhersagen Struktur, Funktion, Zellstandort, Wasserlöslichkeit und Membranbindung, Speicherbereich und variabler Bereich usw.
Daunter ist MegaMolBART ein mit 1,4 Milliarden Molekülen trainiertes generatives Chemiemodell (SMILES-String), das für a verwendet werden kann Eine Vielzahl chemischer Informatikanwendungen, und BioNeMo bietet ProtT5 und ESM1 sowie andere Transformer-basierte Proteinsprachenmodelle.
BioNeMo bietet auch OpenFold, ein Deep-Learning-Modell zur Vorhersage der 3D-Struktur neuartiger Proteinsequenzen liest Millionen von Proteinen. Dieses Modell nutzt die Technologie, die neuronale Netze verwenden, um Texte zu verstehen, und lernt, wie die Natur Protein-Aminosäuresequenzen konstruiert Im Vergleich zu den ursprünglich in der Natur vorkommenden Proteinen ist es stabiler und kann unter extremen Bedingungen wie Energiemangel oder hoher Temperatur eine seiner Funktionen erfüllen
.Darüber hinaus können mithilfe künstlicher Intelligenz auch Aminosäuresequenzen passend zum Rückgrat entworfen werden, wodurch die Stabilität bestimmter Proteine wie Enzyme und Antikörper verbessert werden kann. Künstliche Intelligenz spielt eine sehr wichtige Rolle bei der Entwicklung von Proteinen unterschiedlicher Größe und unterschiedlicher Konformation. In Zukunft kann sie auch dazu beitragen, immer mehr nützliche Proteine zu entwickeln, einschließlich neuer biologischer Materialien, die zur Reduzierung der Umweltverschmutzung und zur Verbesserung der Umwelt eingesetzt werden können .
Das obige ist der detaillierte Inhalt vonUnendliche Möglichkeiten Das generative KI-Modell von NVIDIA beschleunigt die Proteinsynthese. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!

Heiße KI -Werkzeuge

Undresser.AI Undress
KI-gestützte App zum Erstellen realistischer Aktfotos

AI Clothes Remover
Online-KI-Tool zum Entfernen von Kleidung aus Fotos.

Undress AI Tool
Ausziehbilder kostenlos

Clothoff.io
KI-Kleiderentferner

AI Hentai Generator
Erstellen Sie kostenlos Ai Hentai.

Heißer Artikel

Heiße Werkzeuge

Notepad++7.3.1
Einfach zu bedienender und kostenloser Code-Editor

SublimeText3 chinesische Version
Chinesische Version, sehr einfach zu bedienen

Senden Sie Studio 13.0.1
Leistungsstarke integrierte PHP-Entwicklungsumgebung

Dreamweaver CS6
Visuelle Webentwicklungstools

SublimeText3 Mac-Version
Codebearbeitungssoftware auf Gottesniveau (SublimeText3)

Heiße Themen
 1382
1382
 52
52
 CentOS Shutdown -Befehlszeile
Apr 14, 2025 pm 09:12 PM
CentOS Shutdown -Befehlszeile
Apr 14, 2025 pm 09:12 PM
Der Befehl centOS stilldown wird heruntergefahren und die Syntax wird von [Optionen] ausgeführt [Informationen]. Zu den Optionen gehören: -h das System sofort stoppen; -P schalten Sie die Leistung nach dem Herunterfahren aus; -r neu starten; -t Wartezeit. Zeiten können als unmittelbar (jetzt), Minuten (Minuten) oder als bestimmte Zeit (HH: MM) angegeben werden. Hinzugefügten Informationen können in Systemmeldungen angezeigt werden.
 Was sind die Backup -Methoden für Gitlab auf CentOS?
Apr 14, 2025 pm 05:33 PM
Was sind die Backup -Methoden für Gitlab auf CentOS?
Apr 14, 2025 pm 05:33 PM
Backup- und Wiederherstellungsrichtlinie von GitLab im Rahmen von CentOS -System Um die Datensicherheit und Wiederherstellung der Daten zu gewährleisten, bietet GitLab on CentOS eine Vielzahl von Sicherungsmethoden. In diesem Artikel werden mehrere gängige Sicherungsmethoden, Konfigurationsparameter und Wiederherstellungsprozesse im Detail eingeführt, um eine vollständige GitLab -Sicherungs- und Wiederherstellungsstrategie aufzubauen. 1. Manuell Backup Verwenden Sie den GitLab-RakegitLab: Backup: Befehl erstellen, um die manuelle Sicherung auszuführen. Dieser Befehl unterstützt wichtige Informationen wie GitLab Repository, Datenbank, Benutzer, Benutzergruppen, Schlüssel und Berechtigungen. Die Standardsicherungsdatei wird im Verzeichnis/var/opt/gitlab/backups gespeichert. Sie können /etc /gitlab ändern
 So überprüfen Sie die CentOS -HDFS -Konfiguration
Apr 14, 2025 pm 07:21 PM
So überprüfen Sie die CentOS -HDFS -Konfiguration
Apr 14, 2025 pm 07:21 PM
Vollständige Anleitung zur Überprüfung der HDFS -Konfiguration in CentOS -Systemen In diesem Artikel wird die Konfiguration und den laufenden Status von HDFS auf CentOS -Systemen effektiv überprüft. Die folgenden Schritte helfen Ihnen dabei, das Setup und den Betrieb von HDFs vollständig zu verstehen. Überprüfen Sie die Hadoop -Umgebungsvariable: Stellen Sie zunächst sicher, dass die Hadoop -Umgebungsvariable korrekt eingestellt ist. Führen Sie im Terminal den folgenden Befehl aus, um zu überprüfen, ob Hadoop ordnungsgemäß installiert und konfiguriert ist: Hadoopsion-Check HDFS-Konfigurationsdatei: Die Kernkonfigurationsdatei von HDFS befindet sich im/etc/hadoop/conf/verzeichnis, wobei core-site.xml und hdfs-site.xml von entscheidender Bedeutung sind. verwenden
 Wie ist die GPU -Unterstützung für Pytorch bei CentOS?
Apr 14, 2025 pm 06:48 PM
Wie ist die GPU -Unterstützung für Pytorch bei CentOS?
Apr 14, 2025 pm 06:48 PM
Aktivieren Sie die Pytorch -GPU -Beschleunigung am CentOS -System erfordert die Installation von CUDA-, CUDNN- und GPU -Versionen von Pytorch. Die folgenden Schritte führen Sie durch den Prozess: Cuda und Cudnn Installation Bestimmen Sie die CUDA-Version Kompatibilität: Verwenden Sie den Befehl nvidia-smi, um die von Ihrer NVIDIA-Grafikkarte unterstützte CUDA-Version anzuzeigen. Beispielsweise kann Ihre MX450 -Grafikkarte CUDA11.1 oder höher unterstützen. Download und installieren Sie Cudatoolkit: Besuchen Sie die offizielle Website von Nvidiacudatoolkit und laden Sie die entsprechende Version gemäß der höchsten CUDA -Version herunter und installieren Sie sie, die von Ihrer Grafikkarte unterstützt wird. Installieren Sie die Cudnn -Bibliothek:
 Detaillierte Erklärung des Docker -Prinzips
Apr 14, 2025 pm 11:57 PM
Detaillierte Erklärung des Docker -Prinzips
Apr 14, 2025 pm 11:57 PM
Docker verwendet Linux -Kernel -Funktionen, um eine effiziente und isolierte Anwendungsumgebung zu bieten. Sein Arbeitsprinzip lautet wie folgt: 1. Der Spiegel wird als schreibgeschützte Vorlage verwendet, die alles enthält, was Sie für die Ausführung der Anwendung benötigen. 2. Das Union File System (UnionFS) stapelt mehrere Dateisysteme, speichert nur die Unterschiede, speichert Platz und beschleunigt. 3. Der Daemon verwaltet die Spiegel und Container, und der Kunde verwendet sie für die Interaktion. 4. Namespaces und CGroups implementieren Container -Isolation und Ressourcenbeschränkungen; 5. Mehrere Netzwerkmodi unterstützen die Containerverbindung. Nur wenn Sie diese Kernkonzepte verstehen, können Sie Docker besser nutzen.
 CentOS installieren MySQL
Apr 14, 2025 pm 08:09 PM
CentOS installieren MySQL
Apr 14, 2025 pm 08:09 PM
Die Installation von MySQL auf CentOS umfasst die folgenden Schritte: Hinzufügen der entsprechenden MySQL Yum -Quelle. Führen Sie den Befehl mySQL-server aus, um den MySQL-Server zu installieren. Verwenden Sie den Befehl mySQL_SECURE_INSTALLATION, um Sicherheitseinstellungen vorzunehmen, z. B. das Festlegen des Stammbenutzerkennworts. Passen Sie die MySQL -Konfigurationsdatei nach Bedarf an. Tune MySQL -Parameter und optimieren Sie Datenbanken für die Leistung.
 CentOS8 startet SSH
Apr 14, 2025 pm 09:00 PM
CentOS8 startet SSH
Apr 14, 2025 pm 09:00 PM
Der Befehl zum Neustart des SSH -Dienstes lautet: SystemCTL Neustart SSHD. Detaillierte Schritte: 1. Zugriff auf das Terminal und eine Verbindung zum Server; 2. Geben Sie den Befehl ein: SystemCTL Neustart SSHD; 1. Überprüfen Sie den Dienststatus: SystemCTL -Status SSHD.
 Wie man eine verteilte Schulung von Pytorch auf CentOS betreibt
Apr 14, 2025 pm 06:36 PM
Wie man eine verteilte Schulung von Pytorch auf CentOS betreibt
Apr 14, 2025 pm 06:36 PM
Pytorch Distributed Training on CentOS -System erfordert die folgenden Schritte: Pytorch -Installation: Die Prämisse ist, dass Python und PIP im CentOS -System installiert sind. Nehmen Sie abhängig von Ihrer CUDA -Version den entsprechenden Installationsbefehl von der offiziellen Pytorch -Website ab. Für CPU-Schulungen können Sie den folgenden Befehl verwenden: PipinstallTorChTorChVisionTorChaudio Wenn Sie GPU-Unterstützung benötigen, stellen Sie sicher, dass die entsprechende Version von CUDA und CUDNN installiert ist und die entsprechende Pytorch-Version für die Installation verwenden. Konfiguration der verteilten Umgebung: Verteiltes Training erfordert in der Regel mehrere Maschinen oder mehrere Maschinen-Mehrfach-GPUs. Ort



