 Technologie-Peripheriegeräte
Technologie-Peripheriegeräte
 KI
KI
 1200-mal schneller! Das MIT entwickelt eine neue Generation der KI für die Arzneimittelforschung und -entwicklung, um das alte Modell zu überwinden
1200-mal schneller! Das MIT entwickelt eine neue Generation der KI für die Arzneimittelforschung und -entwicklung, um das alte Modell zu überwinden
1200-mal schneller! Das MIT entwickelt eine neue Generation der KI für die Arzneimittelforschung und -entwicklung, um das alte Modell zu überwinden
Wie wir alle wissen, ist das gesamte Universum mit unzähligen Molekülen gefüllt.
Wie viele dieser Moleküle haben potenziell arzneimittelähnliche Eigenschaften und können zur Entwicklung lebensrettender Medikamente genutzt werden? Ist es eine Million? Oder Milliarden? Oder Billionen?
Die Antwort lautet: 10 hoch 60.
Eine solch große Zahl hat den Fortschritt der Forschung und Entwicklung neuer Medikamente erheblich verzögert. Für sich schnell ausbreitende Krankheiten wie COVID-19 gibt es derzeit kein spezifisches Medikament. Dies liegt auch daran, dass die Arten und Mengen der Moleküle zu groß sind Der Rechenbereich geht weit über das hinaus, was bestehende Arzneimitteldesignmodelle leisten können.
Ein Forschungsteam am MIT glaubt nicht an dieses Übel. Es klappt nicht, oder? Dann ist es doch in Ordnung, das Vorgängermodell zu beschleunigen, oder?
Diese Beschleunigung beträgt das 1200-fache.
Sie untersuchten ein geometrisches Deep-Learning-Modell namens „EquiBind“, das 1.200-mal schneller ist als das bisher schnellste rechnerische Molekular-Docking-Modell „QuickVina2-W“, und kombinierten erfolgreich arzneimittelähnliche Moleküle mit Proteinen Scheitern einer Medikamentenstudie.
Das Forschungspapier wird auf der ICML 2022 veröffentlicht.
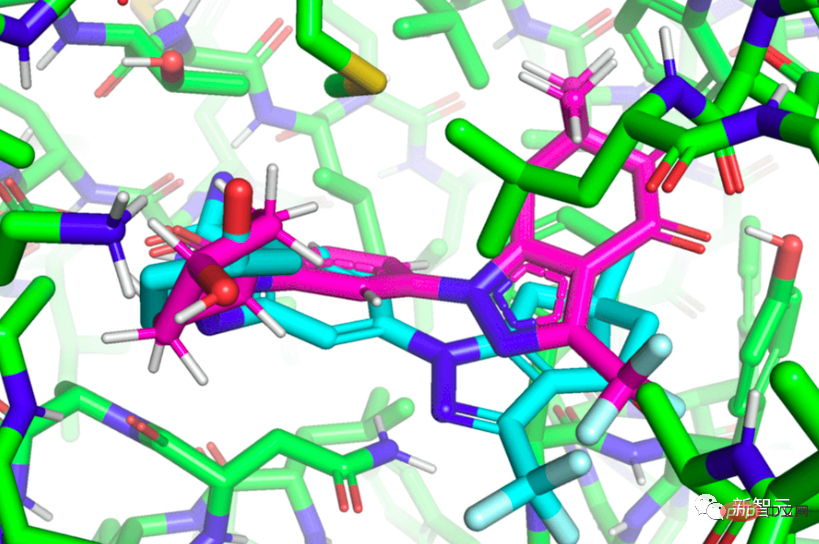
Erste Einführung in „EquiBind“
„EquiBind“ basiert auf seinem Vorgänger „EquiDock“. „EquiDock“ nutzt die vom verstorbenen MIT-KI-Forscher Octavian-Eugen Ganea entwickelte Technologie, um die beiden Arten von Proteinen zu kombinieren. Ganea ist außerdem Co-Autorin des „EquiBind“-Artikels.
Bevor mit der Arzneimittelentwicklung begonnen werden kann, müssen Forscher vielversprechende wirkstoffähnliche Moleküle finden, die während des Arzneimittelentwicklungsprozesses korrekt an bestimmte Proteinziele binden oder „andocken“ können.
Nach erfolgreichem Andocken an das Protein kann die Kombination des Arzneimittels (Ligand) die Funktion des Proteins beeinträchtigen. Geschieht dies mit einem der lebenswichtigen Proteine der Bakterien, kann es die Bakterien abtöten und so den Körper schützen.
Der Prozess der Arzneimittelentwicklung kann jedoch sowohl aus wirtschaftlicher als auch aus rechnerischer Sicht kostspielig sein. Der Forschungs- und Entwicklungsprozess kann bis zur endgültigen Zulassung und Prüfung durch die FDA mehr als zehn Jahre dauern.
Noch wichtiger ist, dass 90 % der Medikamente nach Versuchen am Menschen scheitern, weil sie keine Wirkung zeigen oder zu viele Nebenwirkungen haben.
Eine Möglichkeit für Pharmaunternehmen, diese Kosten auszugleichen, besteht also darin, den Preis des Medikaments zu erhöhen, das sie letztendlich erfolgreich entwickeln.
Steigen Sie ein in „EquiBind“
Derzeit läuft der Berechnungsprozess zum Auffinden vielversprechender Wirkstoffkandidatenmoleküle wie folgt ab: Die meisten der fortschrittlichsten Rechenmodelle basieren auf einer großen Anzahl von Kandidatenproben, gekoppelt mit Bewertung, Rangfolge und Feinabstimmung. Tuning usw. Methode, um die beste „Übereinstimmung“ zwischen Ligand und Protein zu erhalten.
Hannes Stärk, ein Doktorand im ersten Jahr an der Fakultät für Elektrotechnik und Informatik des MIT und Hauptautor dieser Arbeit, verglich die typische „Ligand-Protein“-Bindungsmethode mit dem „Versuch, einen Schlüssel in ein Schlüsselloch zu stecken“. „Gesperrt“.

Typische Models verbringen Zeit damit, jede „Passform“ zu bewerten, bevor sie das beste Modell auswählen. Im Gegensatz dazu muss „EquiBind“ die Zieltasche des Proteins nicht im Voraus kennen und kann die genaue Schlüsselposition in nur einem Schritt, dem sogenannten „Blind Docking“, direkt vorhersagen.
Im Gegensatz zu den meisten Modellen, die mehrere Versuche erfordern, um die günstige Position eines Liganden in einem Protein zu finden, verfügt „EquiBind“ bereits über integrierte Funktionen zum geometrischen Denken, die dem Modell helfen, die zugrunde liegenden physikalischen Eigenschaften des Moleküls zu lernen und erfolgreich zu verallgemeinern. erfolgreich zu verallgemeinern, um bessere Vorhersagen zu treffen, wenn auf neue oder nicht erkennbare Daten gestoßen wird.
Die Veröffentlichung dieser Ergebnisse erregte schnell die Aufmerksamkeit von Branchenexperten, darunter Pat Walters, Chief Data Officer von Relay Therapeutics.
Walters schlug vor, dass das Forschungsteam sein Modell an einem bestehenden Medikament und Protein ausprobieren könnte, das bei Lungenkrebs, Leukämie und Magen-Darm-Tumoren eingesetzt wird. Obwohl es den meisten herkömmlichen Docking-Methoden nicht gelingt, Liganden an diese Proteine erfolgreich zu binden, ist EquiBind erfolgreich.
Walters sagte: „EquiBind bietet eine einzigartige Lösung für das Andockproblem, indem es Posenvorhersage und Bindungsstellenidentifizierung kombiniert.“
„Und diese Methode nutzt Informationen aus Tausenden veröffentlichten Kristallstrukturen.“ Wir waren überrascht, dass EquiBind es schaffen konnte „in der richtigen Tasche, wenn alles andere völlig falsch war oder nur eines richtig war“, sagte Stärk. Wir sind sehr begeistert von diesem Ergebnis! , und dieses Feedback hat dem Team geholfen, über den praktischen Einsatz des Berechnungsmodells nachzudenken. Stärk hofft immer noch, beim kommenden ICML im Juli eine andere Perspektive zu finden.
Stärk sagte: „Das Feedback, auf das ich mich am meisten freue, sind Vorschläge, wie wir das Modell weiter verbessern können.“
„Ich würde gerne mit diesen Forschern diskutieren, ihnen sagen, was meiner Meinung nach die nächsten Schritte sein könnten, und sie ermutigen, weiterzumachen und das Modell in ihren eigenen Arbeiten und Methoden zu verwenden. Wir haben bereits viele Forscher kontaktiert, um uns zu fragen.“ Wird dieses Modell für ihr Problem nützlich sein?
Darüber hinaus ist dieser Artikel auch eine Erinnerung an Octavian-Eugen Gane, der entscheidende Beiträge zur geometrischen maschinellen Lernforschung leistete und viele Studenten großzügig betreute. Er war ein herausragender Gelehrter mit einer bescheidenen Seele .
In der ersten Hälfte dieses Jahres hat er uns während einer Wandertour für immer verlassen.
Das obige ist der detaillierte Inhalt von1200-mal schneller! Das MIT entwickelt eine neue Generation der KI für die Arzneimittelforschung und -entwicklung, um das alte Modell zu überwinden. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!

Heiße KI -Werkzeuge

Undresser.AI Undress
KI-gestützte App zum Erstellen realistischer Aktfotos

AI Clothes Remover
Online-KI-Tool zum Entfernen von Kleidung aus Fotos.

Undress AI Tool
Ausziehbilder kostenlos

Clothoff.io
KI-Kleiderentferner

AI Hentai Generator
Erstellen Sie kostenlos Ai Hentai.

Heißer Artikel

Heiße Werkzeuge

Notepad++7.3.1
Einfach zu bedienender und kostenloser Code-Editor

SublimeText3 chinesische Version
Chinesische Version, sehr einfach zu bedienen

Senden Sie Studio 13.0.1
Leistungsstarke integrierte PHP-Entwicklungsumgebung

Dreamweaver CS6
Visuelle Webentwicklungstools

SublimeText3 Mac-Version
Codebearbeitungssoftware auf Gottesniveau (SublimeText3)

Heiße Themen
 1377
1377
 52
52
 Das weltweit leistungsstärkste Open-Source-MoE-Modell ist da, mit chinesischen Fähigkeiten, die mit GPT-4 vergleichbar sind, und der Preis beträgt nur fast ein Prozent von GPT-4-Turbo
May 07, 2024 pm 04:13 PM
Das weltweit leistungsstärkste Open-Source-MoE-Modell ist da, mit chinesischen Fähigkeiten, die mit GPT-4 vergleichbar sind, und der Preis beträgt nur fast ein Prozent von GPT-4-Turbo
May 07, 2024 pm 04:13 PM
Stellen Sie sich ein Modell der künstlichen Intelligenz vor, das nicht nur die Fähigkeit besitzt, die traditionelle Datenverarbeitung zu übertreffen, sondern auch eine effizientere Leistung zu geringeren Kosten erzielt. Dies ist keine Science-Fiction, DeepSeek-V2[1], das weltweit leistungsstärkste Open-Source-MoE-Modell, ist da. DeepSeek-V2 ist ein leistungsstarkes MoE-Sprachmodell (Mix of Experts) mit den Merkmalen eines wirtschaftlichen Trainings und einer effizienten Inferenz. Es besteht aus 236B Parametern, von denen 21B zur Aktivierung jedes Markers verwendet werden. Im Vergleich zu DeepSeek67B bietet DeepSeek-V2 eine stärkere Leistung, spart gleichzeitig 42,5 % der Trainingskosten, reduziert den KV-Cache um 93,3 % und erhöht den maximalen Generierungsdurchsatz auf das 5,76-fache. DeepSeek ist ein Unternehmen, das sich mit allgemeiner künstlicher Intelligenz beschäftigt
 KI untergräbt die mathematische Forschung! Der Gewinner der Fields-Medaille und der chinesisch-amerikanische Mathematiker führten 11 hochrangige Arbeiten an | Gefällt mir bei Terence Tao
Apr 09, 2024 am 11:52 AM
KI untergräbt die mathematische Forschung! Der Gewinner der Fields-Medaille und der chinesisch-amerikanische Mathematiker führten 11 hochrangige Arbeiten an | Gefällt mir bei Terence Tao
Apr 09, 2024 am 11:52 AM
KI verändert tatsächlich die Mathematik. Vor kurzem hat Tao Zhexuan, der diesem Thema große Aufmerksamkeit gewidmet hat, die neueste Ausgabe des „Bulletin of the American Mathematical Society“ (Bulletin der American Mathematical Society) weitergeleitet. Zum Thema „Werden Maschinen die Mathematik verändern?“ äußerten viele Mathematiker ihre Meinung. Der gesamte Prozess war voller Funken, knallhart und aufregend. Der Autor verfügt über eine starke Besetzung, darunter der Fields-Medaillengewinner Akshay Venkatesh, der chinesische Mathematiker Zheng Lejun, der NYU-Informatiker Ernest Davis und viele andere bekannte Wissenschaftler der Branche. Die Welt der KI hat sich dramatisch verändert. Viele dieser Artikel wurden vor einem Jahr eingereicht.
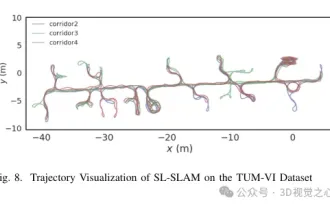 Jenseits von ORB-SLAM3! SL-SLAM: Szenen mit wenig Licht, starkem Jitter und schwacher Textur werden verarbeitet
May 30, 2024 am 09:35 AM
Jenseits von ORB-SLAM3! SL-SLAM: Szenen mit wenig Licht, starkem Jitter und schwacher Textur werden verarbeitet
May 30, 2024 am 09:35 AM
Heute diskutieren wir darüber, wie Deep-Learning-Technologie die Leistung von visionbasiertem SLAM (Simultaneous Localization and Mapping) in komplexen Umgebungen verbessern kann. Durch die Kombination von Methoden zur Tiefenmerkmalsextraktion und Tiefenanpassung stellen wir hier ein vielseitiges hybrides visuelles SLAM-System vor, das die Anpassung in anspruchsvollen Szenarien wie schlechten Lichtverhältnissen, dynamischer Beleuchtung, schwach strukturierten Bereichen und starkem Jitter verbessern soll. Unser System unterstützt mehrere Modi, einschließlich erweiterter Monokular-, Stereo-, Monokular-Trägheits- und Stereo-Trägheitskonfigurationen. Darüber hinaus wird analysiert, wie visuelles SLAM mit Deep-Learning-Methoden kombiniert werden kann, um andere Forschungen zu inspirieren. Durch umfangreiche Experimente mit öffentlichen Datensätzen und selbst abgetasteten Daten demonstrieren wir die Überlegenheit von SL-SLAM in Bezug auf Positionierungsgenauigkeit und Tracking-Robustheit.
 Hallo, elektrischer Atlas! Der Boston Dynamics-Roboter erwacht wieder zum Leben, seltsame 180-Grad-Bewegungen machen Musk Angst
Apr 18, 2024 pm 07:58 PM
Hallo, elektrischer Atlas! Der Boston Dynamics-Roboter erwacht wieder zum Leben, seltsame 180-Grad-Bewegungen machen Musk Angst
Apr 18, 2024 pm 07:58 PM
Boston Dynamics Atlas tritt offiziell in die Ära der Elektroroboter ein! Gestern hat sich der hydraulische Atlas einfach „unter Tränen“ von der Bühne der Geschichte zurückgezogen. Heute gab Boston Dynamics bekannt, dass der elektrische Atlas im Einsatz ist. Es scheint, dass Boston Dynamics im Bereich kommerzieller humanoider Roboter entschlossen ist, mit Tesla zu konkurrieren. Nach der Veröffentlichung des neuen Videos wurde es innerhalb von nur zehn Stunden bereits von mehr als einer Million Menschen angesehen. Die alten Leute gehen und neue Rollen entstehen. Das ist eine historische Notwendigkeit. Es besteht kein Zweifel, dass dieses Jahr das explosive Jahr der humanoiden Roboter ist. Netizens kommentierten: Die Weiterentwicklung der Roboter hat dazu geführt, dass die diesjährige Eröffnungsfeier wie Menschen aussieht, und der Freiheitsgrad ist weitaus größer als der von Menschen. Aber ist das wirklich kein Horrorfilm? Zu Beginn des Videos liegt Atlas ruhig auf dem Boden, scheinbar auf dem Rücken. Was folgt, ist atemberaubend
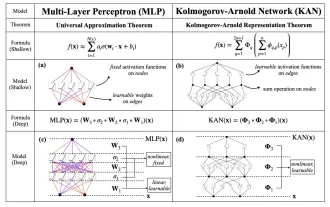 KAN, das MLP ersetzt, wurde durch Open-Source-Projekte auf Faltung erweitert
Jun 01, 2024 pm 10:03 PM
KAN, das MLP ersetzt, wurde durch Open-Source-Projekte auf Faltung erweitert
Jun 01, 2024 pm 10:03 PM
Anfang dieses Monats schlugen Forscher des MIT und anderer Institutionen eine vielversprechende Alternative zu MLP vor – KAN. KAN übertrifft MLP in Bezug auf Genauigkeit und Interpretierbarkeit. Und es kann MLP, das mit einer größeren Anzahl von Parametern ausgeführt wird, mit einer sehr kleinen Anzahl von Parametern übertreffen. Beispielsweise gaben die Autoren an, dass sie KAN nutzten, um die Ergebnisse von DeepMind mit einem kleineren Netzwerk und einem höheren Automatisierungsgrad zu reproduzieren. Konkret verfügt DeepMinds MLP über etwa 300.000 Parameter, während KAN nur etwa 200 Parameter hat. KAN hat eine starke mathematische Grundlage wie MLP und basiert auf dem universellen Approximationssatz, während KAN auf dem Kolmogorov-Arnold-Darstellungssatz basiert. Wie in der folgenden Abbildung gezeigt, hat KAN
 Google ist begeistert: JAX-Leistung übertrifft Pytorch und TensorFlow! Es könnte die schnellste Wahl für das GPU-Inferenztraining werden
Apr 01, 2024 pm 07:46 PM
Google ist begeistert: JAX-Leistung übertrifft Pytorch und TensorFlow! Es könnte die schnellste Wahl für das GPU-Inferenztraining werden
Apr 01, 2024 pm 07:46 PM
Die von Google geförderte Leistung von JAX hat in jüngsten Benchmark-Tests die von Pytorch und TensorFlow übertroffen und belegt bei 7 Indikatoren den ersten Platz. Und der Test wurde nicht auf der TPU mit der besten JAX-Leistung durchgeführt. Obwohl unter Entwicklern Pytorch immer noch beliebter ist als Tensorflow. Aber in Zukunft werden möglicherweise mehr große Modelle auf Basis der JAX-Plattform trainiert und ausgeführt. Modelle Kürzlich hat das Keras-Team drei Backends (TensorFlow, JAX, PyTorch) mit der nativen PyTorch-Implementierung und Keras2 mit TensorFlow verglichen. Zunächst wählen sie eine Reihe von Mainstream-Inhalten aus
 Tesla-Roboter arbeiten in Fabriken, Musk: Der Freiheitsgrad der Hände wird dieses Jahr 22 erreichen!
May 06, 2024 pm 04:13 PM
Tesla-Roboter arbeiten in Fabriken, Musk: Der Freiheitsgrad der Hände wird dieses Jahr 22 erreichen!
May 06, 2024 pm 04:13 PM
Das neueste Video von Teslas Roboter Optimus ist veröffentlicht und er kann bereits in der Fabrik arbeiten. Bei normaler Geschwindigkeit sortiert es Batterien (Teslas 4680-Batterien) so: Der Beamte hat auch veröffentlicht, wie es bei 20-facher Geschwindigkeit aussieht – auf einer kleinen „Workstation“, pflücken und pflücken und pflücken: Dieses Mal wird es freigegeben. Eines der Highlights Der Vorteil des Videos besteht darin, dass Optimus diese Arbeit in der Fabrik völlig autonom und ohne menschliches Eingreifen während des gesamten Prozesses erledigt. Und aus Sicht von Optimus kann es auch die krumme Batterie aufnehmen und platzieren, wobei der Schwerpunkt auf der automatischen Fehlerkorrektur liegt: In Bezug auf die Hand von Optimus gab der NVIDIA-Wissenschaftler Jim Fan eine hohe Bewertung ab: Die Hand von Optimus ist der fünffingrige Roboter der Welt am geschicktesten. Seine Hände sind nicht nur taktil
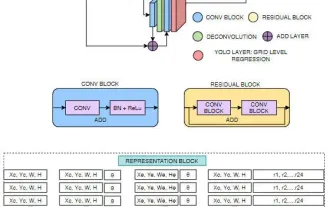 FisheyeDetNet: der erste Zielerkennungsalgorithmus basierend auf einer Fischaugenkamera
Apr 26, 2024 am 11:37 AM
FisheyeDetNet: der erste Zielerkennungsalgorithmus basierend auf einer Fischaugenkamera
Apr 26, 2024 am 11:37 AM
Die Zielerkennung ist ein relativ ausgereiftes Problem in autonomen Fahrsystemen, wobei die Fußgängererkennung einer der ersten Algorithmen ist, die eingesetzt werden. In den meisten Arbeiten wurde eine sehr umfassende Recherche durchgeführt. Die Entfernungswahrnehmung mithilfe von Fischaugenkameras für die Rundumsicht ist jedoch relativ wenig untersucht. Aufgrund der großen radialen Verzerrung ist es schwierig, die standardmäßige Bounding-Box-Darstellung in Fischaugenkameras zu implementieren. Um die obige Beschreibung zu vereinfachen, untersuchen wir erweiterte Begrenzungsrahmen-, Ellipsen- und allgemeine Polygondesigns in Polar-/Winkeldarstellungen und definieren eine mIOU-Metrik für die Instanzsegmentierung, um diese Darstellungen zu analysieren. Das vorgeschlagene Modell „fisheyeDetNet“ mit polygonaler Form übertrifft andere Modelle und erreicht gleichzeitig 49,5 % mAP auf dem Valeo-Fisheye-Kameradatensatz für autonomes Fahren



