 Technologie-Peripheriegeräte
Technologie-Peripheriegeräte
 KI
KI
 Ein chinesisches Team hat erfolgreich KI entwickelt, um geeignete Medikamente für Krebspatienten vorherzusagen, und die Ergebnisse wurden in der Unterzeitschrift „Nature' veröffentlicht
Ein chinesisches Team hat erfolgreich KI entwickelt, um geeignete Medikamente für Krebspatienten vorherzusagen, und die Ergebnisse wurden in der Unterzeitschrift „Nature' veröffentlicht
Ein chinesisches Team hat erfolgreich KI entwickelt, um geeignete Medikamente für Krebspatienten vorherzusagen, und die Ergebnisse wurden in der Unterzeitschrift „Nature' veröffentlicht
Mit nur einer KI können die klinischen Reaktionen von 9.808 Krebspatienten auf Medikamente vollständig vorhergesagt werden.
Und die Ergebnisse stimmen mit klinischen Beobachtungen überein.
Dies ist das neueste Ergebnis des CODE-AE (kontextbewusster Dekonfundierungs-Autoencoder) von Lei Xies Team an der City University of New York.

Es schlägt ein neuartiges kontextbezogenes Autokodierungsmodell vor, das die spezifischen Reaktionen verschiedener Patienten auf Medikamente vorhersagen kann.
Dies wird erhebliche Auswirkungen auf die Entwicklung neuer Medikamente und klinische Studien haben.
Wissen Sie, nach dem traditionellen Modell dauert es fast 10 Jahre, ein neues Medikament zu entwickeln, zu testen und vollständig zu vermarkten, und die verbrauchten Mittel sind beispiellos riesig und erreichen leicht eine Milliarde US-Dollar.
Der Zyklus ist so lang, weil die Reaktion neuer Medikamente im menschlichen Körper schwer vorherzusagen ist und oft wiederholte Versuche zum Testen erforderlich sind.
Wenn KI mithilfe von Daten Vorhersagen treffen kann, wird dies die Zeit bis zur Markteinführung neuer Medikamente erheblich verkürzen und die Kosten senken.
Derzeit wurde diese Forschung in der Nature-Unterzeitschrift „Nature Machine Intelligence“ veröffentlicht.
Einfach ausgedrückt: CODE-AE verwendet Daten aus der In-vitro-Zellvalidierung neuer Arzneimittel, um die Reaktion des Arzneimittels im menschlichen Körper vorherzusagen .
Dadurch wird die Abhängigkeit des KI-Modelltrainings von klinischen Patientendaten vermieden.
Der Hauptgrund, warum KI in der Vergangenheit bei der Vorhersage des klinischen Ansprechens nicht sehr effektiv war, ist, dass es zu schwierig ist, umfangreiche und kontinuierliche Daten zum klinischen Ansprechen zu sammeln.
Aus Mechanismussicht unterteilen Forscher Arzneimittelbiomarker in Quelldomäne und Zieldomäne.
Die Quelldomäne stellt eine andere Domäne als die Testprobe dar, verfügt jedoch über umfangreiche Überwachungsinformationen, die als In-vitro-Zellverifizierungsdaten verstanden werden können.
Die Zieldomäne ist die Domäne, in der sich die Testprobe befindet. Sie hat keine oder nur wenige Etiketten, also Patientendaten.
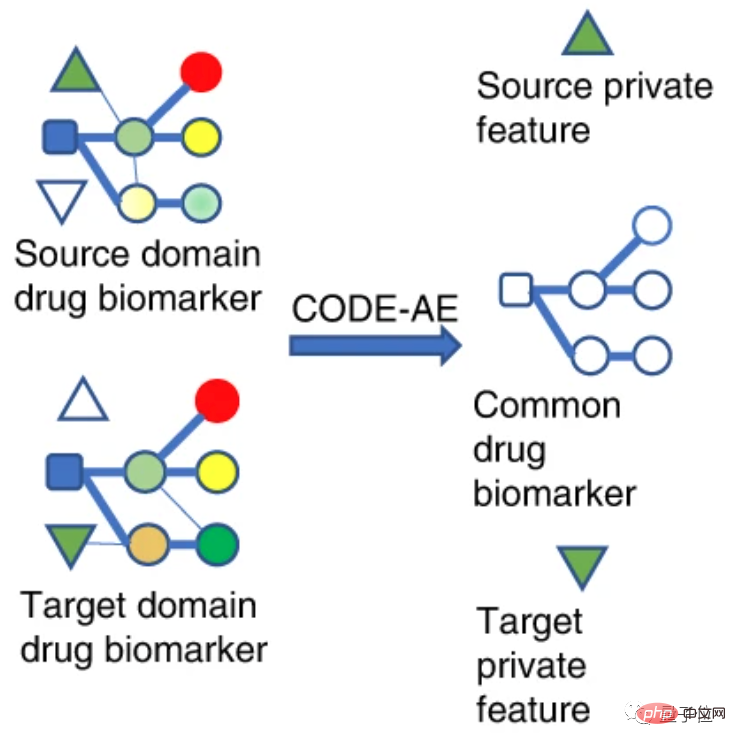
Ordnen Sie Datenmerkmale aus verschiedenen Feldern demselben Merkmalsraum zu, sodass ihre Abstände in diesem Raum so gering wie möglich sind.
So kann die auf der Quelldomäne im Merkmalsraum trainierte Zielfunktion auf die Zieldomäne übertragen werden, um die Genauigkeit in der Zieldomäne zu verbessern.
Im Kontext dieser Forschung sind sowohl die Quelldomäne als auch die Zieldomäne Datenmerkmale von Arzneimittelbiomarkern, also Datenmerkmale von Arzneimittelzielen.
Konkret ist das Modellgerüst in drei Teile unterteilt: Vortraining, Feinabstimmung und Inferenz.
Das Vortraining nutzt hauptsächlich selbstüberwachtes Lernen, um ein Feature-Codierungsmodul zu erstellen, um die unbeschrifteten Genexpressionsprofile von In-vitro-Zelldaten und Patientendaten im Einbettungsraum abzubilden. Auf diese Weise können einige Störfaktoren eliminiert werden und die latente Verteilung der beiden Daten kann konsistent sein, um systematische Verzerrungen zu beseitigen.
Die Feinabstimmungsphase besteht darin, ein überwachtes Modell auf der Grundlage des Vortrainings hinzuzufügen und markierte In-vitro-Zelldaten für das Training zu verwenden.
Abschließend werden in der Inferenzphase die aus dem Vortraining gewonnenen Patienten zunächst disambiguiert und eingebettet, und dann wird das abgestimmte Modell verwendet, um die Reaktion des Patienten auf das Medikament vorherzusagen.

In diesem Modus weist CODE-AE zwei Eigenschaften auf.
Erstens kann es allgemeine biologische Signale und private Darstellungen in inkohärenten Proben extrahieren und so Interferenzen durch unterschiedliche Datenmuster beseitigen.
Zweitens kann nach der Trennung des Arzneimittelreaktionssignals und der Störfaktoren auch eine lokale Ausrichtung erreicht werden.
Zusammenfassend kann CODE-AE als der Prozess der Auswahl einzigartiger Merkmale im inkohärenten Datenmuster-Einbettungsraum von beschrifteten und unbeschrifteten Daten verstanden werden.
Um die Wirksamkeit des Modells zu demonstrieren, sagten die Forscher die Medikamenteneignung von 9.808 Krebspatienten voraus.
Wenn die vom Modell vorhergesagten Standortergebnisse für den Zustand des Patienten mit dem von ihm verwendeten Medikamentenziel in Zusammenhang stehen, beweist dies, dass die Vorhersage korrekt ist.
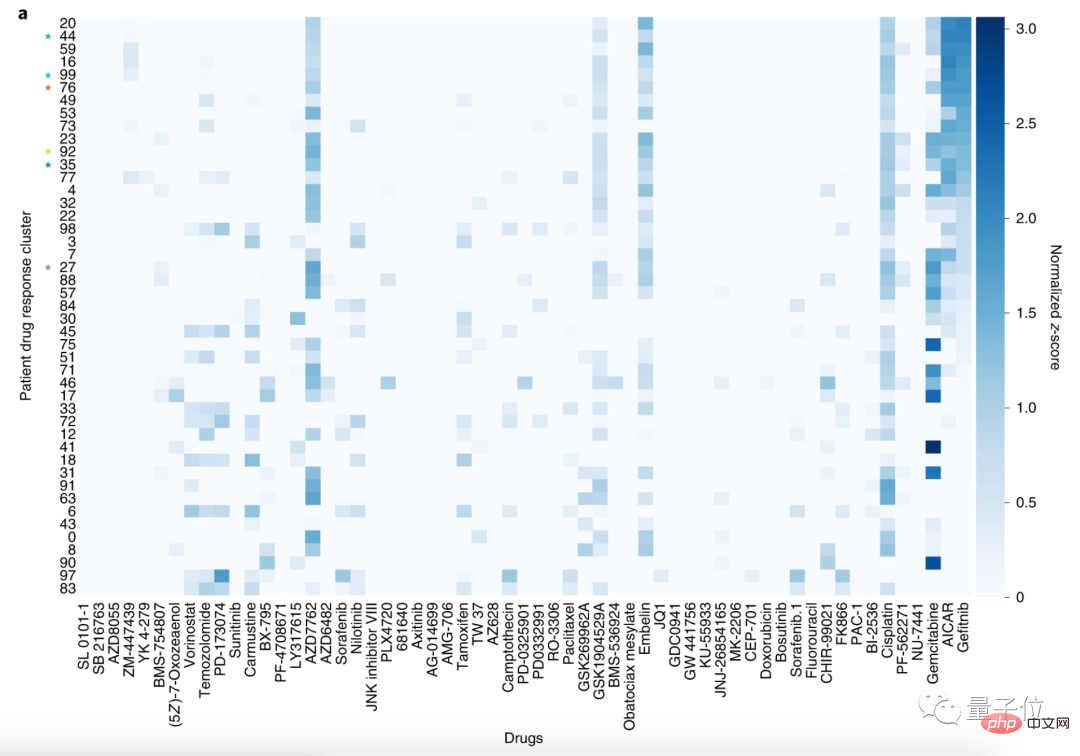
Die Forscher teilten dann die Patienten in 100 Cluster und die 59 Medikamente in 30 Cluster ein.
Durch diese Analysemethode können Patienten mit ähnlichen Medikamentenreaktionsprofilen in Gruppen zusammengefasst werden.
Hier nehmen wir die Häufung von Patienten mit Plattenepithelkarzinomen der Lunge (LSCC) und Patienten mit nicht-kleinzelligem Lungenkrebs (NSCLC) als Beispiel.
Unter den 59 Medikamenten sind Gefitinib, AICAR und Gemcitabin die empfindlichsten Medikamente für LSCC.
Das Ziel von Gefitinib und AICAR ist unter anderem ein epidermaler Wachstumsfaktorrezeptor (EGFR), und Gemcitabin wird häufig zur Behandlung von nichtkleinzelligem Lungenkrebs ohne EGFR-Mutationen eingesetzt.
In der Arbeit wurde festgestellt, dass CODE-AE im Einklang mit der Wirkungsweise dieser Arzneimittel feststellte, dass Patienten, die Gefitinib und AICAR verwendeten, ähnliche Arzneimittelreaktionsprofile aufwiesen.
Mit anderen Worten: CODE-AE hat das richtige Ziel für die Patientenbehandlung entdeckt, das heißt, es kann anwendbare Medikamente vorhersagen.
Das oben genannte Forschungsteam stammt von der City University of New York.
Der korrespondierende Autor ist Lei Xie, die einen Bachelor-Abschluss in Polymerphysik an der Universität für Wissenschaft und Technologie in China erworben hat.
Abschluss mit einem Master-Abschluss in Informatik an der Rutgers University, aber mit einem Doktortitel in Chemie.

Es versteht sich, dass der nächste Schritt des Forschungsteams darin bestehen wird, die Vorhersagefunktion von CODE-AE in Bezug auf Konzentration und Metabolismus der klinischen Reaktion neuer Medikamente zu entwickeln.
Die Forscher sagten, dass das KI-Modell auch angepasst werden könnte, um die Nebenwirkungen von Medikamenten auf den menschlichen Körper vorherzusagen.
Erwähnenswert ist, dass sich die Unterzeitschrift „Nature Machine Intelligence“ von Nature speziell auf die interdisziplinäre angewandte Forschung in den Bereichen künstliche Intelligenz und Biowissenschaften konzentriert und jedes Jahr durchschnittlich etwa 60 Beiträge enthält.
Papieradresse: https://www.nature.com/articles/s42256-022-00541-0
Referenzlink: https://phys.org/news/2022-10-ai-accurately-human-response -drug.html
Das obige ist der detaillierte Inhalt vonEin chinesisches Team hat erfolgreich KI entwickelt, um geeignete Medikamente für Krebspatienten vorherzusagen, und die Ergebnisse wurden in der Unterzeitschrift „Nature' veröffentlicht. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!

Heiße KI -Werkzeuge

Undresser.AI Undress
KI-gestützte App zum Erstellen realistischer Aktfotos

AI Clothes Remover
Online-KI-Tool zum Entfernen von Kleidung aus Fotos.

Undress AI Tool
Ausziehbilder kostenlos

Clothoff.io
KI-Kleiderentferner

Video Face Swap
Tauschen Sie Gesichter in jedem Video mühelos mit unserem völlig kostenlosen KI-Gesichtstausch-Tool aus!

Heißer Artikel

Heiße Werkzeuge

Notepad++7.3.1
Einfach zu bedienender und kostenloser Code-Editor

SublimeText3 chinesische Version
Chinesische Version, sehr einfach zu bedienen

Senden Sie Studio 13.0.1
Leistungsstarke integrierte PHP-Entwicklungsumgebung

Dreamweaver CS6
Visuelle Webentwicklungstools

SublimeText3 Mac-Version
Codebearbeitungssoftware auf Gottesniveau (SublimeText3)

Heiße Themen
 Worldcoin (WLD) Preisprognose 2025-2031: Wird WLD bis 2031 $ erreichen?
Apr 21, 2025 pm 02:42 PM
Worldcoin (WLD) Preisprognose 2025-2031: Wird WLD bis 2031 $ erreichen?
Apr 21, 2025 pm 02:42 PM
Worldcoin (WLD) fällt auf dem Kryptowährungsmarkt mit seinen einzigartigen biometrischen Überprüfungs- und Datenschutzschutzmechanismen auf, die die Aufmerksamkeit vieler Investoren auf sich ziehen. WLD hat mit seinen innovativen Technologien, insbesondere in Kombination mit OpenAI -Technologie für künstliche Intelligenz, außerdem unter Altcoins gespielt. Aber wie werden sich die digitalen Vermögenswerte in den nächsten Jahren verhalten? Lassen Sie uns den zukünftigen Preis von WLD zusammen vorhersagen. Die Preisprognose von 2025 WLD wird voraussichtlich im Jahr 2025 ein signifikantes Wachstum in WLD erzielen. Die Marktanalyse zeigt, dass der durchschnittliche WLD -Preis 1,31 USD mit maximal 1,36 USD erreichen kann. In einem Bärenmarkt kann der Preis jedoch auf rund 0,55 US -Dollar fallen. Diese Wachstumserwartung ist hauptsächlich auf Worldcoin2 zurückzuführen.
 Wie lautet das Analysetabieren der Bitcoin -Struktur mit der Fertigprodukt? Wie zeichne ich?
Apr 21, 2025 pm 07:42 PM
Wie lautet das Analysetabieren der Bitcoin -Struktur mit der Fertigprodukt? Wie zeichne ich?
Apr 21, 2025 pm 07:42 PM
Zu den Schritten zum Zeichnen eines Bitcoin -Strukturanalyse -Diagramms gehören: 1.. Bestimmen Sie den Zweck und die Zielgruppe der Zeichnung, 2. Wählen Sie das richtige Werkzeug aus, 3. Entwerfen Sie das Framework und füllen Sie die Kernkomponenten aus, 4. Siehe vorhandene Vorlage. Vollständige Schritte stellen sicher, dass das Diagramm genau und leicht zu verstehen ist.
 Was bedeutet Cross-Chain-Transaktion? Was sind die Cross-Chain-Transaktionen?
Apr 21, 2025 pm 11:39 PM
Was bedeutet Cross-Chain-Transaktion? Was sind die Cross-Chain-Transaktionen?
Apr 21, 2025 pm 11:39 PM
Börsen, die Cross-Chain-Transaktionen unterstützen: 1. Binance, 2. Uniswap, 3. Sushiswap, 4. Kurvenfinanzierung, 5. Thorchain, 6. 1inch Exchange, 7. DLN-Handel, diese Plattformen unterstützen Multi-Chain-Asset-Transaktionen durch verschiedene Technologien.
 Die zehn Top-Empfehlungen zur Plattform für Echtzeitdaten auf den Währungskreismärkten werden veröffentlicht
Apr 22, 2025 am 08:12 AM
Die zehn Top-Empfehlungen zur Plattform für Echtzeitdaten auf den Währungskreismärkten werden veröffentlicht
Apr 22, 2025 am 08:12 AM
Zu den für Anfängern geeigneten Kryptowährungsdatenplattformen gehören CoinMarketCap und nicht-kleine Trompete. 1. CoinmarketCap bietet globale Rangliste für den Preis, den Marktwert und der Handelsvolumen für Anfänger für Anfänger und Grundanalyse. 2. Das nichtklammernde Angebot bietet eine chinesisch-freundliche Schnittstelle, die chinesischen Benutzern geeignet ist, um potenzielle Projekte mit geringem Risiko schnell zu untersuchen.
 Aavenomics ist eine Empfehlung, das Aave -Protokoll -Token zu ändern und Token -Rückkauf einzuführen, die die Quorum -Anzahl von Personen erreicht hat.
Apr 21, 2025 pm 06:24 PM
Aavenomics ist eine Empfehlung, das Aave -Protokoll -Token zu ändern und Token -Rückkauf einzuführen, die die Quorum -Anzahl von Personen erreicht hat.
Apr 21, 2025 pm 06:24 PM
Aavenomics ist ein Vorschlag zur Änderung des Aave -Protokoll -Tokens und zur Einführung von Token -Repos, die ein Quorum für Aavedao implementiert hat. Marc Zeller, Gründer der AAVE -Projektkette (ACI), kündigte dies auf X an und stellte fest, dass sie eine neue Ära für die Vereinbarung markiert. Marc Zeller, Gründer der Aave Chain Initiative (ACI), kündigte auf X an, dass der Aavenomics -Vorschlag das Modifizieren des Aave -Protokoll -Tokens und die Einführung von Token -Repos umfasst, hat ein Quorum für Aavedao erreicht. Laut Zeller ist dies eine neue Ära für die Vereinbarung. AVEDAO -Mitglieder stimmten überwiegend für die Unterstützung des Vorschlags, der am Mittwoch 100 pro Woche betrug
 Rexas Finance (RXS) kann Solana (SOL), Cardano (ADA), XRP und Dogecoin (Doge) im Jahr 2025 übertreffen
Apr 21, 2025 pm 02:30 PM
Rexas Finance (RXS) kann Solana (SOL), Cardano (ADA), XRP und Dogecoin (Doge) im Jahr 2025 übertreffen
Apr 21, 2025 pm 02:30 PM
Auf dem volatilen Kryptowährungsmarkt suchen Anleger nach Alternativen, die über die beliebten Währungen hinausgehen. Obwohl bekannte Kryptowährungen wie Solana (SOL), Cardano (ADA), XRP und Doge (DOGE) auch Herausforderungen wie Marktgefühle, regulatorische Unsicherheit und Skalierbarkeit gegenübersehen. Ein neues aufstrebendes Projekt, Rexasfinance (RXS), entsteht jedoch. Es stützt sich nicht auf Prominenteffekte oder Hype, sondern konzentriert sich auf die Kombination der realen Vermögenswerte (RWA) mit Blockchain-Technologie, um den Anlegern eine innovative Möglichkeit zum Investieren zu bieten. Diese Strategie hofft, eines der erfolgreichsten Projekte von 2025 zu sein. Rexasfi
 Rangliste der Hebelbörsen im Währungskreis Die neuesten Empfehlungen der zehn meistgezogenen Börsen im Währungskreis
Apr 21, 2025 pm 11:24 PM
Rangliste der Hebelbörsen im Währungskreis Die neuesten Empfehlungen der zehn meistgezogenen Börsen im Währungskreis
Apr 21, 2025 pm 11:24 PM
Die Plattformen, die im Jahr 2025 im Leveraged Trading, Security und Benutzererfahrung hervorragende Leistung haben, sind: 1. OKX, geeignet für Hochfrequenzhändler und bieten bis zu 100-fache Hebelwirkung; 2. Binance, geeignet für Mehrwährungshändler auf der ganzen Welt und bietet 125-mal hohe Hebelwirkung; 3. Gate.io, geeignet für professionelle Derivate Spieler, die 100 -fache Hebelwirkung bietet; 4. Bitget, geeignet für Anfänger und Sozialhändler, die bis zu 100 -fache Hebelwirkung bieten; 5. Kraken, geeignet für stetige Anleger, die fünfmal Hebelwirkung liefert; 6. Bybit, geeignet für Altcoin -Entdecker, die 20 -fache Hebelwirkung bietet; 7. Kucoin, geeignet für kostengünstige Händler, die 10-fache Hebelwirkung bietet; 8. Bitfinex, geeignet für das Seniorenspiel
 Was sind die Hybrid -Blockchain -Handelsplattformen?
Apr 21, 2025 pm 11:36 PM
Was sind die Hybrid -Blockchain -Handelsplattformen?
Apr 21, 2025 pm 11:36 PM
Vorschläge für die Auswahl eines Kryptowährungsaustauschs: 1. Für die Liquiditätsanforderungen ist Priorität Binance, Gate.io oder OKX aufgrund seiner Bestelltiefe und der starken Volatilitätsbeständigkeit. 2. Compliance and Security, Coinbase, Kraken und Gemini haben strenge regulatorische Bestätigung. 3. Innovative Funktionen, Kucoins sanftes Stakel und Derivatdesign von Bitbit eignen sich für fortschrittliche Benutzer.





