 Technologie-Peripheriegeräte
Technologie-Peripheriegeräte
 KI
KI
 Vollständiges Codebeispiel für Deep Learning für medizinische Bilder: Segmentierung von Bildern aus MRT-Gehirnscans mit Pytorch
Vollständiges Codebeispiel für Deep Learning für medizinische Bilder: Segmentierung von Bildern aus MRT-Gehirnscans mit Pytorch
Vollständiges Codebeispiel für Deep Learning für medizinische Bilder: Segmentierung von Bildern aus MRT-Gehirnscans mit Pytorch
Bildsegmentierung ist eine der wichtigsten Aufgaben in der medizinischen Bildanalyse und oft der erste und kritischste Schritt in vielen klinischen Anwendungen. Bei der MRT-Analyse des Gehirns wird die Bildsegmentierung üblicherweise zur Messung und Visualisierung anatomischer Strukturen, zur Analyse von Gehirnveränderungen, zur Abgrenzung pathologischer Bereiche sowie zur chirurgischen Planung und bildgesteuerten Eingriffen verwendet. Die Segmentierung ist eine Voraussetzung für die meisten morphologischen Analysen.
In diesem Artikel stellen wir vor, wie man mit QuickNAT Bilder des menschlichen Gehirns segmentiert. Verwenden Sie MONAI, PyTorch und gängige Python-Bibliotheken für die Datenvisualisierung und Berechnungen wie NumPy, TorchIO und Matplotlib.
In diesem Artikel geht es hauptsächlich um die folgenden Aspekte:
- Einrichten des Datensatzes und Erkunden der Daten
- Verarbeiten und Vorbereiten des Datensatzes Richtiges Modelltraining
- Erstellen einer Trainingsschleife
- Bewerten des Modells und Analysieren der Ergebnisse
Der vollständige Code wird am Ende dieses Artikels bereitgestellt.
Festlegen des Datenverzeichnisses
Der erste Schritt zur Verwendung von MONAI besteht darin, die Umgebungsvariable MONAI_DATA_DIRECTORY festzulegen, um das Verzeichnis anzugeben. Wenn nicht angegeben, wird ein temporäres Verzeichnis verwendet.
<code>directory = os.environ.get("MONAI_DATA_DIRECTORY") root_dir = tempfile.mkdtemp() if directory is None else directory print(root_dir)</code>Datensatz einrichten
Eine der größten Herausforderungen bei der Skalierung von CNN-Modellen zur Gehirnsegmentierung ist die begrenzte Verfügbarkeit von von Menschen kommentierten Trainingsdaten. Die Autoren stellen eine neue Trainingsstrategie vor, die große Datensätze ohne manuelle Beschriftungen und kleine Datensätze mit manuellen Beschriftungen nutzt.
Verwenden Sie zunächst vorhandene Softwaretools (z. B. FreeSurfer), um automatisch generierte Segmentierungen aus großen, unbeschrifteten Datensätzen zu erhalten, und verwenden Sie diese Tools dann, um das Netzwerk vorab zu trainieren. Im zweiten Schritt erfolgt die Feinabstimmung des Netzwerks anhand kleinerer manuell annotierter Daten [2].
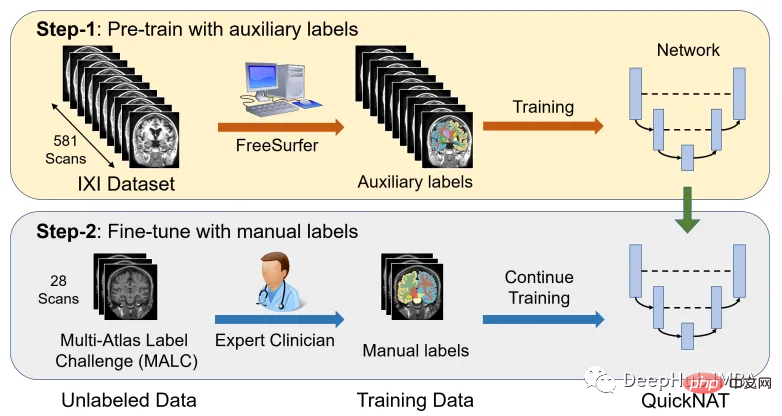
Der IXI-Datensatz besteht aus 581 unbeschrifteten MRT-T1-Scans gesunder Probanden. Die Daten wurden in drei verschiedenen Krankenhäusern in London gesammelt. Der Hauptnachteil bei der Verwendung dieses Datensatzes besteht darin, dass die Beschriftungen nicht öffentlich verfügbar sind. Um den gleichen Ansatz wie im Forschungsbericht zu verfolgen, wird in diesem Artikel FreeSurfer verwendet, um Segmentierungen für diese MRT-T1-Scans zu generieren.
FreeSurfer ist ein Softwarepaket zur Analyse und Visualisierung von Strukturen. Download- und Installationsanweisungen finden Sie hier. Alle kortikalen Rekonstruktionsprozesse können direkt mit dem Befehl „recon-all“ durchgeführt werden.
Obwohl FreeSurfer ein sehr nützliches Tool ist, um große Mengen unbeschrifteter Daten zu nutzen und das Netzwerk überwacht zu trainieren, dauert das Scannen zum Generieren dieser Etiketten bis zu 5 Stunden, daher verwenden wir hier direkt den OASIS-Datensatz, um das zu trainieren Modell ist der OASIS-Datensatz ein kleinerer Datensatz mit öffentlich verfügbaren manuellen Anmerkungen.
OASIS ist ein Projekt, das darauf abzielt, der wissenschaftlichen Gemeinschaft Datensätze zur Bildgebung des Gehirns frei zugänglich zu machen. OASIS-1 ist ein Datensatz, der aus Querschnitten von 39 Probanden besteht und wie folgt erhalten wurde:
<code>resource = "https://download.nrg.wustl.edu/data/oasis_cross-sectional_disc1.tar.gz" md5 = "c83e216ef8654a7cc9e2a30a4cdbe0cc" compressed_file = os.path.join(root_dir, "oasis_cross-sectional_disc1.tar.gz") data_dir = os.path.join(root_dir, "Oasis_Data") if not os.path.exists(data_dir): download_and_extract(resource, compressed_file, data_dir, md5)</code>
Datenexploration
Wenn Sie „oasis_crosssection_disc1.tar.gz“ öffnen, werden Sie feststellen, dass jeder Proband einen anderen Ordner hat. Für das Thema OAS1_0001_MR1 sieht es beispielsweise so aus:
Pfad der Spiegeldatendatei: disc1OAS1_0001_MR1PROCESSEDMPRAGET88_111 oas1_0001_mr1_mpr_n4_anon_111_t88_masked_ggc .img
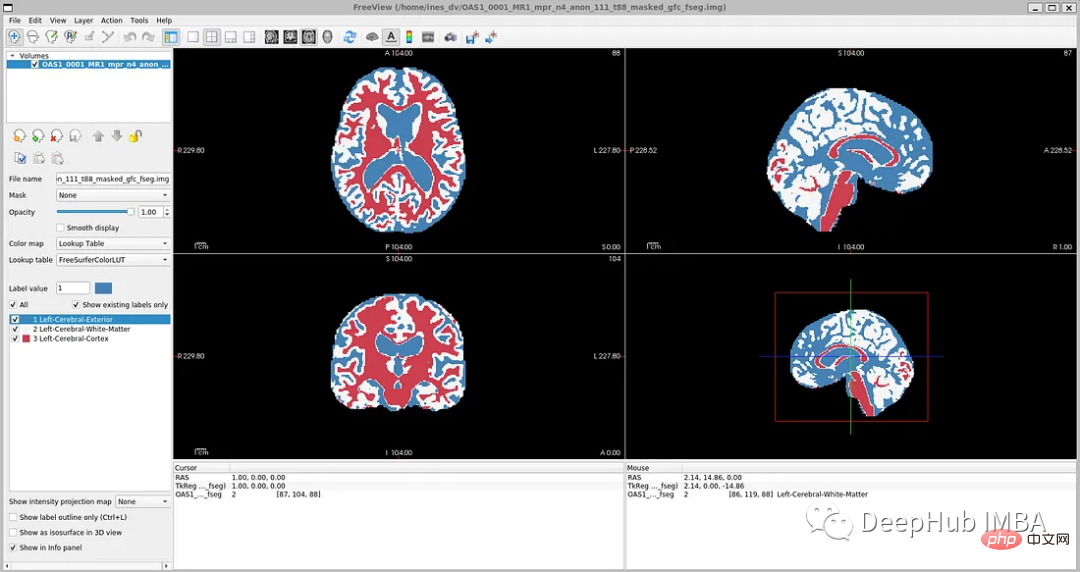
Tag-Datei: disc1OAS1_0001_MR1FSL_SEGOAS1_0001_MR1_mpr_n4_anon_111_t88_masked_gfc_fseg.img
Laden und Vorverarbeiten der Daten
Laden Sie den Datensatz herunter und nachdem es in ein temporäres Verzeichnis extrahiert wurde, muss es neu strukturiert werden. Unser Verzeichnis soll so aussehen:
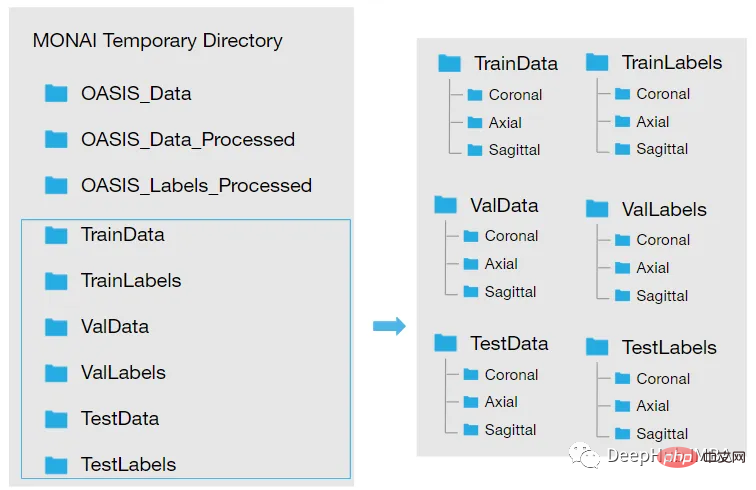
Wir müssen also die folgenden Schritte ausführen, um die Daten zu laden:
wird. IMG-Dateien werden in . konvertiert. nii-Datei und speichern Sie sie in einem neuen Ordner: Erstellen Sie zwei neue Ordner. Oasis_Data_Processed enthält die verarbeiteten MRT-T1-Scans für jedes Subjekt und Oasis_Labels_Processed enthält die entsprechenden Beschriftungen.
<code>new_path_data= root_dir + '/Oasis_Data_Processed/' if not os.path.exists(new_path_data): os.makedirs(new_path_data) new_path_labels= root_dir + '/Oasis_Labels_Processed/' if not os.path.exists(new_path_labels): os.makedirs(new_path_labels)</code>
Dann bedienen Sie es einfach:
<code>for i in [x for x in range(1, 43) if x != 8 and x != 24 and x != 36]: if i </code>
Der spezifische Code wird nicht mehr eingefügt. Wenn Sie interessiert sind, schauen Sie sich den endgültigen vollständigen Code an. Der nächste Schritt besteht darin, die Bild- und Beschriftungsdateinamen zu lesen
<code>image_files = sorted(glob(os.path.join(root_dir + '/Oasis_Data_Processed', '*.nii'))) label_files = sorted(glob(os.path.join(root_dir + '/Oasis_Labels_Processed', '*.nii'))) files = [{'image': image_name, 'label': label_name} for image_name, label_name in zip(image_files, label_files)]</code>Um Bilder mit entsprechenden Beschriftungen zu visualisieren, können Sie TorchIO verwenden, eine Python-Bibliothek zum Laden, Vorverarbeiten, Verbessern und Abtasten mehrdimensionaler medizinischer Bilder für Deep Learning.
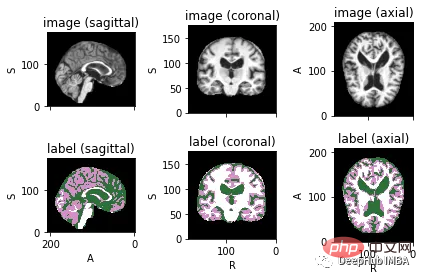
<code>image_filename = root_dir + '/Oasis_Data_Processed/OAS1_0001_MR1_mpr_n4_anon_111_t88_masked_gfc.nii' label_filename = root_dir + '/Oasis_Labels_Processed/OAS1_0001_MR1_mpr_n4_anon_111_t88_masked_gfc_fseg.nii' subject = torchio.Subject(image=torchio.ScalarImage(image_filename), label=torchio.LabelMap(label_filename)) subject.plot()</code>
下面就是将数据分成3部分——训练、验证和测试。将数据分成三个不同的类别的目的是建立一个可靠的机器学习模型,避免过拟合。
我们将整个数据集分成三个部分:
Train: 80%,Validation: 10%,Test: 10%
<code>train_inds, val_inds, test_inds = partition_dataset(data = np.arange(len(files)), ratios = [8, 1, 1], shuffle = True) train = [files[i] for i in sorted(train_inds)] val = [files[i] for i in sorted(val_inds)] test = [files[i] for i in sorted(test_inds)] print(f"Training count: {len(train)}, Validation count: {len(val)}, Test count: {len(test)}")</code>因为模型需要的是二维切片,所以将每个切片保存在不同的文件夹中,如下图所示。这两个代码单元将训练集的每个MRI体积的切片保存为“.png”格式。
<code>Save coronal slices for training images dir = root_dir + '/TrainData' os.makedirs(os.path.join(dir, "Coronal")) path = root_dir + '/TrainData/Coronal/' for file in sorted(glob(os.path.join(root_dir + '/TrainData', '*.nii'))): image=torchio.ScalarImage(file) data = image.data filename = os.path.basename(file) filename = os.path.splitext(filename) for i in range(0, 208): slice = data[0, :, i] array = slice.numpy() data_dir = root_dir + '/TrainData/Coronal/' + filename[0] + '_slice' + str(i) + '.png' plt.imsave(fname = data_dir, arr = array, format = 'png', cmap = plt.cm.gray)</code>
同理,下面是保存标签:
<code>dir = root_dir + '/TrainLabels' os.makedirs(os.path.join(dir, "Coronal")) path = root_dir + '/TrainLabels/Coronal/' for file in sorted(glob(os.path.join(root_dir + '/TrainLabels', '*.nii'))): label = torchio.LabelMap(file) data = label.data filename = os.path.basename(file) filename = os.path.splitext(filename) for i in range(0, 208): slice = data[0, :, i] array = slice.numpy() data_dir = root_dir + '/TrainLabels/Coronal/' + filename[0] + '_slice' + str(i) + '.png' plt.imsave(fname = data_dir, arr = array, format = 'png')</code>
为训练和验证定义图像的变换处理
在本例中,我们将使用Dictionary Transforms,其中数据是Python字典。
<code>train_images_coronal = [] for file in sorted(glob(os.path.join(root_dir + '/TrainData/Coronal', '*.png'))): train_images_coronal.append(file) train_images_coronal = natsort.natsorted(train_images_coronal) train_labels_coronal = [] for file in sorted(glob(os.path.join(root_dir + '/TrainLabels/Coronal', '*.png'))): train_labels_coronal.append(file) train_labels_coronal= natsort.natsorted(train_labels_coronal) val_images_coronal = [] for file in sorted(glob(os.path.join(root_dir + '/ValData/Coronal', '*.png'))): val_images_coronal.append(file) val_images_coronal = natsort.natsorted(val_images_coronal) val_labels_coronal = [] for file in sorted(glob(os.path.join(root_dir + '/ValLabels/Coronal', '*.png'))): val_labels_coronal.append(file) val_labels_coronal = natsort.natsorted(val_labels_coronal) train_files_coronal = [{'image': image_name, 'label': label_name} for image_name, label_name in zip(train_images_coronal, train_labels_coronal)] val_files_coronal = [{'image': image_name, 'label': label_name} for image_name, label_name in zip(val_images_coronal, val_labels_coronal)]</code>现在我们将应用以下变换:
LoadImaged:加载图像数据和元数据。我们使用' PILReader '来加载图像和标签文件。ensure_channel_first设置为True,将图像数组形状转换为通道优先。
Rotate90d:我们将图像和标签旋转90度,因为当我们下载它们时,它们方向是不正确的。
ToTensord:将输入的图像和标签转换为张量。
NormalizeIntensityd:对输入进行规范化。
<code>train_transforms = Compose([ LoadImaged(keys = ['image', 'label'], reader=PILReader(converter=lambda image: image.convert("L")), ensure_channel_first = True), Rotate90d(keys = ['image', 'label'], k = 2), ToTensord(keys = ['image', 'label']), NormalizeIntensityd(keys = ['image'])]) val_transforms = Compose([ LoadImaged(keys = ['image', 'label'], reader=PILReader(converter=lambda image: image.convert("L")), ensure_channel_first = True), Rotate90d(keys = ['image', 'label'], k = 2), ToTensord(keys = ['image', 'label']), NormalizeIntensityd(keys = ['image'])])</code>MaskColorMap将我们定义了一个新的转换,将相应的像素值以一种格式映射为多个标签。这种转换在语义分割中是必不可少的,因为我们必须为每个可能的类别提供二元特征。One-Hot Encoding将对应于原始类别的每个样本的特征赋值为1。
因为OASIS-1数据集只有3个大脑结构标签,对于更详细的分割,理想的情况是像他们在研究论文中那样对28个皮质结构进行注释。在OASIS-1下载说明中,可以找到使用FreeSurfer获得的更多大脑结构的标签。
所以本文将分割更多的神经解剖结构。我们要将模型的参数num_classes修改为相应的标签数量,以便模型的输出是具有N个通道的特征映射,等于num_classes。
为了简化本教程,我们将使用以下标签,比OASIS-1但是要比FreeSurfer的少:
- Label 0: Background
- Label 1: LeftCerebralExterior
- Label 2: LeftWhiteMatter
- Label 3: LeftCerebralCortex
所以MaskColorMap的代码如下:
<code>class MaskColorMap(Enum):Background = (30)LeftCerebralExterior = (91)LeftWhiteMatter = (137)LeftCerebralCortex = (215)</code>
数据集和数据加载
数据集和数据加载器从存储中提取数据,并将其分批发送给训练循环。这里我们使用monai.data.Dataset加载之前定义的训练和验证字典,并对输入数据应用相应的转换。dataloader用于将数据集加载到内存中。我们将为训练和验证以及每个视图定义一个数据集和数据加载器。
为了方便演示,我们使用通过使用torch.utils.data.Subset,在指定的索引处创建一个子集,只是用部分数据训练加快演示速度。
<code>train_dataset_coronal = Dataset(data=train_files_coronal, transform = train_transforms) train_loader_coronal = DataLoader(train_dataset_coronal, batch_size = 1, shuffle = True) val_dataset_coronal = Dataset(data = val_files_coronal, transform = val_transforms) val_loader_coronal = DataLoader(val_dataset_coronal, batch_size = 1, shuffle = False) # We will use a subset of the dataset subset_train = list(range(90, len(train_dataset_coronal), 120)) train_dataset_coronal_subset = torch.utils.data.Subset(train_dataset_coronal, subset_train) train_loader_coronal_subset = DataLoader(train_dataset_coronal_subset, batch_size = 1, shuffle = True) subset_val = list(range(90, len(val_dataset_coronal), 50)) val_dataset_coronal_subset = torch.utils.data.Subset(val_dataset_coronal, subset_val) val_loader_coronal_subset = DataLoader(val_dataset_coronal_subset, batch_size = 1, shuffle = False)</code>
定义模型
给定一组MRI脑扫描I = {I1,…In}及其对应的分割S = {S1,…Sn},我们想要学习一个函数fseg: I -> S。我们将这个函数表示为F-CNN模型,称为QuickNAT:
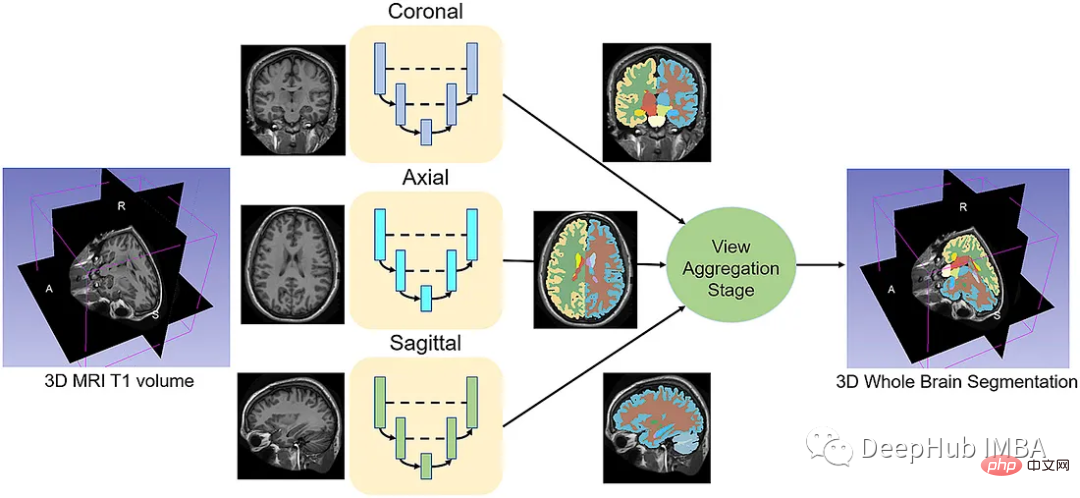
QuickNAT由三个二维f - cnn组成,分别在coronal, axial, sagittal视图上操作,然后通过聚合步骤推断最终的分割结果,该分割结果由三个网络的概率图组合而成。每个F-CNN都有一个编码器/解码器架构,其中有4个编码器和4个解码器,并由瓶颈层分隔。最后一层是带有softmax的分类器块。该架构还包括每个编码器/解码器块内的残差链接。
<code>class QuickNat(nn.Module): """A PyTorch implementation of QuickNAT """ def __init__(self, params): """:param params: {'num_channels':1,'num_filters':64,'kernel_h':5,'kernel_w':5,'stride_conv':1,'pool':2,'stride_pool':2,'num_classes':28'se_block': False,'drop_out':0.2}""" super(QuickNat, self).__init__() # from monai.networks.blocks import squeeze_and_excitation as se # self.cSE = ChannelSELayer(num_channels, reduction_ratio) # self.encode1 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE) # params["num_channels"] = params["num_filters"] # self.encode2 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE) # self.encode3 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE) # self.encode4 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE) # self.bottleneck = sm.DenseBlock(params, se_block_type=se.SELayer.CSSE) # params["num_channels"] = params["num_filters"] * 2 # self.decode1 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE) # self.decode2 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE) # self.decode3 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE) # self.decode4 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE) # self.encode1 = EncoderBlock(params, se_block_type=se.ChannelSELayer) self.encode1 = EncoderBlock(params, se_block_type=se.SELayer.CSSE) params["num_channels"] = params["num_filters"] self.encode2 = EncoderBlock(params, se_block_type=se.SELayer.CSSE) self.encode3 = EncoderBlock(params, se_block_type=se.SELayer.CSSE) self.encode4 = EncoderBlock(params, se_block_type=se.SELayer.CSSE) self.bottleneck = DenseBlock(params, se_block_type=se.SELayer.CSSE) params["num_channels"] = params["num_filters"] * 2 self.decode1 = DecoderBlock(params, se_block_type=se.SELayer.CSSE) self.decode2 = DecoderBlock(params, se_block_type=se.SELayer.CSSE) self.decode3 = DecoderBlock(params, se_block_type=se.SELayer.CSSE) self.decode4 = DecoderBlock(params, se_block_type=se.SELayer.CSSE) params["num_channels"] = params["num_filters"] self.classifier = ClassifierBlock(params) def forward(self, input): """:param input: X:return: probabiliy map """ e1, out1, ind1 = self.encode1.forward(input) e2, out2, ind2 = self.encode2.forward(e1) e3, out3, ind3 = self.encode3.forward(e2) e4, out4, ind4 = self.encode4.forward(e3) bn = self.bottleneck.forward(e4) d4 = self.decode4.forward(bn, out4, ind4) d3 = self.decode1.forward(d4, out3, ind3) d2 = self.decode2.forward(d3, out2, ind2) d1 = self.decode3.forward(d2, out1, ind1) prob = self.classifier.forward(d1) return prob def enable_test_dropout(self): """Enables test time drop out for uncertainity:return:""" attr_dict = self.__dict__["_modules"] for i in range(1, 5): encode_block, decode_block = ( attr_dict["encode" + str(i)], attr_dict["decode" + str(i)],) encode_block.drop_out = encode_block.drop_out.apply(nn.Module.train) decode_block.drop_out = decode_block.drop_out.apply(nn.Module.train) @property def is_cuda(self): """Check if model parameters are allocated on the GPU.""" return next(self.parameters()).is_cuda def save(self, path): """Save model with its parameters to the given path. Conventionally thepath should end with '*.model'. Inputs:- path: path string""" print("Saving model... %s" % path) torch.save(self.state_dict(), path) def predict(self, X, device=0, enable_dropout=False): """Predicts the output after the model is trained.Inputs:- X: Volume to be predicted""" self.eval() print("tensor size before transformation", X.shape) if type(X) is np.ndarray: # X = torch.tensor(X, requires_grad=False).type(torch.FloatTensor) X = ( torch.tensor(X, requires_grad=False).type(torch.FloatTensor).cuda(device, non_blocking=True)) elif type(X) is torch.Tensor and not X.is_cuda: X = X.type(torch.FloatTensor).cuda(device, non_blocking=True) print("tensor size ", X.shape) if enable_dropout: self.enable_test_dropout() with torch.no_grad(): out = self.forward(X) max_val, idx = torch.max(out, 1) idx = idx.data.cpu().numpy() prediction = np.squeeze(idx) print("prediction shape", prediction.shape) del X, out, idx, max_val return prediction</code>损失函数
神经网络的训练需要一个损失函数来计算模型误差。训练的目标是最小化预测输出和目标输出之间的损失。我们的模型使用Dice Loss 和Weighted Logistic Loss的联合损失函数进行优化,其中权重补偿数据中的高类不平衡,并鼓励正确分割解剖边界。
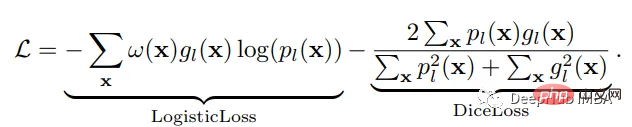
优化器
优化算法允许我们继续更新模型的参数并最小化损失函数的值,我们设置了以下的超参数:
学习率:初始设置为0.1,10次后降低1阶。这可以通过学习率调度器来实现。
权重衰减:0.0001。
批量大小:1。
动量:设置为0.95的高值,以补偿由于小批量大小而产生的噪声梯度。
训练网络
现在可以训练模型了。对于QuickNAT需要在3个(coronal, axial, sagittal)2d切片上训练3个模型。然后再聚合步骤中组合三个模型的概率生成最终结果,但是本文中只演示在coronal视图的2D切片上训练一个F-CNN模型,因为其他两个与之类似。
<code>num_epochs = 20 start_epoch = 1 val_interval = 1 train_loss_epoch_values = [] val_loss_epoch_values = [] best_ds_mean = -1 best_ds_mean_epoch = -1 ds_mean_train_values = [] ds_mean_val_values = [] # ds_LCE_values = [] # ds_LWM_values = [] # ds_LCC_values = [] print("START TRAINING. : model name = ", "quicknat") for epoch in range(start_epoch, num_epochs): print("==== Epoch ["+ str(epoch) + " / "+ str(num_epochs)+ "] DONE ====") checkpoint_name = CHECKPOINT_DIR + "/checkpoint_epoch_" + str(epoch) + "." + CHECKPOINT_EXTENSION print(checkpoint_name) state = { "epoch": epoch, "arch": "quicknat", "state_dict": model_coronal.state_dict(), "optimizer": optimizer.state_dict(), "scheduler": scheduler.state_dict(),} save_checkpoint(state = state, filename = checkpoint_name) print("\n==== Epoch [ %d / %d ] START ====" % (epoch, num_epochs)) steps_per_epoch = len(train_dataset_coronal_subset) / train_loader_coronal_subset.batch_size model_coronal.train() train_loss_epoch = 0 val_loss_epoch = 0 step = 0 predictions_train = [] labels_train = [] predictions_val = [] labels_val = [] for i_batch, sample_batched in enumerate(train_loader_coronal_subset): inputs = sample_batched['image'].type(torch.FloatTensor) labels = sample_batched['label'].type(torch.LongTensor) # print(f"Train Input Shape: {inputs.shape}") labels = labels.squeeze(1) _img_channels, _img_height, _img_width = labels.shape encoded_label= np.zeros((_img_height, _img_width, 1)).astype(int) for j, cls in enumerate(MaskColorMap): encoded_label[np.all(labels == cls.value, axis = 0)] = j labels = encoded_label labels = torch.from_numpy(labels) labels = torch.permute(labels, (2, 1, 0)) # print(f"Train Label Shape: {labels.shape}") # plt.title("Train Label") # plt.imshow(labels[0, :, :]) # plt.show() optimizer.zero_grad() outputs = model_coronal(inputs) loss = loss_function(outputs, labels) loss.backward() optimizer.step() scheduler.step() with torch.no_grad(): _, batch_output = torch.max(outputs, dim = 1) # print(f"Train Prediction Shape: {batch_output.shape}") # plt.title("Train Prediction") # plt.imshow(batch_output[0, :, :]) # plt.show() predictions_train.append(batch_output.cpu()) labels_train.append(labels.cpu()) train_loss_epoch += loss.item() print(f"{step}/{len(train_dataset_coronal_subset) // train_loader_coronal_subset.batch_size}, Training_loss: {loss.item():.4f}") step += 1 predictions_train_arr, labels_train_arr = torch.cat(predictions_train), torch.cat(labels_train) # print(predictions_train_arr.shape) dice_metric(predictions_train_arr, labels_train_arr) ds_mean_train = dice_metric.aggregate().item() ds_mean_train_values.append(ds_mean_train) dice_metric.reset() train_loss_epoch /= step train_loss_epoch_values.append(train_loss_epoch) print(f"Epoch {epoch + 1} Train Average Loss: {train_loss_epoch:.4f}") if (epoch + 1) % val_interval == 0: model_coronal.eval() step = 0 with torch.no_grad(): for i_batch, sample_batched in enumerate(val_loader_coronal_subset): inputs = sample_batched['image'].type(torch.FloatTensor) labels = sample_batched['label'].type(torch.LongTensor) # print(f"Val Input Shape: {inputs.shape}") labels = labels.squeeze(1) integer_encoded_labels = [] _img_channels, _img_height, _img_width = labels.shape encoded_label= np.zeros((_img_height, _img_width, 1)).astype(int) for j, cls in enumerate(MaskColorMap): encoded_label[np.all(labels == cls.value, axis = 0)] = j labels = encoded_label labels = torch.from_numpy(labels) labels = torch.permute(labels, (2, 1, 0)) # print(f"Val Label Shape: {labels.shape}") # plt.title("Val Label") # plt.imshow(labels[0, :, :]) # plt.show() val_outputs = model_coronal(inputs) val_loss = loss_function(val_outputs, labels) predicted = torch.argmax(val_outputs, dim = 1) # print(f"Val Prediction Shape: {predicted.shape}") # plt.title("Val Prediction") # plt.imshow(predicted[0, :, :]) # plt.show() predictions_val.append(predicted) labels_val.append(labels) val_loss_epoch += val_loss.item() print(f"{step}/{len(val_dataset_coronal_subset) // val_loader_coronal_subset.batch_size}, Validation_loss: {val_loss.item():.4f}") step += 1 predictions_val_arr, labels_val_arr = torch.cat(predictions_val), torch.cat(labels_val) dice_metric(predictions_val_arr, labels_val_arr) # dice_metric_batch(predictions_val_arr, labels_val_arr) ds_mean_val = dice_metric.aggregate().item() ds_mean_val_values.append(ds_mean_val) # ds_mean_val_batch = dice_metric_batch.aggregate() # ds_LCE = ds_mean_val_batch[0].item() # ds_LCE_values.append(ds_LCE) # ds_LWM = ds_mean_val_batch[1].item() # ds_LWM_values.append(ds_LWM) # ds_LCC = ds_mean_val_batch[2].item() # ds_LCC_values.append(ds_LCC) dice_metric.reset() # dice_metric_batch.reset() if ds_mean_val > best_ds_mean: best_ds_mean = ds_mean_val best_ds_mean_epoch = epoch + 1 torch.save(model_coronal.state_dict(), os.path.join(BESTMODEL_DIR, "best_metric_model_coronal.pth")) print("Saved new best metric model coronal") print( f"Current Epoch: {epoch + 1} Current Mean Dice score is: {ds_mean_val:.4f}" f"\nBest Mean Dice score: {best_ds_mean:.4f} " # f"\nMean Dice score Left Cerebral Exterior: {ds_LCE:.4f} Mean Dice score Left White Matter: {ds_LWM:.4f} Mean Dice score Left Cerebral Cortex: {ds_LCC:.4f} " f"at Epoch: {best_ds_mean_epoch}") val_loss_epoch /= step val_loss_epoch_values.append(val_loss_epoch) print(f"Epoch {epoch + 1} Average Validation Loss: {val_loss_epoch:.4f}") print("FINISH.")</code>代码也是传统的Pytorch的训练步骤,就不详细解释了
绘制损失和精度曲线
训练曲线表示模型的学习情况,验证曲线表示模型泛化到未见实例的情况。我们使用matplotlib来绘制图形。还可以使用TensorBoard,它使理解和调试深度学习程序变得更容易,并且是实时的。
<code>epoch = range(1, num_epochs + 1) # Plot Loss Curves plt.figure(figsize=(18, 6)) plt.subplot(1, 3, 1) plt.plot(epoch, train_loss_epoch_values, label='Training Loss') plt.plot(epoch, val_loss_epoch_values, label='Validation Loss') plt.title('Training and Validation Loss') plt.xlabel('Epoch') plt.legend() plt.figure() plt.show() # Plot Train Dice Coefficient Curve plt.figure(figsize=(18, 6)) plt.subplot(1, 3, 2) x = [(i + 1) for i in range(len(ds_mean_train_values))] plt.plot(x, ds_mean_train_values, 'blue', label = 'Train Mean Dice Score') plt.title("Training Mean Dice Coefficient") plt.xlabel('Epoch') plt.ylabel('Mean Dice Score') plt.show() # Plot Validation Dice Coefficient Curve plt.figure(figsize=(18, 6)) plt.subplot(1, 3, 3) x = [(i + 1) for i in range(len(ds_mean_val_values))] plt.plot(x, ds_mean_val_values, 'orange', label = 'Validation Mean Dice Score') plt.title("Validation Mean Dice Coefficient") plt.xlabel('Epoch') plt.ylabel('Mean Dice Score') plt.show()</code>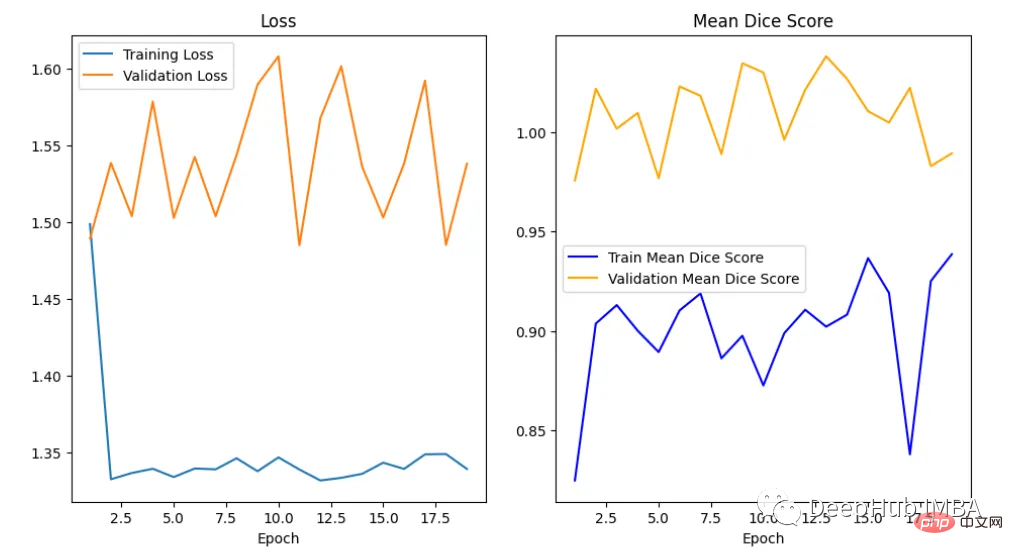
在曲线中,我们可以看到模型是过拟合的,因为验证损失上升而训练损失下降。这是深度学习算法中一个常见的陷阱,其中模型最终会记住训练数据,而无法对未见过的数据进行泛化。
避免过度拟合的技巧:
- 用更多的数据进行训练:更大的数据集可以减少过拟合。
- 数据增强:如果我们不能收集更多的数据,我们可以应用数据增强来人为地增加数据集的大小。
- 添加正则化:正则化是一种限制我们的网络学习过于复杂的模型的技术,因此可能会过度拟合。
评估网络
我们如何度量模型的性能?一个成功的预测是一个最大限度地扩大预测和真实之间的重叠。
这一目标的两个相关但不同的指标是Dice和Intersection / Union (IoU)系数,后者也被称为Jaccard系数。两个指标都在0(无重叠)和1(完全重叠)之间。
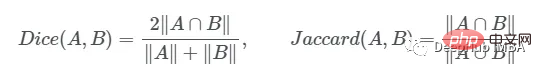
这两种指标都可以用于类似的情况,但是区别在于Dice Score倾向于平均表现,而IoU则帮助你理解最坏情况下的表现。
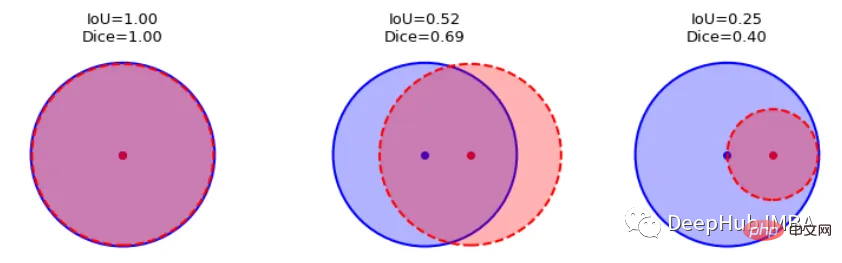
我们可以逐个类地检查度量标准,或者取所有类的平均值。这里将使用monai.metrics.DiceMetric来计算分数。一个更通用的方法是使用torchmetrics,但是因为这里使用了monai框架,所以就直接使用它内置的函数了。
我们可以看到Dice得分曲线的行为相当不寻常。主要是因为验证平均Dice得分高于1,这是不可能的,因为这个度量是在0和1之间。我们无法确定这种行为的主要原因,但我们建议在多类问题中为每个类单独提供度量计算,并始终提供可视化示例以进行可视化评估。
结果分析
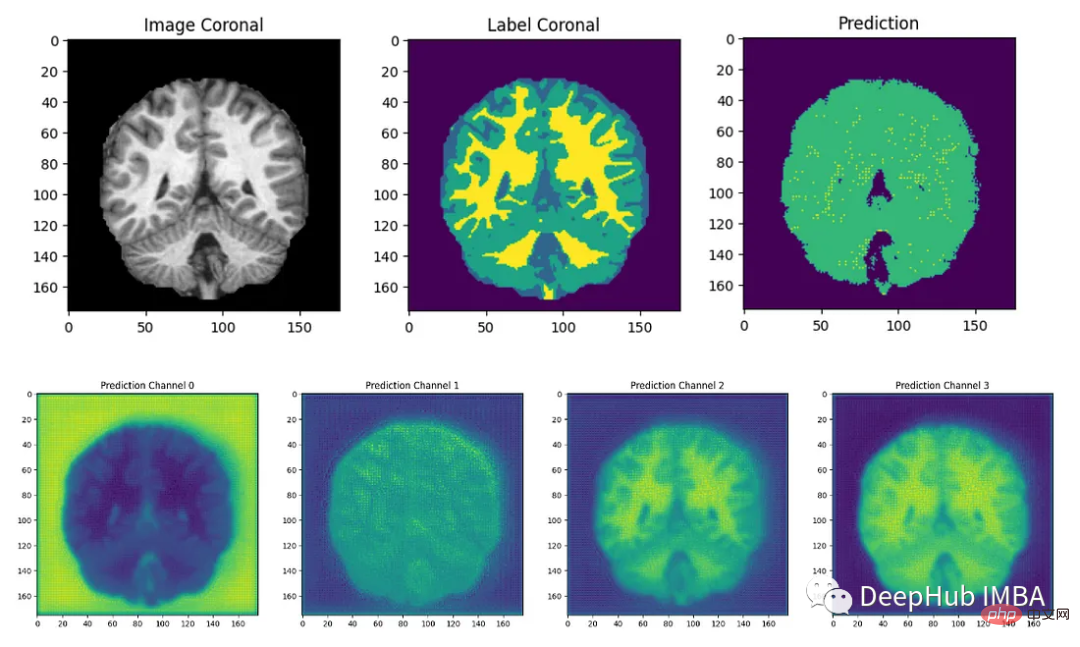
最后我们要看看模型是如何推广到未知数据的这个模型预测的几乎所有东西都是左脑白质,一些像素是左脑皮层。尽管它的预测似乎是正确的,但仍有很大的改进空间,因为我们的模型太小了,可以选择更深的模型获得更好的效果。
总结
在本文中,我们介绍了如何训练QuickNAT来完成具有挑战性的大脑分割任务。我们尽可能遵循作者在他们的研究论文中解释的学习策略,这是本教程为了方便演示只在最简单的步骤上进行了演示,文本的完整代码:
https://github.com/inesdv26/Brain-Segmentation
Das obige ist der detaillierte Inhalt vonVollständiges Codebeispiel für Deep Learning für medizinische Bilder: Segmentierung von Bildern aus MRT-Gehirnscans mit Pytorch. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!

Heiße KI -Werkzeuge

Undresser.AI Undress
KI-gestützte App zum Erstellen realistischer Aktfotos

AI Clothes Remover
Online-KI-Tool zum Entfernen von Kleidung aus Fotos.

Undress AI Tool
Ausziehbilder kostenlos

Clothoff.io
KI-Kleiderentferner

AI Hentai Generator
Erstellen Sie kostenlos Ai Hentai.

Heißer Artikel

Heiße Werkzeuge

Notepad++7.3.1
Einfach zu bedienender und kostenloser Code-Editor

SublimeText3 chinesische Version
Chinesische Version, sehr einfach zu bedienen

Senden Sie Studio 13.0.1
Leistungsstarke integrierte PHP-Entwicklungsumgebung

Dreamweaver CS6
Visuelle Webentwicklungstools

SublimeText3 Mac-Version
Codebearbeitungssoftware auf Gottesniveau (SublimeText3)

Heiße Themen
 1359
1359
 52
52
 Methoden und Schritte zur Verwendung von BERT für die Stimmungsanalyse in Python
Jan 22, 2024 pm 04:24 PM
Methoden und Schritte zur Verwendung von BERT für die Stimmungsanalyse in Python
Jan 22, 2024 pm 04:24 PM
BERT ist ein vorab trainiertes Deep-Learning-Sprachmodell, das 2018 von Google vorgeschlagen wurde. Der vollständige Name lautet BidirektionalEncoderRepresentationsfromTransformers, der auf der Transformer-Architektur basiert und die Eigenschaften einer bidirektionalen Codierung aufweist. Im Vergleich zu herkömmlichen Einweg-Codierungsmodellen kann BERT bei der Textverarbeitung gleichzeitig Kontextinformationen berücksichtigen, sodass es bei Verarbeitungsaufgaben in natürlicher Sprache eine gute Leistung erbringt. Seine Bidirektionalität ermöglicht es BERT, die semantischen Beziehungen in Sätzen besser zu verstehen und dadurch die Ausdrucksfähigkeit des Modells zu verbessern. Durch Vorschulungs- und Feinabstimmungsmethoden kann BERT für verschiedene Aufgaben der Verarbeitung natürlicher Sprache verwendet werden, wie z. B. Stimmungsanalyse und Benennung
 Analyse häufig verwendeter KI-Aktivierungsfunktionen: Deep-Learning-Praxis von Sigmoid, Tanh, ReLU und Softmax
Dec 28, 2023 pm 11:35 PM
Analyse häufig verwendeter KI-Aktivierungsfunktionen: Deep-Learning-Praxis von Sigmoid, Tanh, ReLU und Softmax
Dec 28, 2023 pm 11:35 PM
Aktivierungsfunktionen spielen beim Deep Learning eine entscheidende Rolle. Sie können nichtlineare Eigenschaften in neuronale Netze einführen und es dem Netz ermöglichen, komplexe Eingabe-Ausgabe-Beziehungen besser zu lernen und zu simulieren. Die richtige Auswahl und Verwendung von Aktivierungsfunktionen hat einen wichtigen Einfluss auf die Leistung und Trainingsergebnisse neuronaler Netze. In diesem Artikel werden vier häufig verwendete Aktivierungsfunktionen vorgestellt: Sigmoid, Tanh, ReLU und Softmax. Beginnend mit der Einführung, den Verwendungsszenarien und den Vorteilen. Nachteile und Optimierungslösungen werden besprochen, um Ihnen ein umfassendes Verständnis der Aktivierungsfunktionen zu vermitteln. 1. Sigmoid-Funktion Einführung in die Sigmoid-Funktionsformel: Die Sigmoid-Funktion ist eine häufig verwendete nichtlineare Funktion, die jede reelle Zahl auf Werte zwischen 0 und 1 abbilden kann. Es wird normalerweise verwendet, um das zu vereinheitlichen
 Die perfekte Kombination aus PyCharm und PyTorch: detaillierte Installations- und Konfigurationsschritte
Feb 21, 2024 pm 12:00 PM
Die perfekte Kombination aus PyCharm und PyTorch: detaillierte Installations- und Konfigurationsschritte
Feb 21, 2024 pm 12:00 PM
PyCharm ist eine leistungsstarke integrierte Entwicklungsumgebung (IDE) und PyTorch ist ein beliebtes Open-Source-Framework im Bereich Deep Learning. Im Bereich maschinelles Lernen und Deep Learning kann die Verwendung von PyCharm und PyTorch für die Entwicklung die Entwicklungseffizienz und Codequalität erheblich verbessern. In diesem Artikel wird detailliert beschrieben, wie PyTorch in PyCharm installiert und konfiguriert wird, und es werden spezifische Codebeispiele angehängt, um den Lesern zu helfen, die leistungsstarken Funktionen dieser beiden besser zu nutzen. Schritt 1: Installieren Sie PyCharm und Python
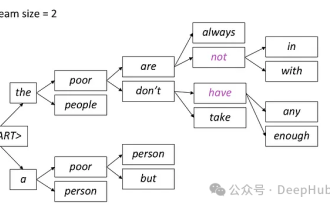 Einführung in fünf Stichprobenmethoden bei Aufgaben zur Generierung natürlicher Sprache und bei der Implementierung von Pytorch-Code
Feb 20, 2024 am 08:50 AM
Einführung in fünf Stichprobenmethoden bei Aufgaben zur Generierung natürlicher Sprache und bei der Implementierung von Pytorch-Code
Feb 20, 2024 am 08:50 AM
Bei Aufgaben zur Generierung natürlicher Sprache ist die Stichprobenmethode eine Technik, um eine Textausgabe aus einem generativen Modell zu erhalten. In diesem Artikel werden fünf gängige Methoden erläutert und mit PyTorch implementiert. 1. GreedyDecoding Bei der Greedy-Decodierung sagt das generative Modell die Wörter der Ausgabesequenz basierend auf der Eingabesequenz Zeit Schritt für Zeit voraus. In jedem Zeitschritt berechnet das Modell die bedingte Wahrscheinlichkeitsverteilung jedes Wortes und wählt dann das Wort mit der höchsten bedingten Wahrscheinlichkeit als Ausgabe des aktuellen Zeitschritts aus. Dieses Wort wird zur Eingabe für den nächsten Zeitschritt und der Generierungsprozess wird fortgesetzt, bis eine Abschlussbedingung erfüllt ist, beispielsweise eine Sequenz mit einer bestimmten Länge oder eine spezielle Endmarkierung. Das Merkmal von GreedyDecoding besteht darin, dass die aktuelle bedingte Wahrscheinlichkeit jedes Mal die beste ist
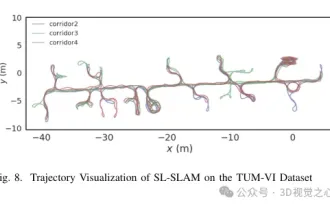 Jenseits von ORB-SLAM3! SL-SLAM: Szenen mit wenig Licht, starkem Jitter und schwacher Textur werden verarbeitet
May 30, 2024 am 09:35 AM
Jenseits von ORB-SLAM3! SL-SLAM: Szenen mit wenig Licht, starkem Jitter und schwacher Textur werden verarbeitet
May 30, 2024 am 09:35 AM
Heute diskutieren wir darüber, wie Deep-Learning-Technologie die Leistung von visionbasiertem SLAM (Simultaneous Localization and Mapping) in komplexen Umgebungen verbessern kann. Durch die Kombination von Methoden zur Tiefenmerkmalsextraktion und Tiefenanpassung stellen wir hier ein vielseitiges hybrides visuelles SLAM-System vor, das die Anpassung in anspruchsvollen Szenarien wie schlechten Lichtverhältnissen, dynamischer Beleuchtung, schwach strukturierten Bereichen und starkem Jitter verbessern soll. Unser System unterstützt mehrere Modi, einschließlich erweiterter Monokular-, Stereo-, Monokular-Trägheits- und Stereo-Trägheitskonfigurationen. Darüber hinaus wird analysiert, wie visuelles SLAM mit Deep-Learning-Methoden kombiniert werden kann, um andere Forschungen zu inspirieren. Durch umfangreiche Experimente mit öffentlichen Datensätzen und selbst abgetasteten Daten demonstrieren wir die Überlegenheit von SL-SLAM in Bezug auf Positionierungsgenauigkeit und Tracking-Robustheit.
 Latente Raumeinbettung: Erklärung und Demonstration
Jan 22, 2024 pm 05:30 PM
Latente Raumeinbettung: Erklärung und Demonstration
Jan 22, 2024 pm 05:30 PM
Latent Space Embedding (LatentSpaceEmbedding) ist der Prozess der Abbildung hochdimensionaler Daten auf niedrigdimensionalen Raum. Im Bereich des maschinellen Lernens und des tiefen Lernens handelt es sich bei der Einbettung latenter Räume normalerweise um ein neuronales Netzwerkmodell, das hochdimensionale Eingabedaten in einen Satz niedrigdimensionaler Vektordarstellungen abbildet. Dieser Satz von Vektoren wird oft als „latente Vektoren“ oder „latent“ bezeichnet Kodierungen". Der Zweck der Einbettung latenter Räume besteht darin, wichtige Merkmale in den Daten zu erfassen und sie in einer prägnanteren und verständlicheren Form darzustellen. Durch die Einbettung latenter Räume können wir Vorgänge wie das Visualisieren, Klassifizieren und Clustern von Daten im niedrigdimensionalen Raum durchführen, um die Daten besser zu verstehen und zu nutzen. Die Einbettung latenter Räume findet in vielen Bereichen breite Anwendung, z. B. bei der Bilderzeugung, der Merkmalsextraktion, der Dimensionsreduzierung usw. Die Einbettung des latenten Raums ist das Wichtigste
 Implementierung eines Rauschentfernungs-Diffusionsmodells mit PyTorch
Jan 14, 2024 pm 10:33 PM
Implementierung eines Rauschentfernungs-Diffusionsmodells mit PyTorch
Jan 14, 2024 pm 10:33 PM
Bevor wir das Funktionsprinzip des Denoising Diffusion Probabilistic Model (DDPM) im Detail verstehen, wollen wir zunächst einige Aspekte der Entwicklung generativer künstlicher Intelligenz verstehen, die auch zu den Grundlagenforschungen von DDPM gehört. VAEVAE verwendet einen Encoder, einen probabilistischen latenten Raum und einen Decoder. Während des Trainings sagt der Encoder den Mittelwert und die Varianz jedes Bildes voraus und tastet diese Werte aus einer Gaußschen Verteilung ab. Das Ergebnis der Abtastung wird an den Decoder weitergeleitet, der das Eingabebild in eine dem Ausgabebild ähnliche Form umwandelt. Zur Berechnung des Verlusts wird die KL-Divergenz verwendet. Ein wesentlicher Vorteil von VAE ist die Fähigkeit, vielfältige Bilder zu erzeugen. In der Abtastphase kann man direkt aus der Gaußschen Verteilung Stichproben ziehen und über den Decoder neue Bilder erzeugen. GAN hat in nur einem Jahr große Fortschritte bei Variational Autoencodern (VAEs) gemacht.
 Tutorial zur Installation von PyCharm mit PyTorch
Feb 24, 2024 am 10:09 AM
Tutorial zur Installation von PyCharm mit PyTorch
Feb 24, 2024 am 10:09 AM
Als leistungsstarkes Deep-Learning-Framework wird PyTorch häufig in verschiedenen maschinellen Lernprojekten eingesetzt. Als leistungsstarke integrierte Python-Entwicklungsumgebung kann PyCharm auch bei der Umsetzung von Deep-Learning-Aufgaben eine gute Unterstützung bieten. In diesem Artikel wird die Installation von PyTorch in PyCharm ausführlich vorgestellt und spezifische Codebeispiele bereitgestellt, um den Lesern den schnellen Einstieg in die Verwendung von PyTorch für Deep-Learning-Aufgaben zu erleichtern. Schritt 1: Installieren Sie PyCharm. Zuerst müssen wir sicherstellen, dass wir es haben



