 Technologie-Peripheriegeräte
Technologie-Peripheriegeräte
 KI
KI
 NVIDIA, Mila und Caltech veröffentlichen gemeinsam ein multimodales molekulares Struktur-Text-Modell, das LLM mit der Arzneimittelforschung kombiniert
NVIDIA, Mila und Caltech veröffentlichen gemeinsam ein multimodales molekulares Struktur-Text-Modell, das LLM mit der Arzneimittelforschung kombiniert
NVIDIA, Mila und Caltech veröffentlichen gemeinsam ein multimodales molekulares Struktur-Text-Modell, das LLM mit der Arzneimittelforschung kombiniert

Autor |. Liu Shengchao
Herausgeber |. Kaixia
Seit 2021 hat die Kombination aus großer Sprache und Multimodalität die Forschungsgemeinschaft für maschinelles Lernen erobert.
Können wir diese Techniken angesichts der Entwicklung großer Modelle und multimodaler Anwendungen auf die Arzneimittelforschung anwenden? Und können diese Textbeschreibungen in natürlicher Sprache neue Perspektiven für dieses herausfordernde Problem eröffnen? Die Antwort lautet „Ja“, und wir sind diesbezüglich optimistisch Das Institute of Technology schlägt ein multimodales Molekülstruktur-Text-Modell MoleculeSTM vor, bei dem die chemische Struktur und die Textbeschreibung von Molekülen durch kontrastive Lernstrategien gemeinsam erlernt werden.
Diese Forschung trägt den Titel „Multimodale Molekülstruktur – Textmodell für textbasiertes Abrufen und Bearbeiten“ und wurde am 18. Dezember 2023 in „Nature Machine Intelligence“ veröffentlicht.
 Link zum Papier: https://www.nature.com/articles/s42256-023-00759-6 muss neu geschrieben werden
Link zum Papier: https://www.nature.com/articles/s42256-023-00759-6 muss neu geschrieben werden
Dr. Liu Shengchao ist der erste Autor und Professor Anima Anandkumar von NVIDIA Research der korrespondierende Autor. Nie Weili, Wang Chengpeng, Lu Jiarui, Qiao Zhuoran, Liu Ling, Tang Jian und Xiao Chaowei sind Co-Autoren.
Dieses Projekt wurde von Dr. Liu Shengchao durchgeführt, nachdem er im März 2022 zu NVIDIA Research kam, unter der Leitung der Lehrer Nie Weili, Lehrer Tang Jian, Lehrer Xiao Chaowei und Lehrer Anima Anandkumar.
Dr. Liu Shengchao sagte: „Unsere Motivation bestand darin, vorläufige Untersuchungen zu LLM und Arzneimittelentwicklung durchzuführen und schließlich MoleculeSTM vorzuschlagen.“ ist sehr einfach und unkompliziert, das heißt, die Beschreibung von Molekülen kann in zwei Kategorien unterteilt werden: interne chemische Struktur und externe Funktionsbeschreibung. Hier verwenden wir eine kontrastive Vortrainingsmethode, um diese beiden Arten von Informationen auszurichten und zu verbinden. Das spezifische Diagramm ist in der Abbildung unten dargestellt
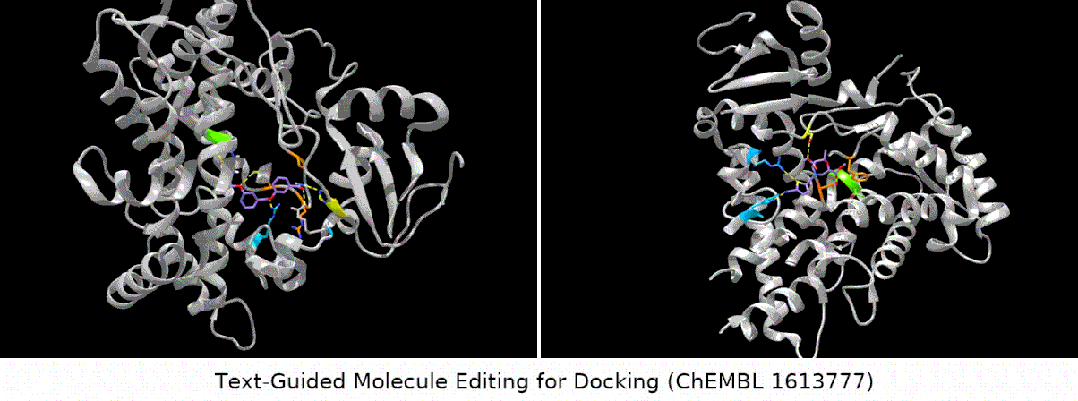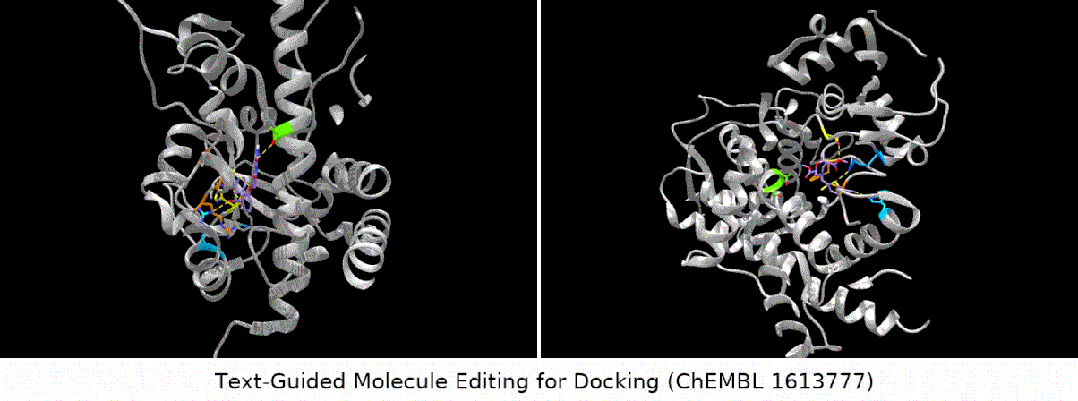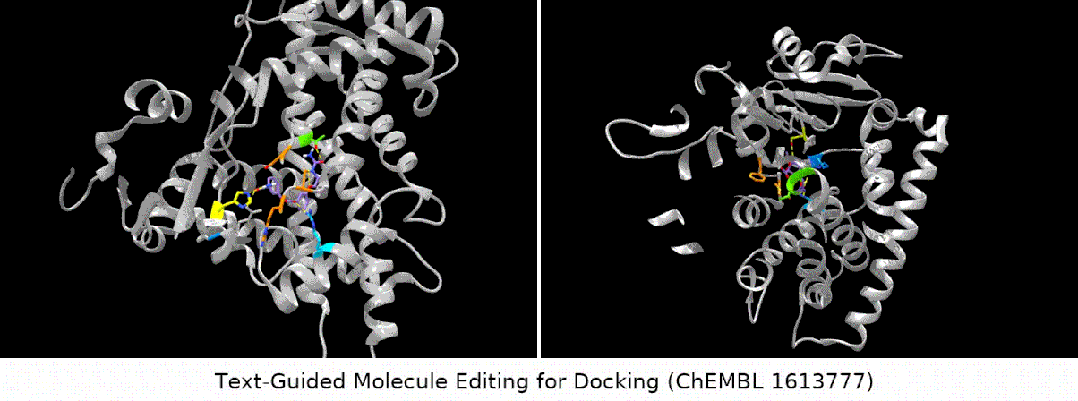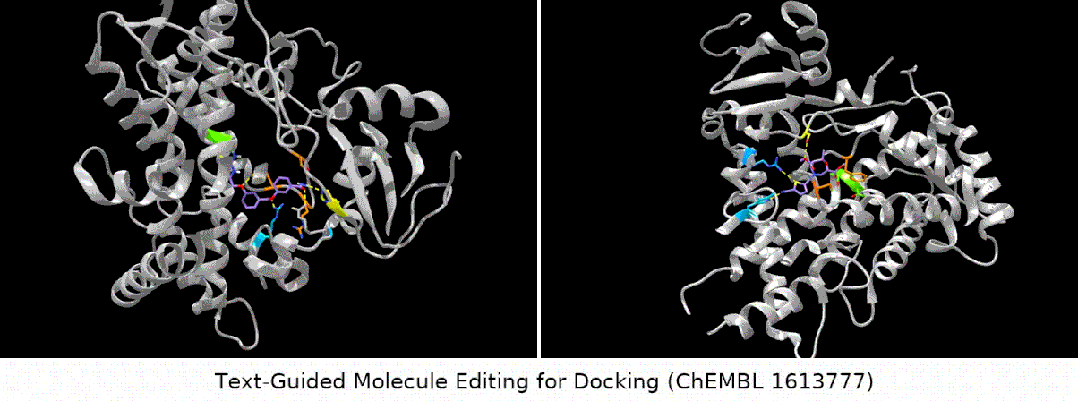 Abbildung: MoleculeSTM-Flussdiagramm.
Abbildung: MoleculeSTM-Flussdiagramm.
Und diese Ausrichtung von MoleculeSTM hat eine sehr gute Eigenschaft: Wenn es einige Aufgaben gibt, die im chemischen Raum schwer zu lösen sind, können wir sie in den natürlichen Sprachraum übertragen. Und Aufgaben in natürlicher Sprache werden aufgrund ihrer Eigenschaften relativ einfacher zu lösen sein. Auf dieser Grundlage haben wir eine Vielzahl nachgelagerter Aufgaben konzipiert, um die Wirksamkeit zu überprüfen. Im Folgenden besprechen wir einige Erkenntnisse im Detail.
Merkmale natürlicher Sprache und großer Sprachmodelle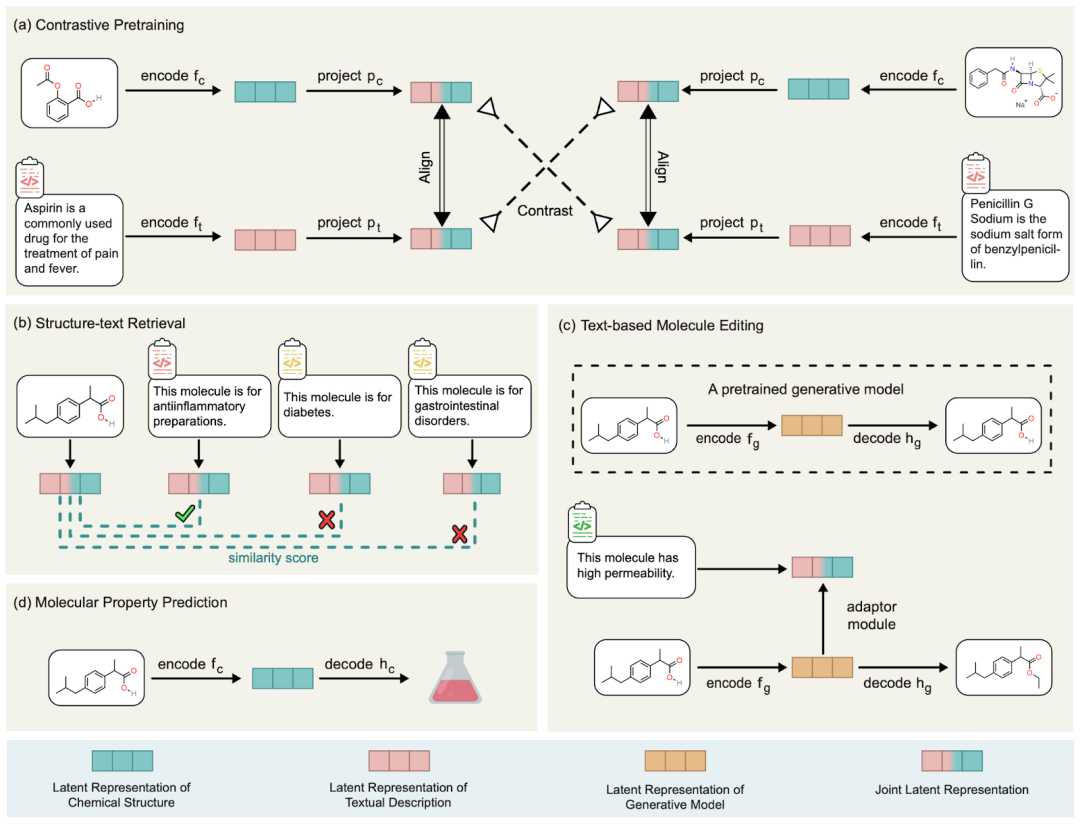
Offener Wortschatz bedeutet, dass wir das gesamte aktuelle menschliche Wissen in natürlicher Sprache ausdrücken können, sodass neues Wissen, das in der Zukunft erscheinen wird, auch in vorhandener Sprache zusammengefasst und zusammengefasst werden kann. Zusammenfassen. Wenn beispielsweise ein neues Protein auftaucht, hoffen wir, seine Funktion in natürlicher Sprache beschreiben zu können.
Kompositionalität bedeutet, dass in natürlicher Sprache ein komplexer Begriff durch mehrere einfache Begriffe gemeinsam ausgedrückt werden kann. Dies ist sehr hilfreich für Aufgaben wie die Bearbeitung mehrerer Attribute: Es ist sehr schwierig, Moleküle im chemischen Raum so zu bearbeiten, dass sie mehrere Eigenschaften gleichzeitig erfüllen, aber wir können mehrere Eigenschaften sehr einfach in natürlicher Sprache ausdrücken.
In unserer aktuellen Arbeit ChatDrug (https://arxiv.org/abs/2305.18090) haben wir die Dialogeigenschaften zwischen natürlicher Sprache und großen Sprachmodellen untersucht. Freunde, die sich dafür interessieren, können es sich ansehen
- Aufgabendesign „Durch Merkmale induziert“ bezieht sich auf die Gestaltung von Planungs- und Anordnungsaufgaben entsprechend den Merkmalen des Produkts oder Systems.
- Bei vorhandenen Sprach-Bild-Aufgaben können sie als kunstbezogene Aufgaben angesehen werden, z. B. bei der Generierung von Bildern oder Texten. Das heißt, ihre Ergebnisse sind unterschiedlich und unsicher. Bei wissenschaftlichen Entdeckungen handelt es sich jedoch um wissenschaftliche Probleme, die in der Regel zu relativ klaren Ergebnissen führen, beispielsweise zur Erzeugung kleiner Moleküle mit bestimmten Funktionen. Dies bringt größere Herausforderungen bei der Aufgabengestaltung mit sich
- Die erste Aufgabe, die wir in Betracht ziehen, besteht darin, rechnerische Simulationen durchführen und Ergebnisse erzielen zu können. Zukünftig werden Ergebnisse der Nasslaborverifizierung berücksichtigt, dies ist jedoch nicht Gegenstand der aktuellen Arbeit.
- Zweitens betrachten wir nur Probleme mit unklaren Ergebnissen. Konkrete Beispiele sind die Verbesserung der Wasserlöslichkeit oder Penetrationsfähigkeit eines bestimmten Moleküls. Manche Probleme führen zu klaren Ergebnissen, wie zum Beispiel das Hinzufügen einer bestimmten funktionellen Gruppe an einer bestimmten Position in einem Molekül. Wir glauben, dass solche Aufgaben für Arzneimittel- und Chemieexperten einfacher und unkomplizierter sind. Es kann also in Zukunft als Proof-of-Concept-Aufgabe verwendet werden, wird aber nicht zum Hauptziel der Aufgabe.
Deshalb haben wir drei große Aufgabenkategorien entwickelt:
- Zero-Shot-Strukturtext-Retrieval;
- Wir werden uns im nächsten Abschnitt auf die zweite Aufgabe konzentrieren
Diese Aufgabe besteht darin, ein Molekül und eine Beschreibung in natürlicher Sprache (z. B. zusätzliche Attribute) einzugeben Gleichzeitig ist es wünschenswert, in der Lage zu sein, komplexe sprachliche Textbeschreibungen neuer Moleküle auszugeben. Dabei handelt es sich um eine textgesteuerte Lead-Optimierung. Die spezifische Methode besteht darin, das bereits trainierte Molekülerzeugungsmodell und unser vorab trainiertes MoleculeSTM zu verwenden, um die Ausrichtung ihrer latenten Räume zu lernen, eine Latentrauminterpolation durchzuführen und dann die Zielmoleküle durch Dekodierung zu generieren. Das Prozessdiagramm ist wie folgt.
Der Inhalt, der neu geschrieben werden muss, ist: ein zweistufiges Prozessdiagramm der textgesteuerten molekularen Bearbeitung ohne Stichprobe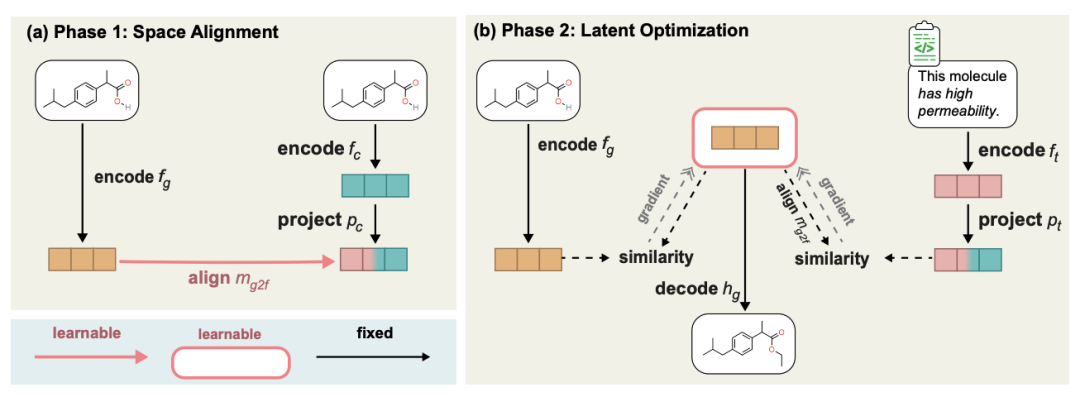 Hier zeigen wir die qualitativen Ergebnisse mehrerer Gruppen der molekularen Bearbeitung, neu formuliert wie folgt: (Die Einzelheiten zu den Ergebnissen der verbleibenden nachgelagerten Aufgaben finden Sie im Originalpapier. Wir betrachten hauptsächlich vier Arten von molekularen Bearbeitungsaufgaben:
Hier zeigen wir die qualitativen Ergebnisse mehrerer Gruppen der molekularen Bearbeitung, neu formuliert wie folgt: (Die Einzelheiten zu den Ergebnissen der verbleibenden nachgelagerten Aufgaben finden Sie im Originalpapier. Wir betrachten hauptsächlich vier Arten von molekularen Bearbeitungsaufgaben:
- Arzneimittelähnlichkeitsbearbeitung: (Anhang D.5) dient dazu, das Eingangsmolekül und das Zielmolekülarzneimittel einander näher zu bringen.
- Nachbarschaftssuche nach patentierten Arzneimitteln: Bei Arzneimitteln, die patentiert wurden, werden häufig Zwischenmedikamente zusammen gemeldet. Was wir hier tun, ist die Kombination des Zwischenwirkstoffs mit der Beschreibung in natürlicher Sprache, um zu sehen, ob daraus der endgültige Zielwirkstoff erzeugt werden kann.
- Bindungsaffinitätseditor: Wir haben mehrere ChEMBL-Assays als Ziele ausgewählt, mit dem Ziel, eine höhere Bindungsaffinität zwischen den Eingabemolekülen und den Zielen zu erreichen.
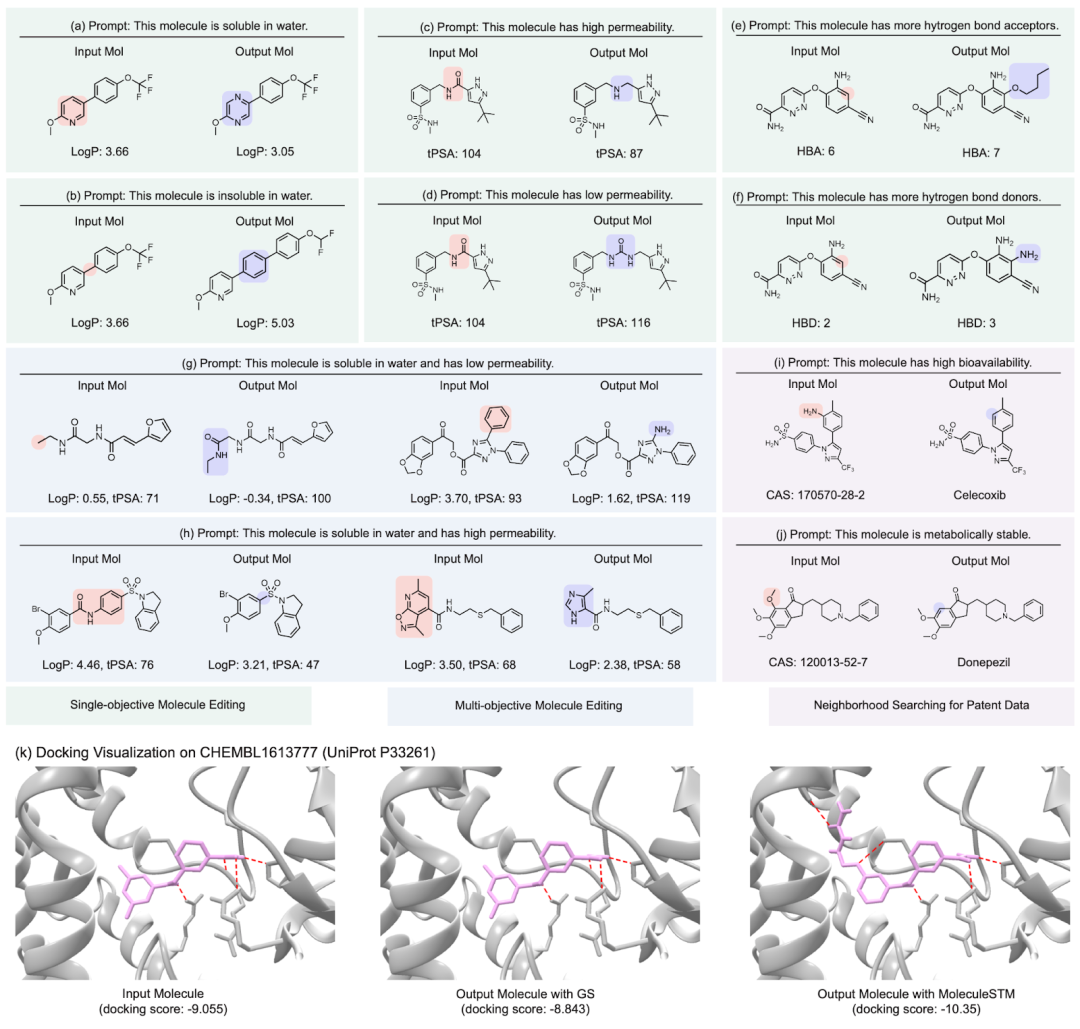 Was noch interessanter ist, ist die letzte Art von Aufgabe. Wir haben festgestellt, dass MoleculeSTM tatsächlich einen Ligandenabgleich basierend auf der Textbeschreibung der Zielproteinverbindung durchführen kann Optimierung. (Hinweis: Die Informationen zur Proteinstruktur werden hier erst nach der Auswertung bekannt.)
Was noch interessanter ist, ist die letzte Art von Aufgabe. Wir haben festgestellt, dass MoleculeSTM tatsächlich einen Ligandenabgleich basierend auf der Textbeschreibung der Zielproteinverbindung durchführen kann Optimierung. (Hinweis: Die Informationen zur Proteinstruktur werden hier erst nach der Auswertung bekannt.)
Das obige ist der detaillierte Inhalt vonNVIDIA, Mila und Caltech veröffentlichen gemeinsam ein multimodales molekulares Struktur-Text-Modell, das LLM mit der Arzneimittelforschung kombiniert. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!

Heiße KI -Werkzeuge

Undresser.AI Undress
KI-gestützte App zum Erstellen realistischer Aktfotos

AI Clothes Remover
Online-KI-Tool zum Entfernen von Kleidung aus Fotos.

Undress AI Tool
Ausziehbilder kostenlos

Clothoff.io
KI-Kleiderentferner

AI Hentai Generator
Erstellen Sie kostenlos Ai Hentai.

Heißer Artikel

Heiße Werkzeuge

Notepad++7.3.1
Einfach zu bedienender und kostenloser Code-Editor

SublimeText3 chinesische Version
Chinesische Version, sehr einfach zu bedienen

Senden Sie Studio 13.0.1
Leistungsstarke integrierte PHP-Entwicklungsumgebung

Dreamweaver CS6
Visuelle Webentwicklungstools

SublimeText3 Mac-Version
Codebearbeitungssoftware auf Gottesniveau (SublimeText3)

Heiße Themen
 1376
1376
 52
52
 „Defect Spectrum' durchbricht die Grenzen der herkömmlichen Fehlererkennung und erreicht erstmals eine hochpräzise und umfassende semantische Fehlererkennung in der Industrie.
Jul 26, 2024 pm 05:38 PM
„Defect Spectrum' durchbricht die Grenzen der herkömmlichen Fehlererkennung und erreicht erstmals eine hochpräzise und umfassende semantische Fehlererkennung in der Industrie.
Jul 26, 2024 pm 05:38 PM
In der modernen Fertigung ist die genaue Fehlererkennung nicht nur der Schlüssel zur Sicherstellung der Produktqualität, sondern auch der Kern für die Verbesserung der Produktionseffizienz. Allerdings mangelt es vorhandenen Datensätzen zur Fehlererkennung häufig an der Genauigkeit und dem semantischen Reichtum, die für praktische Anwendungen erforderlich sind, was dazu führt, dass Modelle bestimmte Fehlerkategorien oder -orte nicht identifizieren können. Um dieses Problem zu lösen, hat ein Spitzenforschungsteam bestehend aus der Hong Kong University of Science and Technology Guangzhou und Simou Technology innovativ den „DefectSpectrum“-Datensatz entwickelt, der eine detaillierte und semantisch reichhaltige groß angelegte Annotation von Industriedefekten ermöglicht. Wie in Tabelle 1 gezeigt, bietet der Datensatz „DefectSpectrum“ im Vergleich zu anderen Industriedatensätzen die meisten Fehleranmerkungen (5438 Fehlerproben) und die detaillierteste Fehlerklassifizierung (125 Fehlerkategorien).
 Das NVIDIA-Dialogmodell ChatQA wurde auf Version 2.0 weiterentwickelt, wobei die angegebene Kontextlänge 128 KB beträgt
Jul 26, 2024 am 08:40 AM
Das NVIDIA-Dialogmodell ChatQA wurde auf Version 2.0 weiterentwickelt, wobei die angegebene Kontextlänge 128 KB beträgt
Jul 26, 2024 am 08:40 AM
Die offene LLM-Community ist eine Ära, in der hundert Blumen blühen und konkurrieren. Sie können Llama-3-70B-Instruct, QWen2-72B-Instruct, Nemotron-4-340B-Instruct, Mixtral-8x22BInstruct-v0.1 und viele andere sehen hervorragende Darsteller. Allerdings weisen offene Modelle im Vergleich zu den proprietären Großmodellen GPT-4-Turbo in vielen Bereichen noch erhebliche Lücken auf. Zusätzlich zu allgemeinen Modellen wurden einige offene Modelle entwickelt, die sich auf Schlüsselbereiche spezialisieren, wie etwa DeepSeek-Coder-V2 für Programmierung und Mathematik und InternVL für visuelle Sprachaufgaben.
 Google AI gewann die Silbermedaille der IMO Mathematical Olympiad, das mathematische Argumentationsmodell AlphaProof wurde eingeführt und Reinforcement Learning ist zurück
Jul 26, 2024 pm 02:40 PM
Google AI gewann die Silbermedaille der IMO Mathematical Olympiad, das mathematische Argumentationsmodell AlphaProof wurde eingeführt und Reinforcement Learning ist zurück
Jul 26, 2024 pm 02:40 PM
Für KI ist die Mathematikolympiade kein Problem mehr. Am Donnerstag hat die künstliche Intelligenz von Google DeepMind eine Meisterleistung vollbracht: Sie nutzte KI, um meiner Meinung nach die eigentliche Frage der diesjährigen Internationalen Mathematikolympiade zu lösen, und war nur einen Schritt davon entfernt, die Goldmedaille zu gewinnen. Der IMO-Wettbewerb, der gerade letzte Woche zu Ende ging, hatte sechs Fragen zu Algebra, Kombinatorik, Geometrie und Zahlentheorie. Das von Google vorgeschlagene hybride KI-System beantwortete vier Fragen richtig und erzielte 28 Punkte und erreichte damit die Silbermedaillenstufe. Anfang dieses Monats hatte der UCLA-Professor Terence Tao gerade die KI-Mathematische Olympiade (AIMO Progress Award) mit einem Millionenpreis gefördert. Unerwarteterweise hatte sich das Niveau der KI-Problemlösung vor Juli auf dieses Niveau verbessert. Beantworten Sie die Fragen meiner Meinung nach gleichzeitig. Am schwierigsten ist es meiner Meinung nach, da sie die längste Geschichte, den größten Umfang und die negativsten Fragen haben
 Der Standpunkt der Natur: Die Erprobung künstlicher Intelligenz in der Medizin ist im Chaos. Was ist zu tun?
Aug 22, 2024 pm 04:37 PM
Der Standpunkt der Natur: Die Erprobung künstlicher Intelligenz in der Medizin ist im Chaos. Was ist zu tun?
Aug 22, 2024 pm 04:37 PM
Herausgeber | ScienceAI Basierend auf begrenzten klinischen Daten wurden Hunderte medizinischer Algorithmen genehmigt. Wissenschaftler diskutieren darüber, wer die Werkzeuge testen soll und wie dies am besten geschieht. Devin Singh wurde Zeuge, wie ein pädiatrischer Patient in der Notaufnahme einen Herzstillstand erlitt, während er lange auf eine Behandlung wartete, was ihn dazu veranlasste, den Einsatz von KI zu erforschen, um Wartezeiten zu verkürzen. Mithilfe von Triage-Daten aus den Notaufnahmen von SickKids erstellten Singh und Kollegen eine Reihe von KI-Modellen, um mögliche Diagnosen zu stellen und Tests zu empfehlen. Eine Studie zeigte, dass diese Modelle die Zahl der Arztbesuche um 22,3 % verkürzen können und die Verarbeitung der Ergebnisse pro Patient, der einen medizinischen Test benötigt, um fast drei Stunden beschleunigt. Der Erfolg von Algorithmen der künstlichen Intelligenz in der Forschung bestätigt dies jedoch nur
 Training mit Millionen von Kristalldaten zur Lösung kristallographischer Phasenprobleme, die Deep-Learning-Methode PhAI wird in Science veröffentlicht
Aug 08, 2024 pm 09:22 PM
Training mit Millionen von Kristalldaten zur Lösung kristallographischer Phasenprobleme, die Deep-Learning-Methode PhAI wird in Science veröffentlicht
Aug 08, 2024 pm 09:22 PM
Herausgeber |KX Bis heute sind die durch die Kristallographie ermittelten Strukturdetails und Präzision, von einfachen Metallen bis hin zu großen Membranproteinen, mit keiner anderen Methode zu erreichen. Die größte Herausforderung, das sogenannte Phasenproblem, bleibt jedoch die Gewinnung von Phaseninformationen aus experimentell bestimmten Amplituden. Forscher der Universität Kopenhagen in Dänemark haben eine Deep-Learning-Methode namens PhAI entwickelt, um Kristallphasenprobleme zu lösen. Ein Deep-Learning-Neuronales Netzwerk, das mithilfe von Millionen künstlicher Kristallstrukturen und den entsprechenden synthetischen Beugungsdaten trainiert wird, kann genaue Elektronendichtekarten erstellen. Die Studie zeigt, dass diese Deep-Learning-basierte Ab-initio-Strukturlösungsmethode das Phasenproblem mit einer Auflösung von nur 2 Angström lösen kann, was nur 10 bis 20 % der bei atomarer Auflösung verfügbaren Daten im Vergleich zur herkömmlichen Ab-initio-Berechnung entspricht
 Um ein neues wissenschaftliches und komplexes Frage-Antwort-Benchmark- und Bewertungssystem für große Modelle bereitzustellen, haben UNSW, Argonne, die University of Chicago und andere Institutionen gemeinsam das SciQAG-Framework eingeführt
Jul 25, 2024 am 06:42 AM
Um ein neues wissenschaftliches und komplexes Frage-Antwort-Benchmark- und Bewertungssystem für große Modelle bereitzustellen, haben UNSW, Argonne, die University of Chicago und andere Institutionen gemeinsam das SciQAG-Framework eingeführt
Jul 25, 2024 am 06:42 AM
Herausgeber | Der Frage-Antwort-Datensatz (QA) von ScienceAI spielt eine entscheidende Rolle bei der Förderung der Forschung zur Verarbeitung natürlicher Sprache (NLP). Hochwertige QS-Datensätze können nicht nur zur Feinabstimmung von Modellen verwendet werden, sondern auch effektiv die Fähigkeiten großer Sprachmodelle (LLMs) bewerten, insbesondere die Fähigkeit, wissenschaftliche Erkenntnisse zu verstehen und zu begründen. Obwohl es derzeit viele wissenschaftliche QS-Datensätze aus den Bereichen Medizin, Chemie, Biologie und anderen Bereichen gibt, weisen diese Datensätze immer noch einige Mängel auf. Erstens ist das Datenformular relativ einfach, die meisten davon sind Multiple-Choice-Fragen. Sie sind leicht auszuwerten, schränken jedoch den Antwortauswahlbereich des Modells ein und können die Fähigkeit des Modells zur Beantwortung wissenschaftlicher Fragen nicht vollständig testen. Im Gegensatz dazu offene Fragen und Antworten
 Identifizieren Sie automatisch die besten Moleküle und reduzieren Sie die Synthesekosten. Das MIT entwickelt ein Algorithmus-Framework für die Entscheidungsfindung im molekularen Design
Jun 22, 2024 am 06:43 AM
Identifizieren Sie automatisch die besten Moleküle und reduzieren Sie die Synthesekosten. Das MIT entwickelt ein Algorithmus-Framework für die Entscheidungsfindung im molekularen Design
Jun 22, 2024 am 06:43 AM
Herausgeber |. Der Einsatz von Ziluo AI bei der Rationalisierung der Arzneimittelforschung nimmt explosionsartig zu. Durchsuchen Sie Milliarden von Kandidatenmolekülen nach solchen, die möglicherweise über Eigenschaften verfügen, die für die Entwicklung neuer Medikamente erforderlich sind. Es sind so viele Variablen zu berücksichtigen, von Materialpreisen bis hin zum Fehlerrisiko, dass es keine leichte Aufgabe ist, die Kosten für die Synthese der besten Kandidatenmoleküle abzuwägen, selbst wenn Wissenschaftler KI einsetzen. Hier entwickelten MIT-Forscher SPARROW, ein quantitatives Entscheidungsalgorithmus-Framework, um automatisch die besten molekularen Kandidaten zu identifizieren und so die Synthesekosten zu minimieren und gleichzeitig die Wahrscheinlichkeit zu maximieren, dass die Kandidaten die gewünschten Eigenschaften aufweisen. Der Algorithmus bestimmte auch die Materialien und experimentellen Schritte, die zur Synthese dieser Moleküle erforderlich sind. SPARROW berücksichtigt die Kosten für die gleichzeitige Synthese einer Charge von Molekülen, da häufig mehrere Kandidatenmoleküle verfügbar sind
 SOTA Performance, eine multimodale KI-Methode zur Vorhersage der Protein-Ligand-Affinität in Xiamen, kombiniert erstmals molekulare Oberflächeninformationen
Jul 17, 2024 pm 06:37 PM
SOTA Performance, eine multimodale KI-Methode zur Vorhersage der Protein-Ligand-Affinität in Xiamen, kombiniert erstmals molekulare Oberflächeninformationen
Jul 17, 2024 pm 06:37 PM
Herausgeber |. KX Im Bereich der Arzneimittelforschung und -entwicklung ist die genaue und effektive Vorhersage der Bindungsaffinität von Proteinen und Liganden für das Arzneimittelscreening und die Arzneimitteloptimierung von entscheidender Bedeutung. Aktuelle Studien berücksichtigen jedoch nicht die wichtige Rolle molekularer Oberflächeninformationen bei Protein-Ligand-Wechselwirkungen. Auf dieser Grundlage schlugen Forscher der Universität Xiamen ein neuartiges Framework zur multimodalen Merkmalsextraktion (MFE) vor, das erstmals Informationen über Proteinoberfläche, 3D-Struktur und -Sequenz kombiniert und einen Kreuzaufmerksamkeitsmechanismus verwendet, um verschiedene Modalitäten zu vergleichen Ausrichtung. Experimentelle Ergebnisse zeigen, dass diese Methode bei der Vorhersage von Protein-Ligand-Bindungsaffinitäten Spitzenleistungen erbringt. Darüber hinaus belegen Ablationsstudien die Wirksamkeit und Notwendigkeit der Proteinoberflächeninformation und der multimodalen Merkmalsausrichtung innerhalb dieses Rahmens. Verwandte Forschungen beginnen mit „S



