 Technology peripherals
Technology peripherals
 AI
AI
 Towards a 'all-rounder' medical model, the Shanghai Jiao Tong University team releases large-scale instruction fine-tuning data, open source models and comprehensive benchmark testing
Towards a 'all-rounder' medical model, the Shanghai Jiao Tong University team releases large-scale instruction fine-tuning data, open source models and comprehensive benchmark testing
Towards a 'all-rounder' medical model, the Shanghai Jiao Tong University team releases large-scale instruction fine-tuning data, open source models and comprehensive benchmark testing
Sep 03, 2024 pm 10:12 PM
編集 | ScienceAI
最近、上海交通大学、上海 AI 研究所、チャイナモバイルなどの共同研究チームが「医学向け多用途大言語モデルの評価と構築に向けて》では、データ、評価、モデルの複数の観点から臨床医学における大規模言語モデルの適用を包括的に分析し、議論しています。
この記事に含まれるすべてのデータ、コード、モデルはオープンソースです。
概要
近年、医療分野において大規模言語モデル(LLM)は大きな進歩を遂げ、一定の成果をあげています。これらのモデルは、Medical Multiple Choice Question Answering (MCQA) ベンチマークで効率的な機能を実証し、UMLS などの専門試験でエキスパート レベルに達しているか、それを超えています。 しかし、LLM は実際の臨床シナリオでの応用にはまだ程遠いです。主な問題は、ICD コードの解釈、臨床手順の予測、電子医療記録 (EHR) データの解析におけるエラーなど、基本的な医療知識の処理におけるモデルの不十分さに集中しています。
これらの問題は重要な点を示しています。現在の評価ベンチマークは主に医療試験の多肢選択式質問に焦点を当てており、実際の臨床シナリオでの LLM の適用を適切に反映していません。 この研究は、新しい評価ベンチマークである MedS-Bench を提案しています。このベンチマークには、多肢選択式の質問が含まれるだけでなく、臨床レポートの要約、治療の推奨、診断、固有表現の認識などの 11 の高度な臨床タスクもカバーされています。 。 研究チームは、このベンチマークを通じて複数の主流の医療モデルを評価したところ、GPT-4、Claude などの最先端のモデルは、数ショットのプロンプトでも、これらの複雑な臨床業務を処理するのは困難に直面しています。
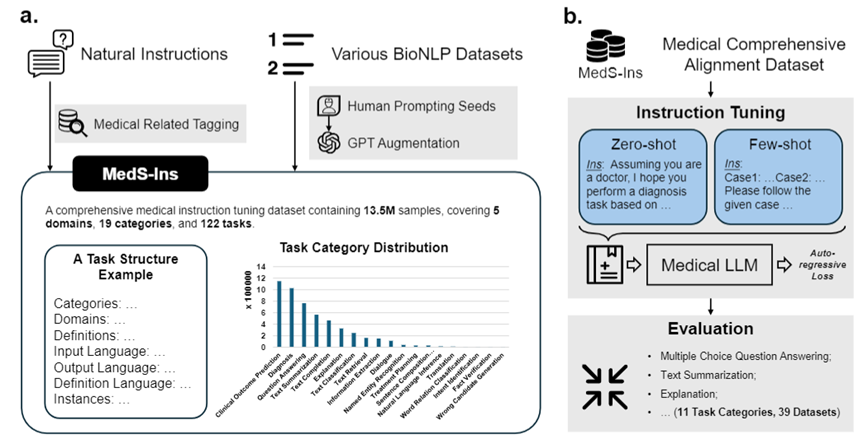
この問題を解決するために、研究チームは Super-NaturalHandling に触発されて、検査、臨床テキスト、学術論文、58 の生物医学テキストからのデータを統合する初の包括的な医療指導微調整データセット MedS-Ins を構築しました。医療知識ベースと日常会話のデータ セットには、122 の臨床タスクをカバーする 1,350 万以上のサンプルが含まれています。 これに基づいて、研究チームはオープンソースの医療言語モデルの指示を調整し、コンテキスト内学習環境におけるモデルの効果を調査しました。 この研究で開発された医療大規模言語モデル MMedIns-Llama 3 は、さまざまな臨床タスクにおいて、GPT-4 や Claude-3.5 などの既存の主要なクローズドソース モデルを上回ります。 MedS-Ins の構築により、実際の臨床シナリオにおける医療大規模言語モデルの機能が大幅に向上し、その適用範囲はオンライン チャットや多肢選択式の質疑応答の制限をはるかに超えています。 私たちは、この進歩が医療言語モデルの開発を促進するだけでなく、将来の臨床現場での人工知能の応用に新たな可能性をもたらすと信じています。テストベンチマークデータセット (MedS-Bench)
臨床応用におけるさまざまな LLM の機能を評価するために、研究チームは MedS-Bench を開発しました。は、従来の多肢選択式の質問を超えた包括的な医療ベンチマークです。以下の図に示すように、MedS-Bench は 39 の既存のデータセットから派生しており、11 のカテゴリをカバーし、合計 52 のタスクが含まれています。 MedS-Bench では、データは微調整を必要とする構造に再フォーマットされます。さらに、各タスクには手動で注釈が付けられたタスク定義が付属しています。関連する 11 のカテゴリは次のとおりです: 多肢選択式質問応答 (MCQA)、テキスト要約、情報抽出 (情報抽出)、説明と根拠、固有表現認識 (NER)、診断、治療計画、臨床転帰予測、テキスト分類、事実検証、および自然言語推論 (NLI)。
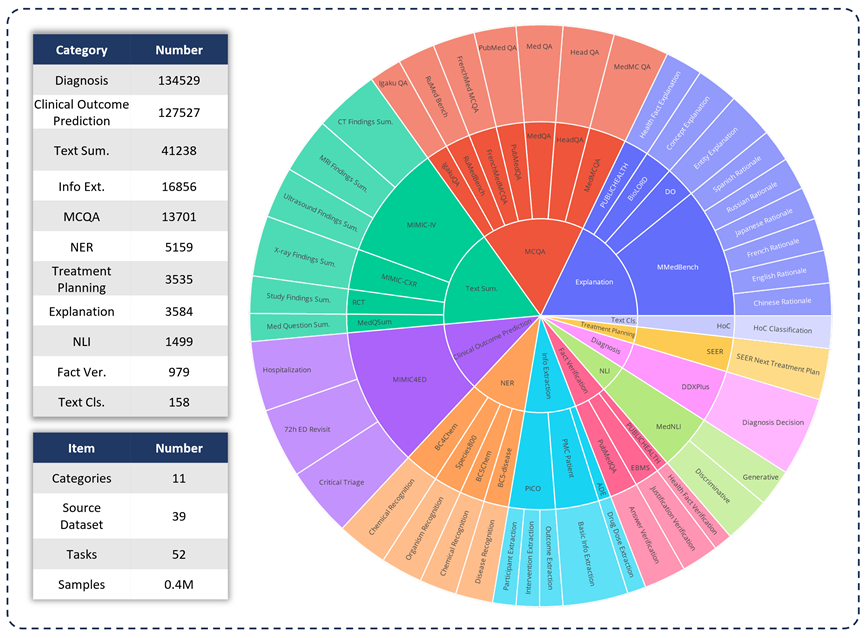
これらのタスク カテゴリの定義に加えて、研究チームは MedS-Bench のテキスト長に関する詳細な統計も実施し、以下の表に示すように、さまざまなタスクを処理するために LLM に必要な機能を識別しました。 LLM 処理タスクに必要な機能は、(i) モデルの内部知識に基づく推論、(ii) 提供されたコンテキストからの事実の取得の 2 つのカテゴリに分類されます。
大まかに言うと、前者には大規模な事前トレーニングからモデルの重みにエンコードされた知識を取得する必要があるタスクが含まれ、後者には要約や情報抽出など、提供されたコンテキストから情報を抽出する必要があるタスクが含まれます。 。表 1 に示すように、モデルがモデルから知識を呼び出すことを必要とするタスクのカテゴリは合計 8 つあり、残りの 3 つのカテゴリのタスクは、特定のコンテキストから事実を取得することを必要とします。
表 1: 使用されたテスト タスクの詳細な統計。

指示微調整データセット (MedS-Ins)
さらに、研究チームはデータセット MedS-Ins の微調整命令もオープンソース化しました。このデータセットは、5 つの異なるテキスト ソースと 19 のタスク カテゴリ、合計 122 の異なる臨床タスクをカバーしています。以下の図は、MedS-Ins の構築プロセスと統計情報をまとめたものです。
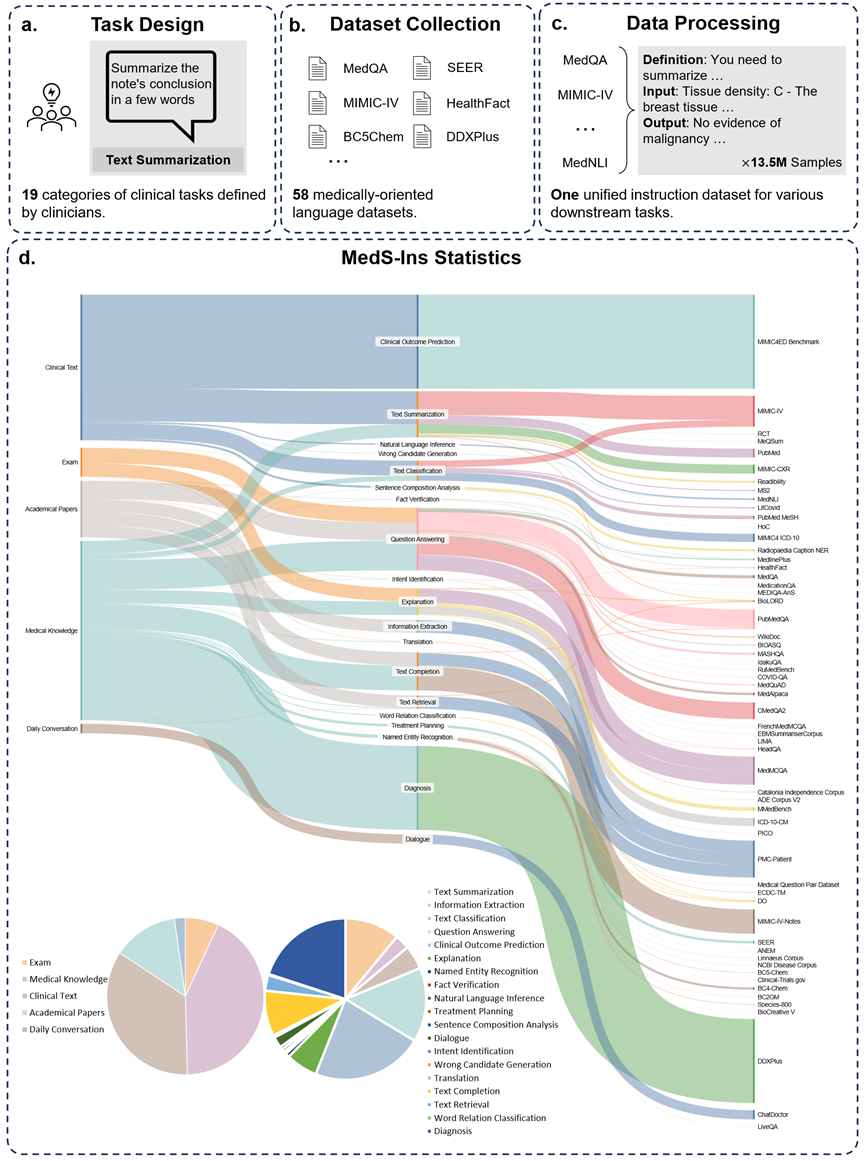
テキストソース
この論文で提案されている指示微調整データセットは、検査、臨床テキスト、学術論文、医学知識ベース、そして日常会話。
検査: このカテゴリには、さまざまな国の健康診断の問題のデータが含まれています。基礎的な医学知識から複雑な臨床手順まで、幅広い医学知識をカバーします。試験問題は医学教育のレベルを理解し評価する重要な手段ですが、試験の高度な標準化により、実際の臨床課題と比較してケースが過度に単純化されることが多いことに注意してください。データセット内のデータの 7% は試験からのものです。
臨床テキスト: このカテゴリのテキストは、病院や臨床センターでの診断、治療、予防のプロセスを含む日常的な臨床実践で作成されます。このようなテキストには、電子健康記録 (EHR)、放射線医学レポート、検査結果、フォローアップの指示、投薬の推奨事項などが含まれます。これらのテキストは疾患の診断と患者管理に不可欠であるため、LLM を効果的に臨床に応用するには正確な分析と理解が不可欠です。データセット内のデータの 35% は臨床テキストからのものです。
学術論文: このカテゴリのデータは医学研究論文からのものであり、医学研究分野における最新の発見と進歩をカバーしています。アクセスが容易で構造化されているため、学術論文からのデータの抽出は比較的簡単です。これらのデータは、モデルが最先端の医学研究情報を習得し、現代医学の発展をより深く理解できるようにモデルを導くのに役立ちます。データセット内のデータの 13% は学術論文からのものです。
医学知識ベース: このカテゴリのデータは、医学百科事典、ナレッジ グラフ、医学用語集など、よく整理された包括的な医学知識で構成されています。これらのデータは医学知識ベースの中核を形成し、医学教育と臨床現場での LLM の応用をサポートします。データセット内のデータの 43% は医学知識から得られています。
日常会話: このカテゴリのデータは、主にオンライン プラットフォームやその他の対話型シナリオからの、医師と患者の間の日常的な相談を指します。これらのデータは医療スタッフと患者の間の実際のやりとりを反映しており、患者のニーズを理解し、医療サービス全体のエクスペリエンスを向上させる上で重要な役割を果たします。データセット内のデータの 2% は日常会話から得られます。
タスクの種類
テキストに含まれる分野の分類に加えて、研究チームは MedS-Ins のサンプルのタスク カテゴリをさらに細分化しました。19 のタスク カテゴリが特定され、各カテゴリは医療大規模言語モデルが持つべき重要な機能を表しています。 。データセットを微調整し、それに応じてモデルを微調整するこの命令を構築することにより、図 2 に示すように、大規模言語モデルは医療アプリケーションを処理するために必要な機能を備えます。
MedS-Ins の 19 のタスク カテゴリには、MedS-Bench ベンチマークの 11 のカテゴリが含まれますが、これらに限定されません。追加のタスク カテゴリは、意図認識、翻訳、単語関係分類、テキスト検索、文構成要素分析、誤り候補生成、対話およびテキスト補完など、医療分野で必要なさまざまな言語および分析タスクをカバーします。一方、MCQA は一般的な Q&A 用です。一般的な Q&A や会話からさまざまな下流の臨床タスクまで、タスク カテゴリの多様性により、医療アプリケーションの包括的な理解を確実にします。
定量的比較
研究チームは、6 つの既存の主流モデル (MEDITRON、Mistral、InternLM 2、Llama 3、GPT-4、および Claude-3.5) のパフォーマンスを広範囲にテストしました。各タスク タイプについて、さまざまな既存の LLM のパフォーマンスが最初に議論され、次に提案された最終モデルである MMedIns-Llama 3 と比較されます。この記事では、すべての結果は 3 ショット プロンプトを使用して取得されています。ただし、以前の研究と一致するように、MCQA タスクではゼロショット プロンプトが使用されます。 GPT-4 や Claude 3.5 などのクローズドソース モデルには費用がかかり、コストによって制限されるため、実験ではタスクごとに 50 ~ 100 のテスト ケースのみがサンプリングされました。包括的なテストの定量化結果は表 2-8 に示されています。
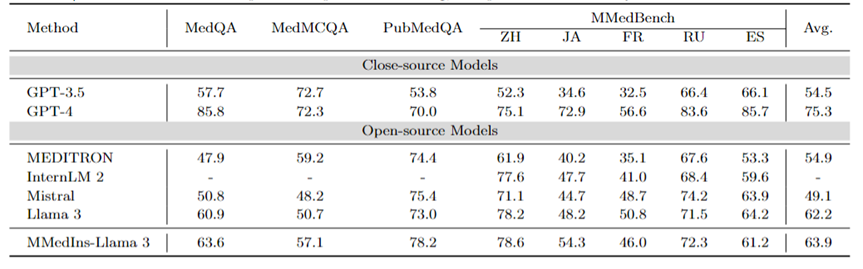
多言語 MCQA: 表 2 に、広く使用されている MCQA ベンチマークの「精度」の評価結果を示します。これらの多肢選択式質問回答データセットでは、既存の大規模言語モデルは非常に高い精度を示しています。たとえば、MedQA では、GPT-4 は人間の専門家とほぼ同等の 85.8 点に達し、Llama 3 も試験に合格できます。スコアは60.9。同様に、英語以外の言語でも、LLM は MMedBench の複数選択の精度において優れた結果を示しています。
結果は、多肢選択式の質問は既存の研究で広く考慮されているため、さまざまな LLM がそのようなタスクに特化して最適化されており、結果としてパフォーマンスが向上している可能性があることを示しています。したがって、LLM の臨床応用をさらに促進するには、より包括的なベンチマークを確立する必要があります。
表 2: 多肢選択式質問の定量的結果は、選択精度 ACC によって測定されます。
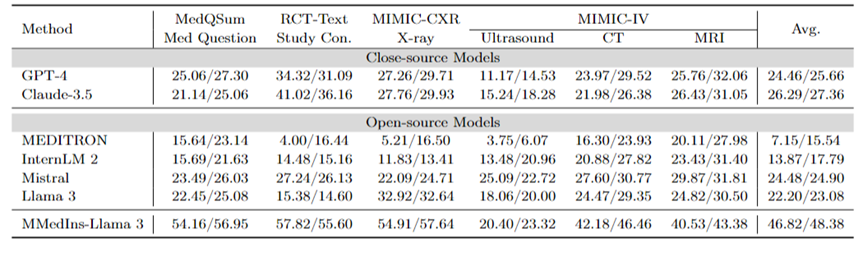
テキスト要約: 表 3 は、テキスト要約タスクにおけるさまざまな言語モデルのパフォーマンスを「BLEU/ROUGE」スコアの形式で報告します。検査は、X 線、CT、MRI、超音波、その他の医療問題など、さまざまな種類のレポートを対象としています。実験結果は、GPT-4 や Claude-3.5 などのクローズドソースの大規模言語モデルが、すべてのオープンソースの大規模言語モデルよりも優れたパフォーマンスを発揮することを示しています。
オープンソース モデルの中で、Mistral が最高の結果を示し、BLEU/ROUGE がそれぞれ 24.48/24.90 で、次に Llama 3 が 22.20/23.08 でした。
この記事で提案されている MMedIns-Llama 3 は、特定の医療教育データセット (MedS-Ins) でトレーニングされており、そのパフォーマンスは、高度なクローズドソース モデル GPT-4 や GPT-4 などの他のモデルよりも大幅に優れています。クロード - 3.5、平均スコア 46.82/48.38。
表 3: テキスト要約タスクの定量的結果。
情報抽出: 表 4 は、「精度」によるさまざまなモデルの情報抽出のパフォーマンスを示しています。 InternLM 2 は、平均スコア 81.58 でこのタスクで良好なパフォーマンスを示し、GPT-4 や Claude-3.5 などのクローズドソース モデルは、平均スコアがそれぞれ 77.49 と 78.86 で他のすべてのオープンソース モデルを上回りました。
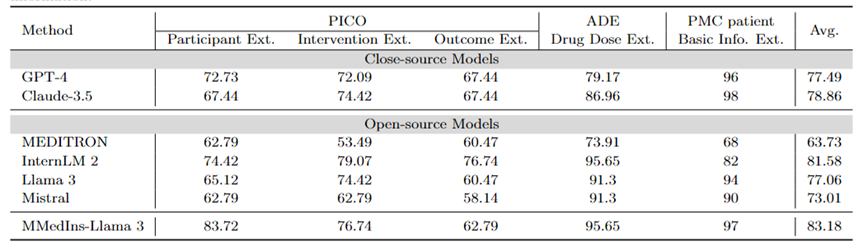
個々のタスクの結果を分析すると、ほとんどの大規模な言語モデルは、特殊な医療データと比較して、基本的な患者情報などのそれほど複雑ではない医療情報を抽出する際に優れたパフォーマンスを発揮することがわかります。たとえば、PMC 患者から基本情報を抽出する場合、ほとんどの大規模言語モデルは 90 ポイントを超えるスコアを獲得し、Claude-3.5 は 98.02 ポイントの最高スコアを達成しました。対照的に、PICO における臨床転帰抽出タスクのパフォーマンスは比較的低かった。この記事で提案されているモデル MMedIns-Llama 3 は、全体的なパフォーマンスが最も高く、平均スコアは 83.18 で、InternLM 2 モデルを 1.6 ポイント上回っています。
表 4: 情報抽出タスクの定量的結果 各指標は精度 (ACC) によって測定されます。 「Ext.」は抽出を意味し、「Info.」は情報を意味します。
医療概念の説明: 表 5 は、「BLEU/ROUGE」スコア、GPT-4 の形式でのさまざまなモデルの医療概念の説明能力を示しています。 、ラマ 3 とミストラルはこのタスクで優れたパフォーマンスを発揮します。
In contrast, Claude-3.5, InternLM 2 and MEDITRON have relatively low scores. The relatively poor performance of MEDITRON may be due to the fact that its training corpus focuses more on academic papers and guidelines, so it lacks the ability to explain medical concepts.
The final model MMedIns-Llama 3 performs significantly better than other models in all concept explanation tasks.
Table 5: Quantitative results on medical concept explanation, each indicator is measured by BLEU-1/ROUGE-1; "Exp." means Explanation.
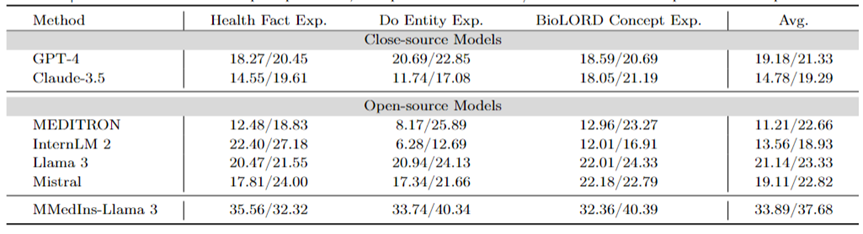
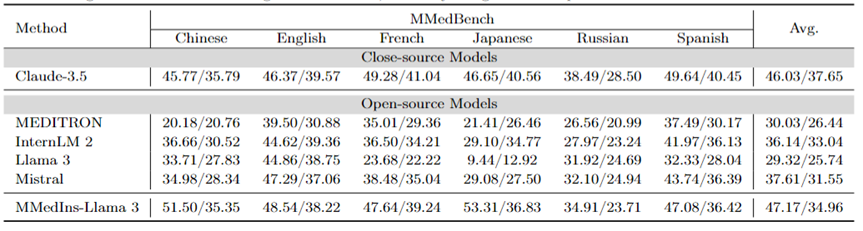
Attribution analysis (Rationale): Table 6 evaluates the performance of each model on the attribution analysis task in the form of "BLEU/ROUGE" score Performance,The inference capabilities of various models in six,languages were compared using the MMedBench dataset.
Among the tested models, the closed-source model Claude-3.5 showed the strongest performance, with an average score of 46.03/37.65. This superior performance may be due to the similarity of the task to generating COT, which is specifically enhanced in many general-purpose LLMs.
Among the open source models, Mistral and InternLM 2 showed comparable performance, with average scores of 37.61/31.55 and 30.03/26.44 respectively. Notably, GPT-4 was excluded from this evaluation because the attribution analysis portion of the MMedBench dataset primarily uses GPT-4 to generate builds, which may introduce testing bias, leading to unfair comparisons.
Consistent with the performance on the concept explanation task, the final model MMedIns-Llama 3 also demonstrated the best overall performance, with an average score of 47.17/34.96 across all languages. This excellent performance may be due to the fact that the selected base language model (MMed-Llama 3) was originally developed for multiple languages. Therefore, even if the instruction tuning does not explicitly target multilingual data, the final model still performs better than other models in multiple languages.
Table 6: Quantitative results on attribution analysis (Rationale), each indicator is measured by BLEU-1/ROUGE-1. There is no GPT-4 here because the original data is constructed based on the GPT-4 generation results and there is fairness bias, so GPT-4 is not compared.
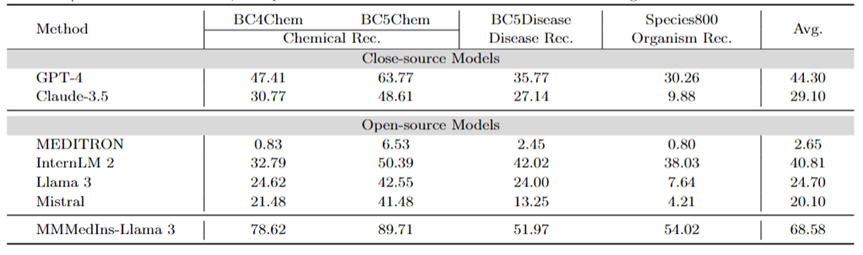
Medical Entity Extraction (NER): Table 7 tests the existing 6 models on the NER task in the form of "F1" score Performance. GPT-4 is the only model that performs well across all named entity recognition (NER) tasks, with an average F1 score of 44.30.
It performs particularly well on the BC5Chem chemical entity recognition task, scoring 63.77. InternLM 2 follows closely behind with an average F1 score of 40.81, performing well on both the BC5Chem and BC5Disease tasks. Llama 3 and Mistral have average F1 scores of 24.70 and 20.10 respectively, which are average performances. MEDITRON is not optimized for NER tasks and performs poorly in this area. MMedIns-Llama 3 performs significantly better than all other models, with an average F1 score of 68.58.
Table 7: Quantitative results on the NER task, each indicator is measured by F1-score; "Rec." stands for "recognition"
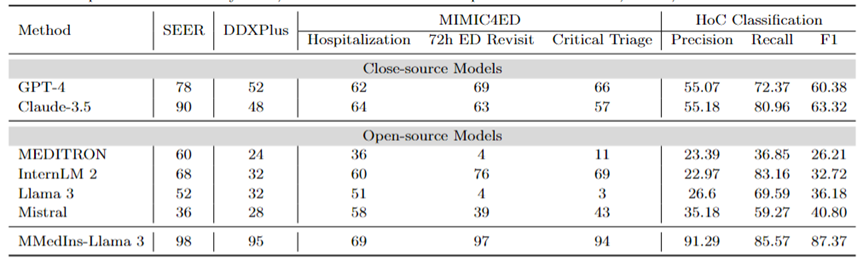
Diagnosis, Treatment Recommendation, and Clinical Outcome Prediction: Table 8 Evaluation of diagnosis, treatment recommendation, and clinical outcome using DDXPlus dataset as diagnostic benchmark, SEER dataset as treatment recommendation benchmark, and MIMIC4ED data as clinical outcome prediction task benchmark The model performance of the three major tasks is predicted, and the results are measured by accuracy, as shown in Table 8.
Here, accuracy metrics can be used to evaluate the generated predictions because each of these datasets reduces the original problem to a choice problem on a closed set. Specifically, DDXPlus uses a predefined list of diseases from which the model must select a disease based on the provided patient background. In SEER, treatment recommendations are divided into eight high-level categories, while in MIMIC4ED, the final clinical outcome decision is always binary (True or False).
Overall, open source LLMs perform worse than closed source LLMs on these tasks, and in some cases they fail to provide meaningful predictions. For example, Llama 3 performs poorly in predicting Critical Triage. For the DDXPlus diagnostic task, InternLM 2 and Llama 3 performed slightly better, with an accuracy of 32. However, closed-source models such as GPT-4 and Claude-3.5 show significantly better performance. For example, Claude-3.5 can achieve an accuracy of 90 on SEER, while GPT-4 has a higher diagnostic accuracy on DDXPlus, with a score of 52, highlighting the huge gap between open source and closed source LLM.
Despite these results, these scores are still not reliable enough for clinical use. In contrast, MMedIns-Llama 3 showed superior accuracy in clinical decision support tasks, such as 98 on SEER, 95 on DDXPlus, and an average accuracy of 86.67 on clinical outcome prediction tasks (Hospitalization, 72h average of ED Revisit, and Critical Triage scores).
Text Classification: Table 8 also shows the evaluation on the HoC multi-label classification task and reports Macro-precision, Macro-recall and Macro-F1 Scores . For this type of task, all candidate labels are input into the language model in the form of a list, and the model is asked to select its corresponding answer, with multiple choices allowed. Accuracy metrics are then calculated based on the final selection output of the model.
GPT-4 and Claude-3.5 perform well on this task. GPT-4’s Macro-F1 score is 60.38, and Claude-3.5 is even better, achieving 63.32. Both models show strong recall capabilities, especially Claude-3.5, which has a Macro-Recall of 80.96. Mistral performed moderately well with a Macro-F1 score of 40.8, balancing precision and recall.
In contrast, the overall performance of Llama 3 and InternLM 2 is poor, with Macro-F1 scores of 36.18 and 32.72 respectively. These models (especially InternLM 2) exhibit high recall but poor precision, resulting in low Macro-F1 scores.
MEDITRON ranks lowest in this task with a Macro-F1 score of 26.21. MMedIns-Llama 3 significantly outperforms all other models, achieving the highest scores in all metrics, with Macro-precision of 91.29, Macro-recall of 85.57, and Macro-F1 score of 87.37. These results highlight the ability of MMedIns-Llama 3 to accurately classify text, making it the most effective model for this type of complex task.
Table 8: Results on four categories of tasks: treatment planning (SEER), diagnosis (DDXPlus), clinical outcome prediction (MIMIC4ED), and text classification (HoC Classification). The results of the first three tasks are based on accuracy, and the text classification results are based on precision, recall, and F1 score.
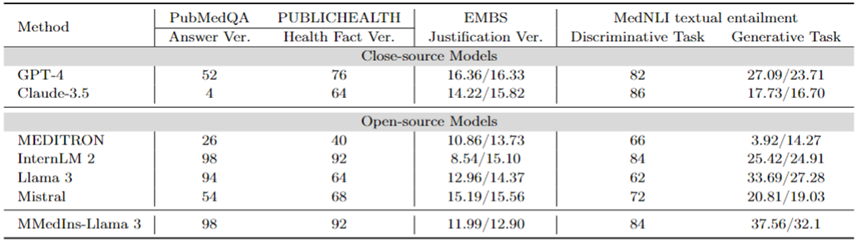
Fact Correction: Table 9 shows the model evaluation results on the fact verification task. For PubMedQA answer validation and HealthFact validation, LLM needs to select an answer from the provided candidate list, so accuracy is used as the evaluation metric.
In contrast, due to EBMS justification validation, the task involves generating free-form text, using BLEU-1 and ROUGE-1 scores to evaluate performance. InternLM 2 achieved the highest accuracy in PubMedQA answer validation and HealthFact validation, with scores of 98 and 92 respectively.
In the EBMS benchmark, GPT-4 shows the strongest performance, with BLEU-1/ROUGE-1 scores of 16.36/16.33 respectively. Claude-3.5 is a close second with scores of 14.22/15.82, but it performs poorly on PubMedQA answer validation.
Llama 3 has an accuracy of 94 and 64 on PubMedQA and HealthFact Verification respectively, and a BLEU-1/ROUGE-1 score of 12.96/14.37. MMedIns-Llama 3 continues to outperform existing models, achieving the highest accuracy score on the PubMedQA answer verification task along with InternLM 2, while in EMBS, MMedIns-Llama 3 achieves 11.99/12.90 in BLEU-1 and ROUGE-1. The results are slightly behind GPT-4.
Medical text implication (NLI): Table 9 also shows the evaluation results on medical text implication (NLI), mainly MedNLI. There are two testing methods, one is a discriminative task (selecting the correct answer from a candidate list), measured by accuracy, and the other is a generation task (generating free-form text answers), measured by the BLEU/ROUGE metric.
InternLM 2 has the highest score among open source LLMs with a score of 84. For closed-source LLM, both GPT-4 and Claude-3.5 show relatively high scores with accuracies of 82 and 86 respectively. In the generation task, Llama 3 has the highest consistency with ground truth, with BLEU and ROUGE scores of 33.69/27.28. Mistral and Llama 3 performed at an average level. GPT-4 follows closely behind with scores of 27.09/23.71, while Claude-3.5 does not perform well in the generation task.
MMedIns-Llama 3 has the highest accuracy in the discrimination task with a score of 84, but is slightly behind Claude-3.5. MMedIns-Llama 3 also performs well in the generation task, with a BLEU/ROUGE score of 37.56/32.17, which is significantly better than other models.
Table 9: Quantitative results on fact verification and text implication tasks. The results are measured by accuracy (ACC) and BLEU/ROUGE; "Ver." in the table is the abbreviation of "verification".
In general, the research team evaluated six mainstream models in various task dimensions. The research results show that the current mainstream LLMs are still quite fragile when dealing with clinical tasks. , which will produce serious performance deficiencies in diverse and complex clinical scenarios.
At the same time, the experimental results also show that by adding more clinical task texts to the instruction data set to strengthen the match between LLM and actual clinical application, the performance of LLM can be greatly enhanced.
Data collection method and training process
This section will introduce the training process in detail, as shown in Figure 3b. The specific method is the same as the previous work MMedLM and PMC-LLaMA, both of which can inject corresponding medical knowledge into the model through further autoregressive training on medical-related corpora, thereby making them perform better in different downstream tasks.
Specifically, the research team started with a multilingual LLM base model (MMed-Llama 3) and further trained it using instruction fine-tuning data from MedS-Ins.
The data for instruction fine-tuning mainly involves two aspects:
Medically filtered natural instruction data: First, from Super-Natural Instructions, the largest instruction data set in the natural field Filter out medical-related tasks. Since Super-NaturalInstructions focuses more on different natural language processing tasks in general fields, the classification granularity of the medical field is relatively coarse.
First, all instructions in the "Healthcare" and "Medicine" categories were extracted, and then more detailed domain labels were manually added to them while the task categories remained unchanged. In addition, many general domain organized instruction fine-tuning datasets also cover some medical-related data, such as LIMA and ShareGPT.
In order to filter out the medical part of these data, the research team used InsTag to conduct coarse-grained classification of the domain of each instruction. Specifically, InsTag is an LLM designed to tag different instruction samples. Given an instruction query, it will analyze which domain and task the instruction belongs to, and based on this, filter out samples labeled as healthcare, medical, or biomedical.
Finally, by filtering the instruction data set in the general domain, 37 tasks were collected, with a total of 75373 samples.
Tips for constructing existing BioNLP datasets: There are many excellent datasets for text analysis in clinical scenarios among existing datasets. However, since most datasets are collected for different purposes, they cannot be directly used to train large language models. However, these existing medical NLP tasks can be incorporated into instruction adaptation by converting them into a format that can be used to train generative models.
Specifically, the research team took MIMIC-IV-Note as an example. MIMIC-IV-Note provides high-quality structured reports with both findings and conclusions, and the generation of findings to conclusions is considered a classic clinical text summary task. First manually write prompts to define the task, for example: "Given the detailed results of ultrasound imaging diagnosis, summarize the findings in a few words." Considering the diversity needs of instruction adjustment, the research team asked 5 people to independently use 3 different prompts to describe a certain task.
This resulted in 15 free text prompts per task, ensuring similar semantics but as diverse wording and formatting as possible. Then, inspired by Self-Instruct, these manually written instructions are used as seed instructions and GPT-4 is asked to rewrite them according to them to obtain more diverse instructions.
Through the above process, an additional 85 tasks were prompted into a unified free question and answer format, and combined with the filtered data, a total of 13.5 million high-quality samples were obtained, covering 122 tasks, called MedS- Ins, and through instruction fine-tuning, a new 8B size medical LLM was trained, and the results showed that this method significantly improved the performance of clinical tasks.
In instruction fine-tuning, the research team focused on two instruction forms:
Zero-sample prompt: Here, the instruction of the task contains some semantic task descriptions as prompts , thus requiring the model to directly answer the question based on its internal model knowledge. In the collected MedS-Ins, the "definition" content of each task can naturally be used as zero-point instruction input. As a variety of different medical task definitions are covered, the model is expected to learn semantic understanding of various task descriptions.
Few-shot tip: Here, the instructions contain a small number of examples that allow the model to learn the approximate requirements of the task from the context. Such instructions can be obtained by simply randomly sampling other cases from the training set for the same task and organizing them using the following simple template:
Fall1: Eingabe: {CASE1_INPUT}, Ausgabe: {CASE1_OUTPUT} ... FallN: Eingabe: {CASEN_INPUT}, Ausgabe: {CASEN_OUTPUT} {INSTRUCTION} Bitte lernen Sie aus den wenigen Schussfällen Sehen Sie, welche Inhalte Sie ausgeben müssen. Eingabe: {INPUT}
Diskussion
Insgesamt liefert dieses Papier mehrere wichtige Beiträge:
Umfassender Bewertungs-Benchmark – MedS-Bench
Die Entwicklung des medizinischen LLM basiert stark auf Multiple-Choice-Frage-Antwort-Benchmark-Tests (MCQA). Dieser enge Bewertungsrahmen ignoriert jedoch die wahren Fähigkeiten von LLM in einer Vielzahl komplexer klinischer Szenarien.
Daher stellt das Forschungsteam in dieser Arbeit MedS-Bench vor, einen umfassenden Benchmark zur Bewertung der Leistung von Closed-Source- und Open-Source-LLMs bei einer Vielzahl klinischer Aufgaben, einschließlich solcher, die Daten erfordern Modelle Die Aufgabe, Fakten aus einem vorgefertigten Korpus abzurufen oder Schlussfolgerungen aus einem bestimmten Kontext zu ziehen.
Die Ergebnisse zeigen, dass bestehende LLMs zwar bei MCQA-Benchmarks gut abschneiden, es ihnen jedoch schwerfällt, sich an die klinische Praxis anzupassen, insbesondere bei Aufgaben wie Behandlungsempfehlungen und -erklärungen. Dieses Ergebnis unterstreicht die Notwendigkeit einer Weiterentwicklung medizinischer Großsprachmodelle, die an ein breiteres Spektrum klinischer und medizinischer Szenarien angepasst sind.
Umfassender Datensatz zur Anweisungsanpassung – MedS-Ins
Das Forschungsteam bezog umfassend Daten aus dem vorhandenen BioNLP-Datensatz und konvertierte diese Proben in ein einheitliches Format, At Gleichzeitig wurde eine halbautomatische Aufforderungsstrategie verwendet, um MedS-Ins, einen neuen Datensatz zur Anpassung medizinischer Verordnungen, zu erstellen und zu entwickeln. Frühere Arbeiten zur Feinabstimmung von Datensätzen für den Unterricht konzentrierten sich hauptsächlich auf die Erstellung von Frage-Antwort-Paaren aus täglichen Gesprächen, Prüfungen oder wissenschaftlichen Arbeiten und ignorierten dabei häufig Texte, die aus der tatsächlichen klinischen Praxis stammen.
Im Gegensatz dazu integriert MedS-Ins ein breiteres Spektrum medizinischer Textressourcen, darunter 5 Haupttextbereiche und 19 Aufgabenkategorien. Diese systematische Analyse der Datenzusammensetzung erleichtert Benutzern das Verständnis der klinischen Anwendungsgrenzen von LLM.
Medizinisches großes Sprachmodell – MMedIns-Llama 3
In Bezug auf das Modell hat das Forschungsteam dies bewiesen, indem es Schulungen zur Feinabstimmung der Anweisungen an MedS-Ins durchgeführt hat Es kann die Ausrichtung von Open-Source-LLM im medizinischen Bereich an den klinischen Anforderungen erheblich verbessern.
Es muss betont werden, dass das endgültige Modell MMedIns-Llama 3 eher ein „Proof-of-Concept“-Modell ist. Es verwendet eine mittlere Parameterskala von 8B. Das endgültige Modell zeigt ein tiefes Verständnis für verschiedene klinische Aufgaben und kann sich durch keine oder eine kleine Anzahl von Anweisungen flexibel an eine Vielzahl medizinischer Szenarien anpassen, ohne dass eine weitere aufgabenspezifische Schulung erforderlich ist.
Die Ergebnisse zeigen, dass MMedIns-Llama 3 bestehende LLMs, einschließlich GPT-4, Claude-3.5 usw., bei bestimmten klinischen Aufgabentypen übertrifft.
Bestehende Einschränkungen
Hier möchte das Forschungsteam auch auf die Einschränkungen dieses Artikels und mögliche zukünftige Verbesserungen hinweisen.
Erstens deckt MedS-Bench derzeit nur 11 klinische Aufgaben ab, was nicht die Komplexität aller klinischen Szenarien vollständig abdeckt. Darüber hinaus wurden zwar sechs gängige LLMs evaluiert, einige der neuesten LLMs fehlten jedoch noch in der Analyse. Um diese Einschränkungen zu beseitigen, plant das Forschungsteam, gleichzeitig mit der Veröffentlichung dieses Artikels ein Medical LLM Leaderboard zu veröffentlichen, mit dem Ziel, mehr Forscher zu ermutigen, den umfassenden Bewertungsmaßstab für medizinisches LLM kontinuierlich zu erweitern und zu verbessern. Durch die Einbeziehung weiterer Aufgabenkategorien aus unterschiedlichen Textquellen in den Bewertungsprozess soll ein tieferes Verständnis für die Entwicklung und Einsatzgrenzen von LLMs in der Medizin gewonnen werden.
Zweitens deckt MedS-Ins zwar mittlerweile ein breites Spektrum medizinischer Aufgaben ab, ist aber immer noch unvollständig und es fehlen einige praktische medizinische Szenarien. Um dieses Problem zu lösen, stellte das Forschungsteam alle gesammelten Daten und Ressourcen als Open-Source-Lösung auf GitHub zur Verfügung. Ich hoffe aufrichtig, dass mehr Kliniker oder Forscher zusammenarbeiten können, um diesen Datensatz zur Anpassung von Anweisungen zu pflegen und zu erweitern, ähnlich wie Super-NaturalInstructions im allgemeinen Bereich. Das Forschungsteam hat detaillierte Anweisungen zum Hochladen auf der GitHub-Seite bereitgestellt und wird sich schriftlich bei allen Mitwirkenden bedanken, die an der Aktualisierung des Datensatzes im Rahmen der iterativen Aktualisierung des Papiers teilgenommen haben.
Drittens plant das Forschungsteam, MedS-Bench und MedS-Ins um weitere Sprachen zu erweitern, um die Entwicklung leistungsfähigerer mehrsprachiger medizinischer LLMs zu unterstützen. Derzeit sind diese Ressourcen hauptsächlich auf Englisch ausgerichtet, obwohl einige mehrsprachige Aufgaben in MedS-Bench und MedS-Ins enthalten sind. Die Ausweitung auf ein breiteres Spektrum von Sprachen wäre eine vielversprechende zukünftige Richtung, um sicherzustellen, dass die jüngsten Fortschritte in der medizinischen KI einer größeren und vielfältigeren Region gleichermaßen zugute kommen können.
Schließlich hat das Forschungsteam den gesamten Code, die Daten und die Bewertungsprozesse als Open Source bereitgestellt. Es besteht die Hoffnung, dass diese Arbeit dazu führen wird, dass sich die Entwicklung medizinischer LLMs stärker auf die Integration dieser leistungsstarken Sprachmodelle in reale klinische Anwendungen konzentriert.
The above is the detailed content of Towards a 'all-rounder' medical model, the Shanghai Jiao Tong University team releases large-scale instruction fine-tuning data, open source models and comprehensive benchmark testing. For more information, please follow other related articles on the PHP Chinese website!

Hot Article

Hot tools Tags

Hot Article

Hot Article Tags

Notepad++7.3.1
Easy-to-use and free code editor

SublimeText3 Chinese version
Chinese version, very easy to use

Zend Studio 13.0.1
Powerful PHP integrated development environment

Dreamweaver CS6
Visual web development tools

SublimeText3 Mac version
God-level code editing software (SublimeText3)

Hot Topics
 Breaking through the boundaries of traditional defect detection, 'Defect Spectrum' achieves ultra-high-precision and rich semantic industrial defect detection for the first time.
Jul 26, 2024 pm 05:38 PM
Breaking through the boundaries of traditional defect detection, 'Defect Spectrum' achieves ultra-high-precision and rich semantic industrial defect detection for the first time.
Jul 26, 2024 pm 05:38 PM
Breaking through the boundaries of traditional defect detection, 'Defect Spectrum' achieves ultra-high-precision and rich semantic industrial defect detection for the first time.
 NVIDIA dialogue model ChatQA has evolved to version 2.0, with the context length mentioned at 128K
Jul 26, 2024 am 08:40 AM
NVIDIA dialogue model ChatQA has evolved to version 2.0, with the context length mentioned at 128K
Jul 26, 2024 am 08:40 AM
NVIDIA dialogue model ChatQA has evolved to version 2.0, with the context length mentioned at 128K
 Google AI won the IMO Mathematical Olympiad silver medal, the mathematical reasoning model AlphaProof was launched, and reinforcement learning is so back
Jul 26, 2024 pm 02:40 PM
Google AI won the IMO Mathematical Olympiad silver medal, the mathematical reasoning model AlphaProof was launched, and reinforcement learning is so back
Jul 26, 2024 pm 02:40 PM
Google AI won the IMO Mathematical Olympiad silver medal, the mathematical reasoning model AlphaProof was launched, and reinforcement learning is so back
 To provide a new scientific and complex question answering benchmark and evaluation system for large models, UNSW, Argonne, University of Chicago and other institutions jointly launched the SciQAG framework
Jul 25, 2024 am 06:42 AM
To provide a new scientific and complex question answering benchmark and evaluation system for large models, UNSW, Argonne, University of Chicago and other institutions jointly launched the SciQAG framework
Jul 25, 2024 am 06:42 AM
To provide a new scientific and complex question answering benchmark and evaluation system for large models, UNSW, Argonne, University of Chicago and other institutions jointly launched the SciQAG framework
 Nature's point of view: The testing of artificial intelligence in medicine is in chaos. What should be done?
Aug 22, 2024 pm 04:37 PM
Nature's point of view: The testing of artificial intelligence in medicine is in chaos. What should be done?
Aug 22, 2024 pm 04:37 PM
Nature's point of view: The testing of artificial intelligence in medicine is in chaos. What should be done?
 Training with millions of crystal data to solve the crystallographic phase problem, the deep learning method PhAI is published in Science
Aug 08, 2024 pm 09:22 PM
Training with millions of crystal data to solve the crystallographic phase problem, the deep learning method PhAI is published in Science
Aug 08, 2024 pm 09:22 PM
Training with millions of crystal data to solve the crystallographic phase problem, the deep learning method PhAI is published in Science
 SOTA performance, Xiamen multi-modal protein-ligand affinity prediction AI method, combines molecular surface information for the first time
Jul 17, 2024 pm 06:37 PM
SOTA performance, Xiamen multi-modal protein-ligand affinity prediction AI method, combines molecular surface information for the first time
Jul 17, 2024 pm 06:37 PM
SOTA performance, Xiamen multi-modal protein-ligand affinity prediction AI method, combines molecular surface information for the first time
 Covering text, positioning and segmentation tasks, Zhiyuan and Hong Kong Chinese jointly proposed the first multi-functional 3D medical multi-modal large model
Jun 22, 2024 am 07:16 AM
Covering text, positioning and segmentation tasks, Zhiyuan and Hong Kong Chinese jointly proposed the first multi-functional 3D medical multi-modal large model
Jun 22, 2024 am 07:16 AM
Covering text, positioning and segmentation tasks, Zhiyuan and Hong Kong Chinese jointly proposed the first multi-functional 3D medical multi-modal large model






