
Éditeur | Violet
Les interactions entre protéines, médicaments et autres biomolécules jouent un rôle essentiel dans divers processus biologiques. Comprendre ces interactions est essentiel pour déchiffrer les mécanismes moléculaires qui sous-tendent les processus biologiques et développer de nouvelles stratégies thérapeutiques. Les protéines font partie des molécules les plus importantes dans les cellules et remplissent diverses fonctions au sein des cellules. Les médicaments régulent souvent les processus physiologiques en interagissant avec des protéines spécifiques. Ces interactions peuvent favoriser ou inhiber des voies de signalisation moléculaires spécifiques. Les méthodes de calcul multi-échelles actuelles reposent souvent trop sur une seule échelle et sous-ajustent les autres échelles. Cela peut être lié au tropisme multi-échelle inégal et à la cupidité inhérente à l’apprentissage multi-échelle.
Afin d'atténuer le déséquilibre d'optimisation, des chercheurs de l'Université Sun Yat-sen et de l'Université Jiao Tong de Shanghai ont proposé un cadre d'apprentissage de représentation multi-échelle MUSE basé sur la maximisation des attentes variables, qui peut intégrer efficacement des informations multi-échelles pour l'apprentissage. Cette stratégie fusionne efficacement les informations multi-échelles entre la structure atomique et les échelles du réseau moléculaire grâce à une supervision mutuelle et une optimisation itérative. Cette approche permet un meilleur transfert d’informations et un meilleur apprentissage. Cette stratégie fusionne efficacement les informations multi-échelles entre la structure atomique et les échelles du réseau moléculaire grâce à une supervision mutuelle et une optimisation itérative.
MUSE+ surpasse non seulement les modèles de pointe actuels sur les tâches d'interaction moléculaire (protéine-protéine, médicament-protéine et médicament), mais surpasse également les modèles de pointe actuels sur les prédictions d'interface protéique. à l’échelle de la structure atomique. Plus important encore, le cadre d’apprentissage multi-échelle peut être étendu à la découverte informatique de médicaments à d’autres échelles.
L'étude, intitulée "
Un cadre variationnel d'attente-maximisation pour un apprentissage multi-échelle équilibré des interactions protéiques et médicamenteuses", a été publiée dans "Nature Communications" le 25 mai.
 Lien papier :
Lien papier :
Prédire ces interactions uniquement à partir de la structure est l'un des défis les plus importants en biologie structurale. Les méthodes informatiques actuelles prédisent principalement les interactions basées sur des réseaux moléculaires ou des informations structurelles et ne les intègrent pas dans un cadre multi-échelle unifié.
Alors que certaines méthodes d'apprentissage multi-vues s'efforcent de fusionner des informations multi-échelles, une manière intuitive d'apprendre des représentations multi-échelles consiste à combiner des graphiques moléculaires avec des réseaux d'interaction et à les optimiser conjointement. Cependant, en raison du déséquilibre et de la gourmandise inhérente à l’apprentissage multi-échelle, ces modèles s’appuient souvent fortement sur une seule échelle. L’incapacité à utiliser efficacement les informations liées à toutes les échelles et à la généralisation est faible.
De plus, un cadre multi-échelle efficace doit non seulement capturer des informations riches à différentes échelles, mais doit également bien préserver les relations sous-jacentes entre elles.
MUSE pour l'apprentissage d'informations multi-échelles sur les protéines et les médicamentsMUSE est une méthode d'apprentissage multi-échelle qui combine la modélisation de la structure moléculaire et l'apprentissage de réseaux d'interactions protéine-médicament via un cadre de maximisation des attentes variationnelles (EM). Le framework EM optimise deux modules, à savoir l'étape d'attente (étape E) et l'étape de maximisation (étape M), dans un processus alterné de plusieurs itérations.
Pendant l'étape E, MUSE utilise les informations structurelles de chaque biomolécule pour apprendre des représentations structurelles efficaces pour l'entraînement à l'étape M en utilisant des interactions connues et des échantillons de renforcement. Il prend comme entrée des paires de protéines et de médicaments et leurs informations structurelles au niveau atomique, augmentées par les interactions prédites en M-étapes. M-step prend en entrée les interactions prédites du réseau d'interactions au niveau moléculaire, des incorporations structurelles et de l'étape E et génère les interactions prédites. L'optimisation itérative entre E-step et M-step garantit une capture interactive de la structure moléculaire et des informations de réseau avec des taux d'apprentissage différents aux deux échelles.
La supervision mutuelle garantit que chaque modèle réduit apprend de manière appropriée, permettant l'utilisation d'informations efficaces à différentes échelles. Ce cadre sera démontré à travers de multiples échelles d'interactions entre protéines et médicaments. Il est analysé que MUSE atténue les caractéristiques déséquilibrées de l'apprentissage multi-échelles et intègre efficacement les informations hiérarchiques et complémentaires provenant de différentes échelles.
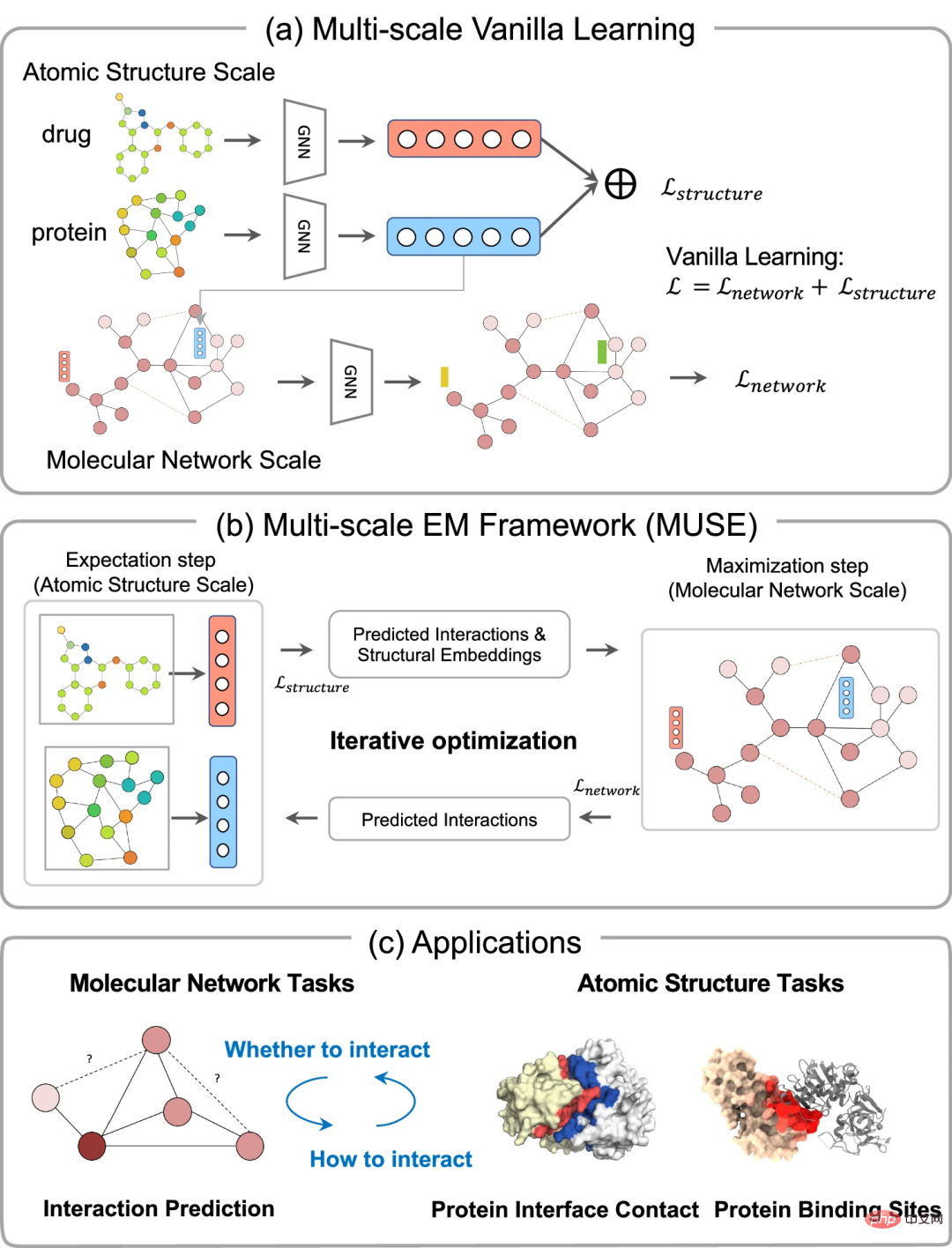
Utilise les informations sur la structure atomique pour améliorer les prédictions à l'échelle du réseau moléculaire
Pour évaluer leur approche, premièrement, la les chercheurs ont utilisé MUSE pour intégrer les informations structurelles des atomes afin d’améliorer les prédictions à l’échelle du réseau moléculaire. MUSE atteint des performances de pointe sur trois tâches de prédiction d’interactions multi-échelles : l’interaction protéine-protéine (PPI), l’interaction médicament-protéine (DPI) et l’interaction médicament-médicament (DDI).
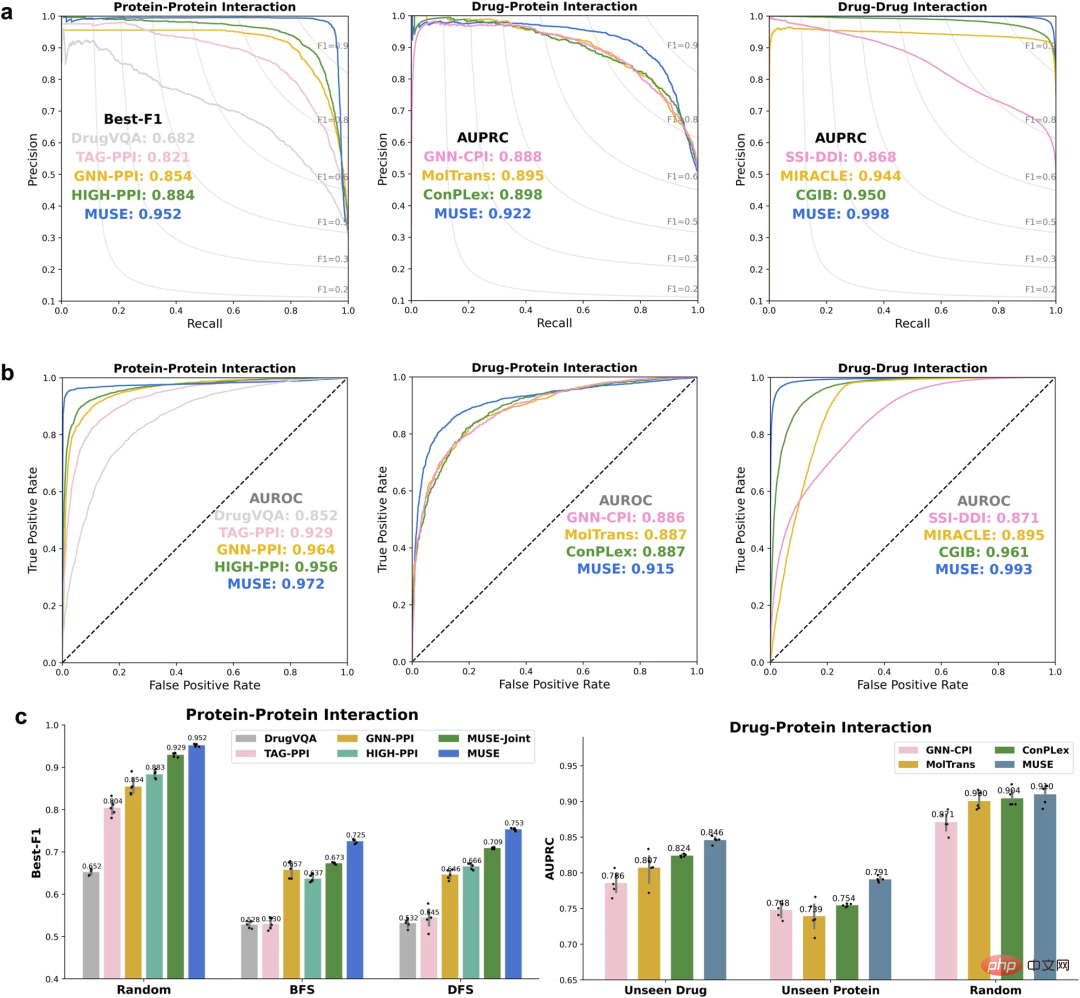
Amélioration des prédictions à l'échelle de la structure atomique à partir de l'échelle du réseau moléculaire
En plus d'utiliser les informations sur la structure atomique pour améliorer les prédictions à l'échelle du réseau moléculaire, les chercheurs ont étudié plus en détail la capacité de MUSE à apprendre et à prédire les structures à l'échelle du réseau moléculaire. échelle de la structure atomique Capacité à caractériser, notamment à prédire les contacts interfaciaux et les sites de liaison associés aux IPP.
Pour évaluer les prédictions de contacts interchaînes de protéines, MUSE a été comparé aux méthodes de pointe du benchmark DIPS-Plus. MUSE surpasse systématiquement toutes les autres méthodes, validant son efficacité et son adaptabilité dans la prédiction de la structure atomique.
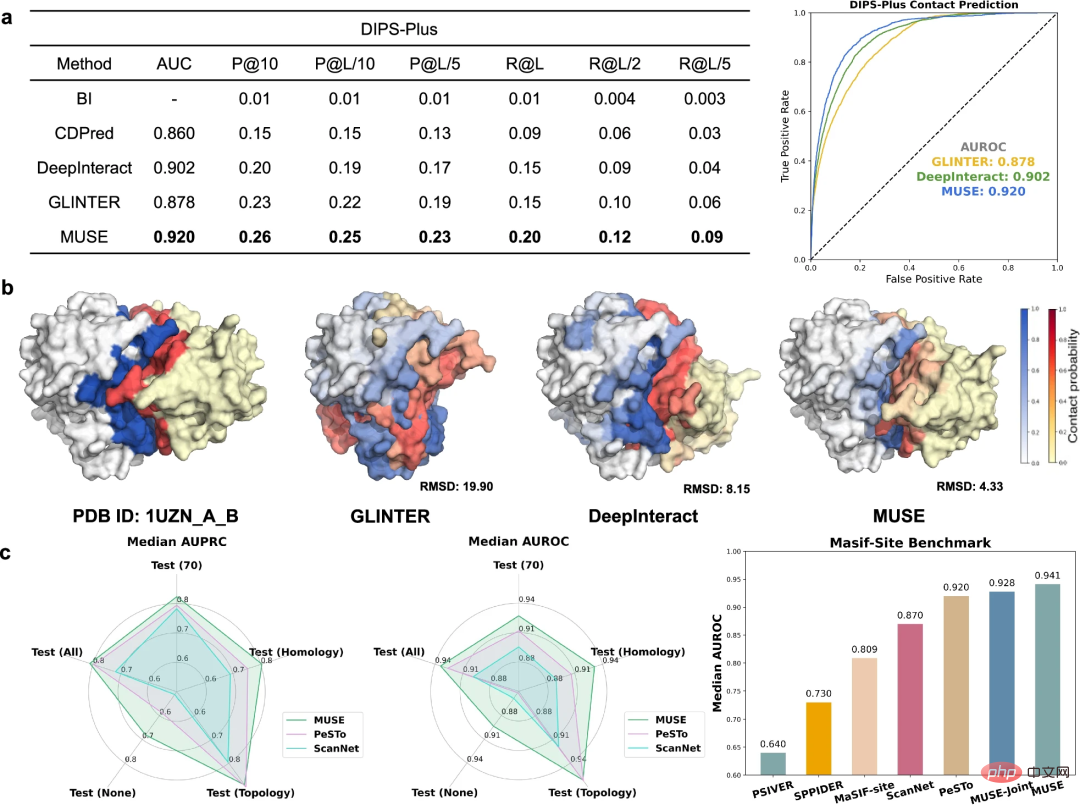
MUSE a été évalué plus en détail pour prédire si les résidus sont directement impliqués dans les interactions protéine-protéine. Les résultats démontrent que l’apprentissage à l’échelle du réseau moléculaire dans MUSE peut fournir des informations précieuses sur les prédictions à l’échelle de la structure atomique.
Atténuer les caractéristiques de déséquilibre de l'apprentissage multi-échelle grâce à l'optimisation itérative
Afin d'explorer pourquoi MUSE peut atteindre des performances supérieures dans la représentation multi-échelle, les chercheurs ont analysé la capacité d'apprentissage de MUSE en fonction des caractéristiques de déséquilibre de l'apprentissage multi-échelle. apprentissage.
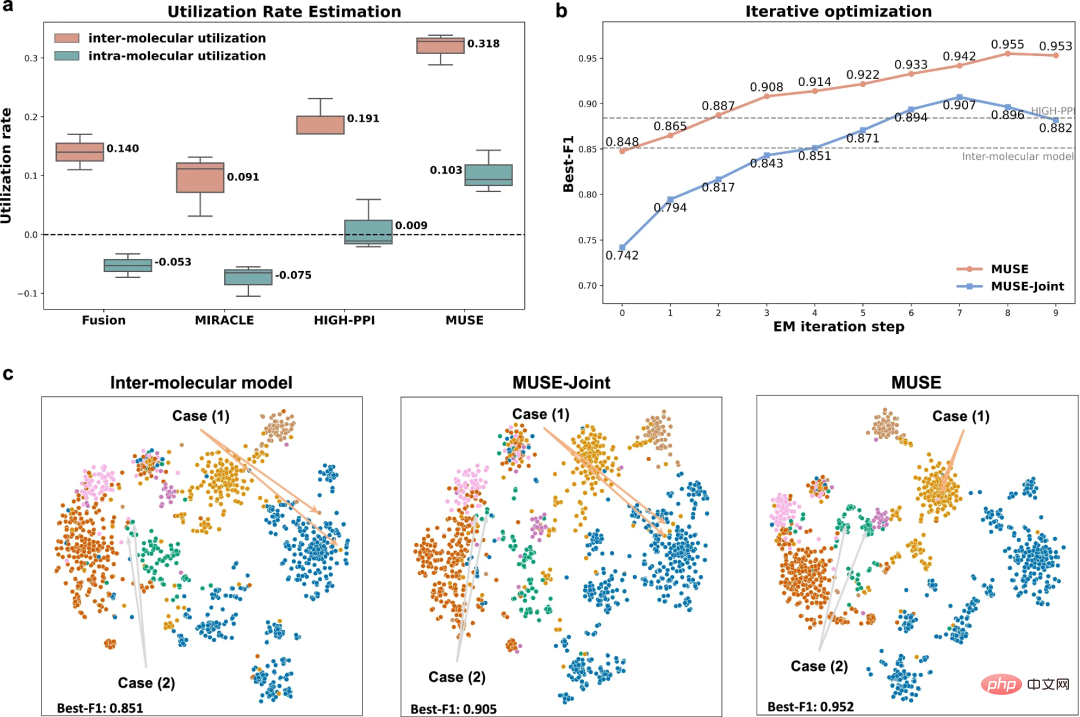
Les résultats montrent que MUSE atténue efficacement les caractéristiques de déséquilibre et l'apprentissage gourmand dans l'apprentissage multi-échelle, garantissant l'utilisation complète des informations à différentes échelles pendant le processus de formation. En outre, des expériences d'analyse du taux d'utilisation ont permis aux chercheurs de comprendre spécifiquement ce que le modèle avait appris et ont démontré que l'utilisation de MUSE pour équilibrer l'apprentissage du modèle à différentes échelles peut améliorer les capacités de généralisation.
Visualisation et interprétation de la représentation multi-échelle apprise
Pour mieux comprendre la représentation multi-échelle apprise, les chercheurs ont étudié la représentation multi-échelle apprise par MUSE sous différents angles, y compris (1) Capture MUSE La capacité de l'atome informations de structure (c'est-à-dire, motifs structurels et intégrations) impliquées dans le PPI, et (2) supervision mutuelle entre la structure atomique apprise et les représentations de réseaux moléculaires.
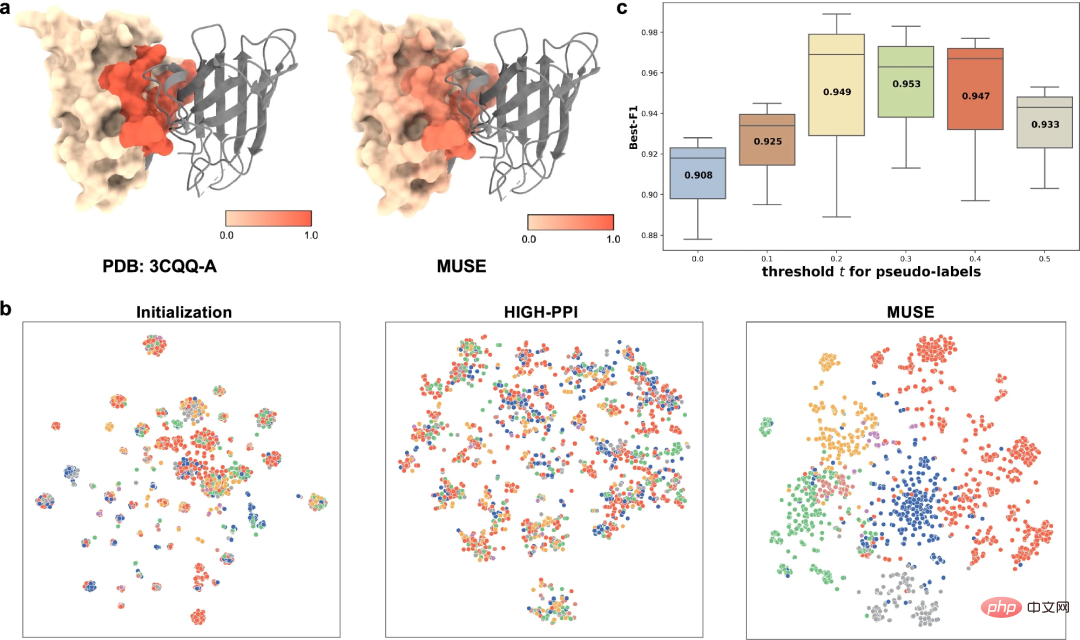
À titre d'exemple de prédiction de site de liaison (ID PDB : 3CQQ-A), MUSE peut identifier avec précision les résidus appartenant au site de liaison avec une précision de 97,7 %. Cela suggère que la supervision mutuelle dans MUSE aide les modèles à l’échelle de la structure atomique à apprendre les sous-structures clés pertinentes pour les interactions.
Enfin, les chercheurs ont également mené des études d'ablation pour étudier l'impact des pseudo-marqueurs prédits à l'échelle de la structure atomique sur l'échelle du réseau moléculaire.
Bien que MUSE démontre des performances de pointe dans les benchmarks, il est encore possible d'améliorer sa capacité à gérer des tâches en aval multi-échelles bruyantes et incomplètes. Ceci peut être réalisé en combinant les connaissances antérieures grâce à des graphiques de connaissances et des techniques d'IA explicables. D’un autre côté, ce cadre conceptuel multi-échelle peut également être étendu à la découverte informatique de médicaments à d’autres échelles.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
 Introduction au rôle des serveurs cloud
Introduction au rôle des serveurs cloud
 Introduction aux indicateurs de performances du disque dur
Introduction aux indicateurs de performances du disque dur
 Comment mettre à jour le pilote de la carte graphique
Comment mettre à jour le pilote de la carte graphique
 Comment supprimer un index dans MySQL
Comment supprimer un index dans MySQL
 ébrécher
ébrécher
 Comment regarder les enregistrements de diffusion en direct sur Douyin
Comment regarder les enregistrements de diffusion en direct sur Douyin
 solution d'exception d'instanciationexception
solution d'exception d'instanciationexception
 Comment récupérer des fichiers supprimés définitivement sur un ordinateur
Comment récupérer des fichiers supprimés définitivement sur un ordinateur