
Editeur : KXIntégration de la transcriptomique spatiale et des données multi-omiques
La transcriptomique spatiale est un développement majeur après la transcriptomique unicellulaire, rendant l'intégration de données multi-omique cruciale.
SpatialGlue : modèle de réseau neuronal graphique avec mécanisme d'attention double
Une équipe de recherche de l'Agence de Singapour pour la science, la technologie et la recherche (A*STAR), BGI et l'hôpital Renji affilié à l'école de médecine de l'université Jiao Tong de Shanghai a proposé un méthode appelée Il s'agit du modèle de réseau neuronal graphique de SpatialGlue, qui intègre des données multi-omiques via un double mécanisme d'attention pour révéler la structure histologiquement pertinente des échantillons de tissus d'une manière spatialement consciente.
Avantages de SpatialGlue
SpatialGlue est capable de combiner plusieurs modalités de données avec leurs contextes spatiaux respectifs. Par rapport à d'autres méthodes, SpatialGlue présente les avantages suivants :
- Capturez plus de détails anatomiques.
- Résolvez les domaines spatiaux (tels que le cortex cérébral) avec plus de précision.
- Identifiez les types de cellules situés dans différentes régions (comme les sous-populations de macrophages de la rate).
Un outil puissant pour l'analyse de l'omique spatiale
Cette étude met en évidence la puissante capacité de l'omique spatiale multimodale à analyser la complexité biologique.
Recherche connexe
La recherche connexe était intitulée "Déchiffrer les domaines spatiaux à partir de la multi-omique spatiale avec SpatialGlue" et a été publiée dans "Nature Methods" le 21 juin.

L'intégration des données multi-omiques spatiales est confrontée à des défis. Actuellement, la technologie spatiale est étendue aux multi-omiques spatiales, c'est-à-dire que différents omiques sont analysés simultanément sur une seule section de tissu. Ces technologies peuvent être largement divisées en deux catégories : basées sur le séquençage et basées sur l’imagerie. Il offre la possibilité de comprendre en profondeur les propriétés des cellules et des tissus émergents.
Pour exploiter pleinement les données spatiales multi-omiques afin de construire une image cohérente du tissu étudié, une intégration spatialement consciente de modalités de données hétérogènes est nécessaire. L'intégration de données multi-omiques est confrontée à des défis importants, car le nombre de caractéristiques des différentes modalités peut varier considérablement et avoir des distributions statistiques différentes. Ce défi est exacerbé lors de la combinaison d’informations spatiales avec le nombre de caractéristiques au sein de chaque modalité de données.
Actuellement, il n’existe aucun outil spécifiquement conçu pour les multi-omiques spatiales acquises à partir de la même coupe de tissu. Par conséquent, des outils spécifiquement adaptés aux données spatiales multi-omiques sont nécessaires pour relever le défi de l’intégration des données spatiales multi-omiques pour l’analyse en aval. En particulier, de nouvelles méthodes permettant l’intégration cross-omique de la perception spatiale sont nécessaires.
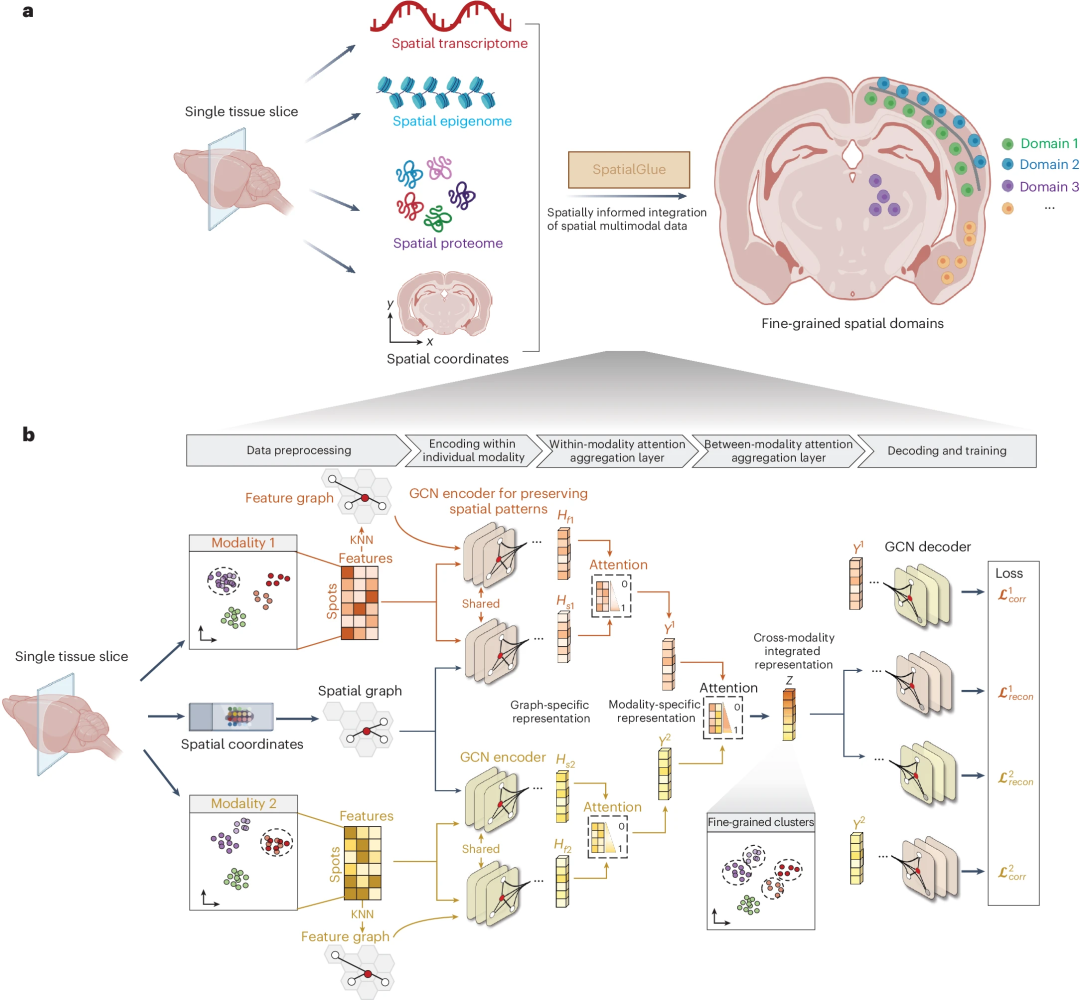
Structure du modèle SpatialGlue SpatialGlue interprète le domaine spatial des échantillons de tissus avec une résolution plus élevée en combinant efficacement les données modales multi-omiques avec des informations spatiales. SpatialGlue est un modèle d'apprentissage profond basé sur les réseaux de neurones graphiques (GNN).
SpatialGlue utilise d'abord l'algorithme k-plus proche voisin (KNN) pour créer un graphe de voisinage spatial à l'aide de coordonnées spatiales et un graphe de voisinage de caractéristiques à l'aide de données d'expression normalisées pour chaque modalité omics.
Ensuite, pour chaque modalité, l'encodeur GNN prend la représentation normalisée et le graphe voisin pour apprendre deux représentations spécifiques au graphe en agrégeant de manière itérative les représentations des voisins. Pour capturer l'importance des différents graphiques, une couche d'agrégation d'attention intra-modale est conçue pour intégrer de manière adaptative des représentations spécifiques aux graphiques et obtenir des représentations spécifiques aux modalités.
Enfin, pour préserver l'importance des différentes modalités, SpatialGlue utilise une couche d'agrégation d'attention intermodale pour intégrer de manière adaptative des représentations spécifiques à une modalité et produire une représentation finale intégrée au blob.

Illustration : Modèle de double attention profonde interprétable pour l'analyse de données spatiales multi-omiques. (Source : article) Pour évaluer l'efficacité du modèle SpatialGlue proposé, les chercheurs ont d'abord vérifié l'importance de l'attention et d'autres composants grâce à une série d'études d'ablation utilisant des données simulées. Par la suite, la sensibilité de SpatialGlue au nombre de voisins de l'entrée, à la dimension de l'analyse en composantes principales (ACP) et au nombre de couches GNN sont caractérisées.
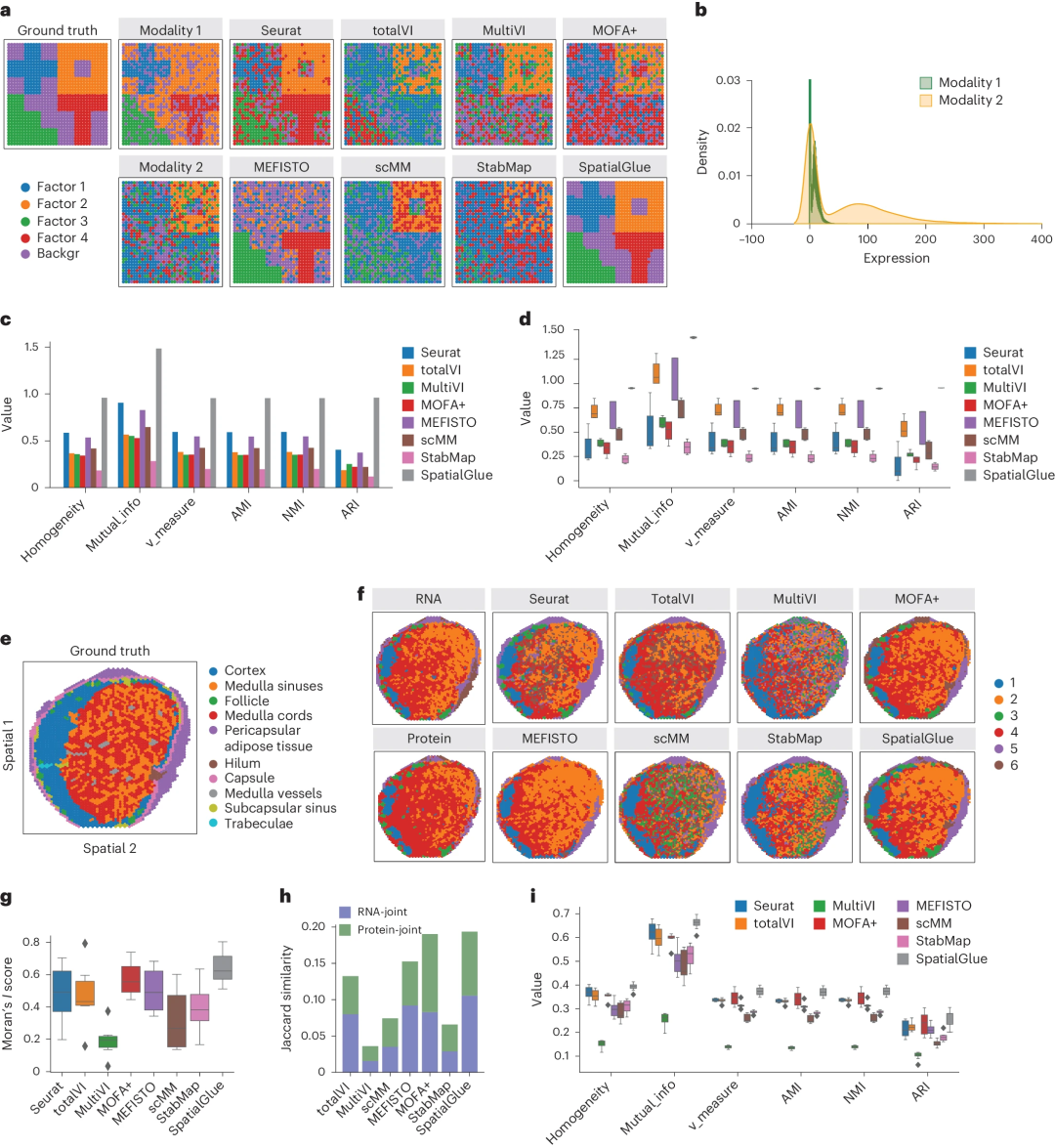
Une résolution plus élevée capture plus de détails anatomiques. Les chercheurs ont d'abord testé SpatialGlue sur des données de ganglions lymphatiques humains simulées et acquises expérimentalement et ont effectué des tests de référence. Les performances quantitatives de SpatialGlue surpassent celles des autres méthodes et capturent davantage de détails anatomiques.
L'analyse comparative quantitative démontre que SpatialGlue surpasse 10 méthodes de pointe à modalité unique et non spatiales sur 5 ensembles de données simulées et 12 du monde réel, soulignant l'importance de l'information spatiale et de l'intégration multi-omique.

1. Illustration : SpatialGlue identifie avec précision les domaines spatiaux dans les données simulées et réelles. (Source : papier)
- Ensuite, SpatialGlue a été appliqué à un ensemble de données sur l'épigénome et le transcriptome du cerveau de souris, révélant des couches corticales plus fines par rapport à l'étude originale, ce qui permet d'approfondir l'étude de la régulation des gènes à une résolution spatiale plus élevée.

Illustration : SpatialGlue dissèque le transcriptome épigénomique spatial d'échantillons de cerveau de souris à une résolution plus élevée. (Source : article)
Enfin, SpatialGlue a été appliqué davantage aux données acquises par Stereo-CITE-seq et SPOTS, démontrant sa large applicabilité à diverses plates-formes technologiques. Les chercheurs ont testé huit méthodes. Dans l’ensemble, SpatialGlue obtient le score le plus élevé en termes de similarité Jaccard et se classe deuxième dans le score I de Moran. Cette excellente performance a ensuite été reproduite en utilisant trois sections supplémentaires de thymus de souris.
Les chercheurs ont déclaré : « Nous pensons que SpatialGlue deviendra un outil d'analyse précieux pour les données spatiales multi-omiques, aujourd'hui et à l'avenir. Il existe plusieurs façons possibles d'étendre SpatialGlue. L'une d'entre elles consiste à utiliser des images comme modalités. Nous prévoyons pour étendre SpatialGlue, fusionnez les données d'image avec des couches d'agrégation attentionnelles au sein ou entre les modalités. Nous prévoyons également d'étendre les capacités de SpatialGlue en intégrant des données multi-omiques acquises à partir de coupes de tissus en série
Remarque : la couverture provient d'Internet
.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!




 Comment supprimer une base de données
Comment supprimer une base de données
 Explication détaillée du fichier de configuration du quartz
Explication détaillée du fichier de configuration du quartz
 Que faire si l'utilisation du processeur est trop élevée
Que faire si l'utilisation du processeur est trop élevée
 Plateforme de trading Bitcoin
Plateforme de trading Bitcoin
 Comment vérifier l'adresse Mac
Comment vérifier l'adresse Mac
 Méthode de récupération de base de données Oracle
Méthode de récupération de base de données Oracle
 Le rôle de la fonction mathématique en langage C
Le rôle de la fonction mathématique en langage C
 Utilisation de while
Utilisation de while