 Périphériques technologiques
Périphériques technologiques
 IA
IA
 Le grand modèle médical 3D open source SAT prend en charge 497 organoïdes et a des performances supérieures à 72 nnU-Nets. Il a été publié par l'équipe de l'Université Jiao Tong de Shanghai.
Le grand modèle médical 3D open source SAT prend en charge 497 organoïdes et a des performances supérieures à 72 nnU-Nets. Il a été publié par l'équipe de l'Université Jiao Tong de Shanghai.
Le grand modèle médical 3D open source SAT prend en charge 497 organoïdes et a des performances supérieures à 72 nnU-Nets. Il a été publié par l'équipe de l'Université Jiao Tong de Shanghai.

Auteur | Université Jiao Tong de Shanghai, Laboratoire d'intelligence artificielle de Shanghai
Éditeur | ScienceAI
Récemment, l'équipe conjointe de l'Université Jiao Tong de Shanghai et du Laboratoire d'intelligence artificielle de Shanghai a publié un grand modèle de segmentation d'images médicales 3D SAT (Segment Anything in scans radiologiques, pilotés par des invites textuelles), sur des images médicales 3D (CT, IRM, TEP), basées sur des invites textuelles pour réaliser une segmentation universelle de 497 types d'organes/lésions du corps humain. Toutes les données, codes et modèles sont open source.

Lien papier :https://arxiv.org/abs/2312.17183
Lien code :https://github.com/zhaoziheng/SAT
Lien données :https://github .com/zhaoziheng/SAT-DS/
Contexte de recherche
La segmentation des images médicales joue un rôle important dans une série de tâches cliniques telles que le diagnostic, la planification chirurgicale et la surveillance des maladies. Cependant, la recherche traditionnelle forme des modèles « dédiés » pour chaque tâche de segmentation spécifique, ce qui fait que chaque modèle « dédié » a un champ d'application relativement limité et est incapable de répondre efficacement et commodément à un large éventail de besoins de segmentation médicale.
Dans le même temps, les grands modèles de langage ont récemment connu un grand succès dans le domaine médical, et pour promouvoir davantage le développement de l'intelligence artificielle médicale générale, il est devenu nécessaire de construire un outil de segmentation médicale capable de connecter les capacités de langage et de positionnement.
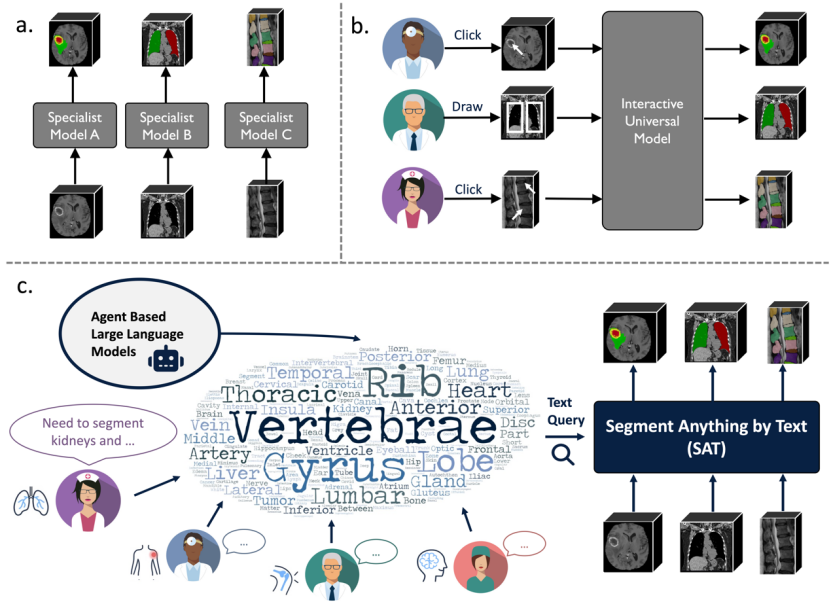
Pour surmonter ces défis, des chercheurs de l'Université Jiao Tong de Shanghai et du Laboratoire d'intelligence artificielle de Shanghai ont proposé le premier modèle de segmentation universel pour les images médicales 3D basé sur l'amélioration des connaissances et utilisant des invites textuelles, appelé SAT (Segment Anything in radiology scans), piloté par Text. invites), et a apporté les trois contributions principales suivantes :
1. Cette étude est la première à explorer l'injection de connaissances sur l'anatomie humaine dans un encodeur de texte pour coder avec précision les termes anatomiques et obtenir des invites de texte. .
2. Cette recherche construit le premier graphe de connaissances médicales multimodal contenant plus de 6 000 concepts d'anatomie humaine. Dans le même temps, le plus grand ensemble de données de segmentation d'images médicales 3D a été construit, appelé SAT-DS, qui rassemble 72 ensembles de données publiques, plus de 22 000 images issues des modalités CT, IRM et TEP, et plus de 302 000 annotations de segmentation, couvrant le corps humain 497. les objectifs de segmentation en 8 parties principales.
3. Basée sur SAT-DS, cette étude a formé deux modèles de tailles différentes : SAT-Pro (447 M de paramètres) et SAT-Nano (110 M de paramètres), et a conçu des expériences pour vérifier la valeur de SAT sous plusieurs angles : SAT The les performances sont équivalentes à celles de 72 modèles experts nnU-Nets (les paramètres sont ajustés et optimisés séparément sur chaque ensemble de données, soit un total d'environ 2,2 milliards de paramètres) et montrent une capacité de généralisation plus forte sur les données SAT hors domaine ; En tant que modèle de segmentation de base pré-entraîné sur des données à grande échelle, il peut afficher de meilleures performances que les nnU-Nets lorsqu'il est transféré à des tâches spécifiques via un réglage fin en aval. De plus, par rapport à MedSAM basé sur des invites de boîte, SAT peut obtenir des résultats plus précis et plus précis ; des performances précises basées sur des invites textuelles. Une segmentation plus efficace ; enfin, sur des données cliniques en dehors du domaine, l'équipe de recherche a démontré que SAT peut être utilisé comme outil proxy pour de grands modèles de langage, dotant directement ces derniers de capacités de localisation et de segmentation dans des tâches telles que comme génération de rapport.
Ce qui suit présentera les détails de l'article original sous trois aspects : les données, le modèle et les résultats expérimentaux.
Construction de données
Graphique de connaissances multimodal : Afin d'obtenir un encodage précis des termes anatomiques, l'équipe de recherche a d'abord collecté un graphe de connaissances multimodal contenant plus de 6 000 concepts de l'anatomie humaine, dont le contenu provient de trois sources :
1. Unified Medical Language System (UMLS) est un dictionnaire biomédical construit par la National Library of Medicine des États-Unis. L’équipe de recherche a extrait près de 230 000 concepts et définitions biomédicales, ainsi qu’un graphique de connaissances couvrant plus d’un million de relations mutuelles.
2. Connaissances faisant autorité en anatomie sur Internet. L'équipe de recherche a examiné 6 502 concepts de l'anatomie humaine et récupéré des informations pertinentes sur Internet à l'aide d'un grand modèle de langage amélioré par la récupération, obtenant plus de 6 000 concepts et définitions et une carte des connaissances couvrant plus de 38 000 relations entre les structures anatomiques.
3. Ensemble de données de segmentation publique. L'équipe de recherche a collecté un ensemble de données publiques de segmentation d'images médicales 3D à grande échelle et a connecté les zones segmentées via des concepts anatomiques (étiquettes de catégorie) avec les connaissances de la base de connaissances textuelle mentionnée ci-dessus pour fournir une comparaison visuelle des connaissances.
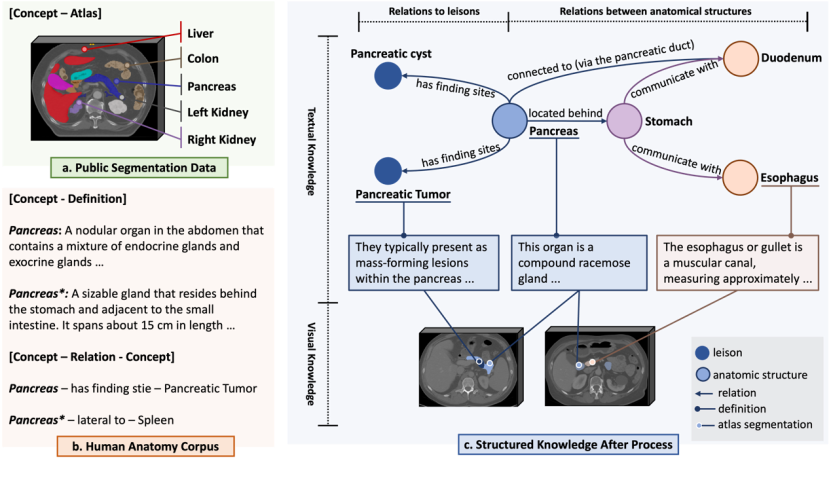
SAT-DS : Afin de former un modèle de segmentation universel, l'équipe de recherche a construit SAT-DS, la plus grande collection de données de segmentation d'images médicales 3D dans le domaine. En particulier, 72 ensembles de données de segmentation publics divers ont été collectés et organisés, comprenant un total de 22 186 images 3D, 302 033 annotations de segmentation, à partir de trois modalités : CT, IRM et TEP, et 497 segmentations couvrant 8 régions principales de la catégorie du corps humain (. structure anatomique ou lésion).
Afin de minimiser les différences entre les ensembles de données hétérogènes, l'équipe de recherche a standardisé l'orientation, l'espacement des voxels, la valeur de gris et d'autres attributs d'image entre différents ensembles de données, et a nommé les différents ensembles de données à l'aide d'une catégorie de segmentation de système de terminologie anatomique unifiée.

Figure 3 : SAT-DS est une collection de données de segmentation d'images médicales 3D diversifiées à grande échelle, couvrant un total de 497 catégories de segmentation dans 8 zones principales du corps humain.
Architecture du modèle
Injection de connaissances : Afin de créer un encodeur rapide capable d'encoder avec précision des termes anatomiques, l'équipe de recherche a d'abord injecté des connaissances anatomiques multimodales dans l'encodeur de texte en utilisant l'apprentissage contrastif.
Comme le montre la figure a ci-dessous, des concepts anatomiques sont utilisés pour connecter les connaissances multimodales en paires, puis un encodeur visuel et un encodeur de texte sont utilisés pour encoder respectivement les connaissances visuelles et textuelles, et les caractéristiques sont apprises par contraste en alignant les visuels. caractéristiques des structures anatomiques avec une connaissance textuelle dans l'espace et en construisant des relations entre les structures anatomiques, nous apprenons un meilleur codage des concepts anatomiques et servons d'indices pour guider la formation de modèles de segmentation visuelle.
Segmentation universelle basée sur des invites textuelles : L'équipe de recherche a en outre conçu un cadre de modèle de segmentation universel basé sur des invites textuelles, comme le montre la figure b ci-dessous, comprenant un encodeur de texte, un encodeur visuel, un décodeur visuel et un décodeur d'invites.
Parmi eux, étant donné que la même structure anatomique présente des différences dans différentes images, le décodeur de repères (décodeur de requête) utilise les caractéristiques de l'image produites par l'encodeur visuel pour améliorer les caractéristiques du concept anatomique, c'est-à-dire les indices de segmentation. Enfin, le produit scalaire est calculé entre l'indice de segmentation et les caractéristiques au niveau des pixels émises par le décodeur visuel pour obtenir le résultat de prédiction de segmentation.

Évaluation du modèle
Cette étude compare SAT à deux méthodes représentatives, à savoir le modèle « spécialisé » nnU-Nets et le modèle de segmentation générale interactif MedSAM. L'évaluation comprend deux aspects : les tests d'ensembles de données dans le domaine (performances de segmentation complètes) et les tests d'ensembles de données hors domaine (capacités de migration de données intercentres). Les résultats de l'évaluation sont intégrés à partir de trois niveaux : ensemble de données, catégorie et région du corps humain :
-
Catégorie : les résultats de segmentation de la même catégorie entre différents ensembles de données sont résumés et moyennés
Région : sur la base des résultats de catégorie, les résultats de catégorie au sein de la même zone d'anatomie humaine sont résumés et moyennés ;
Ensemble de données : traditionnel La méthode d'évaluation du modèle de segmentation, les résultats de segmentation au sein du même ensemble de données sont moyennés
Expérience comparative avec le modèle dédié nnU-Nets
Afin de maximiser le performances des nnU-Nets, l'étude a effectué une analyse de données distincte sur chaque donnée individuelle nnU-Nets formée sur l'ensemble et comparée à SAT. Les paramètres spécifiques sont les suivants :
1 Dans le test dans le domaine, les 72. Les ensembles de données de SAT-DS sont utilisés à des fins de test et de comparaison. Pour SAT, la somme de 72 ensembles de formation est utilisée pour la formation et testée sur 72 ensembles de tests ; pour les nnU-Nets, les résultats de 72 nnU-Nets sur leurs ensembles de tests respectifs sont résumés dans leur ensemble.
2. Dans le test hors domaine, 72 ensembles de données ont été divisés et les ensembles d'entraînement de 49 ensembles de données (nommés SAT-DS-Nano) ont été utilisés pour entraîner SAT-Nano et le test zéro tir ; pour les nnU-Nets, 49 nnU-Nets sont utilisés pour tester sur 10 ensembles de tests hors domaine et les résultats sont résumés.

Résultats des tests dans le domaine : Comme le montre le tableau 1, SAT-Pro a montré des performances très proches de celles de 72 nnU-Nets dans le test dans le domaine et a surpassé les nnU-Nets dans plusieurs domaines. Il convient de noter que SAT peut effectuer 72 tâches de segmentation avec un seul modèle et que la taille du modèle est beaucoup plus petite que l'ensemble des nnU-Nets (comme le montre la figure c ci-dessous).
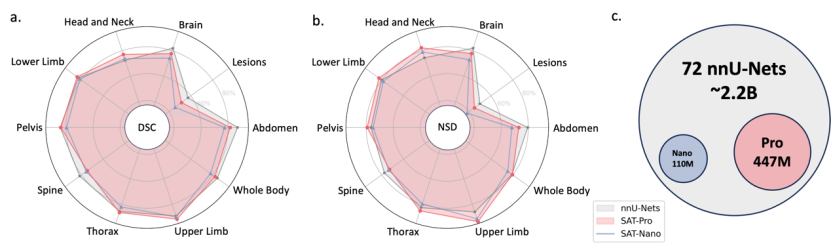
Résultats des tests de migration affinés : L'étude a ensuite testé SAT-Pro sur chaque ensemble de données après un réglage fin séparément, nommé SAT-Pro-Ft. Comme le montre le tableau 1, SAT-Pro-Ft présente des améliorations de performances significatives dans tous les domaines par rapport à SAT-Pro et dépasse nnU-Nets en termes de performances globales.
Résultats des tests hors domaine : Comme le montre le tableau 2, SAT-Nano a dépassé nnU-Nets dans 19 des 20 indicateurs dans 10 ensembles de données, montrant des capacités de migration globalement plus fortes.
Tableau 2 : Comparaison des tests hors domaine entre SAT-Nano, nnU-Nets et MedSAM Les résultats sont présentés en unités d'ensembles de données.

Expérience comparative avec le modèle de segmentation interactif MedSAM
Cette étude utilise directement le point de contrôle public de MedSAM pour les tests et la comparaison SAT. Les paramètres spécifiques sont les suivants :
1. données Nous avons en outre examiné 32 ensembles de données utilisés dans la formation MedSAM à des fins de comparaison.
2. Lors du test hors domaine, 5 ensembles de données qui n'ont pas été utilisés dans la formation MedSAM ont été sélectionnés à des fins de comparaison.
Pour MedSAM, envisagez deux invites Box différentes : en utilisant le plus petit rectangle (Oracle Box) contenant la segmentation de la vérité terrain, enregistré comme MedSAM (Tight) ; en ajoutant des décalages aléatoires basés sur Oracle Box, enregistré comme MedSAM (Loose). En même temps, testez l'effet d'Oracle Box directement en tant que prédiction. Pour SAT, le modèle de l'expérience de comparaison nnU-Nets est directement utilisé pour tester ces ensembles de données sans recyclage.
Résultats des tests dans le domaine :Comme le montre le tableau 3, SAT-Pro fonctionne mieux que MedSAM dans presque tous les domaines, et les performances globales de SAT-Pro et SAT-Nano sont meilleures que MedSAM. Bien que SAT-Pro soit moins performant que MedSAM sur les lésions, Oracle Box lui-même fonctionne assez bien sur les lésions en tant que prédiction, surpassant même MedSAM sur DSC. Cela indique que les performances supérieures de MedSAM dans la segmentation des lésions proviendront probablement des informations préalables solides fournies par Box.
Tableau 3 : Comparaison des tests dans le domaine de SAT-Pro, SAT-Nano et MedSAM, les résultats sont intégrés en unités de régions ou de lésions.

Comparaison qualitative : La figure 6 sélectionne deux exemples typiques à partir des résultats du test dans le domaine pour l'affichage visuel afin de comparer davantage SAT et MedSAM. Comme le montre la figure 6, dans la segmentation du myocarde, l'invite Box est difficile à distinguer entre le myocarde et les ventricules enveloppés par le myocarde. MedSAM a donc également segmenté par erreur les deux ensemble, ce qui montre que l'invite Box est similaire. relation spatiale complexe. Il est facile d’avoir des ambiguïtés, conduisant à une segmentation inexacte.
En revanche, la SAT basée sur des invites textuelles (saisie directe des noms des structures anatomiques) peut distinguer avec précision le myocarde et les ventricules. De plus, comme le montre la segmentation de la tumeur intestinale illustrée à la figure 6, Oracle Box constitue déjà un bon résultat de prédiction pour la cible de la lésion, tandis que le résultat de segmentation de MedSAM peut ne pas être meilleur que l'invite Box obtenue.
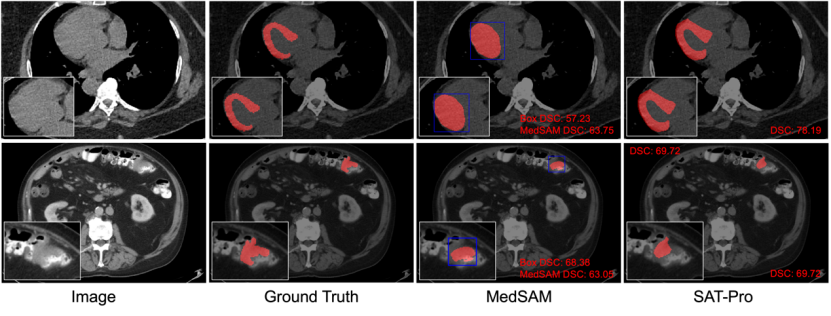
Figure 6 : Comparaison qualitative entre SAT-Pro et MedSAM (Tight). Parmi eux, MedSAM utilise Oracle Box comme invite, et la boîte est marquée en bleu. La première rangée montre un exemple de segmentation du myocarde ; la deuxième rangée montre un exemple de segmentation d'une tumeur intestinale.
Résultats des tests hors domaine : Comme le montre le tableau 2, par rapport à MedSAM (Tight), SAT-Nano a surpassé MedSAM dans 5 indicateurs sur 10 dans 5 ensembles de données. MedSAM (Loose) présente une dégradation évidente des performances dans tous les indicateurs, indiquant que MedSAM est plus sensible au décalage de l'invite Box saisie par l'utilisateur.
Expérience d'ablation
Lors de la conception de SAT, le réseau fédérateur visuel et l'encodeur de texte sont deux éléments clés. Cette recherche tente d'utiliser différentes structures de réseau visuel ou encodeurs de texte dans le cadre SAT, ainsi que des expériences d'ablation générales pour explorer leur influence.
Afin d'économiser le coût des expériences, tous les entraînements et tests de modèles SAT dans les expériences d'ablation sont effectués sur SAT-DS-Nano contenant 49 ensembles de données, qui contiennent 13 303 images 3D, 151 461 annotations de segmentation et 429 catégories divisées.
Réseau fédérateur visuel : Dans le cadre de SAT-Nano, cette étude a sélectionné trois structures de réseau de segmentation principales à des fins de comparaison, à savoir U-Net (110 M de paramètres), SwinUNETR (107 M de paramètres) et U-Mamba (114 M de paramètres). Pour une comparaison équitable, les quantités de paramètres qui les contrôlent dans cette expérience d'ablation sont à peu près similaires. Dans le même temps, afin de calculer la surcharge, l'étape d'injection de connaissances est omise et MedCPT est utilisé directement (MedCPT est un encodeur de texte basé sur la littérature PubMed, formé à l'aide de 225 millions de données de clics d'utilisateurs privés et a obtenu les meilleures performances en une série de tâches de langage médical) car l'encodeur de texte génère des indices. Les trois variantes sont respectivement appelées U-Net-CPT, SwinUNETR-CPT et U-Mamba-CPT.
Comme vous pouvez le voir sur la figure 7, en utilisant U-Net et U-Mamba comme réseau fédérateur visuel, les performances de segmentation finale sont relativement proches, avec U-Net légèrement meilleure que U-Mamba tandis que les performances de segmentation lors de l'utilisation de SwinUNETR ; est un déclin nettement meilleur. Enfin, l’équipe de recherche a choisi U-Net comme réseau fédérateur visuel pour SAT.
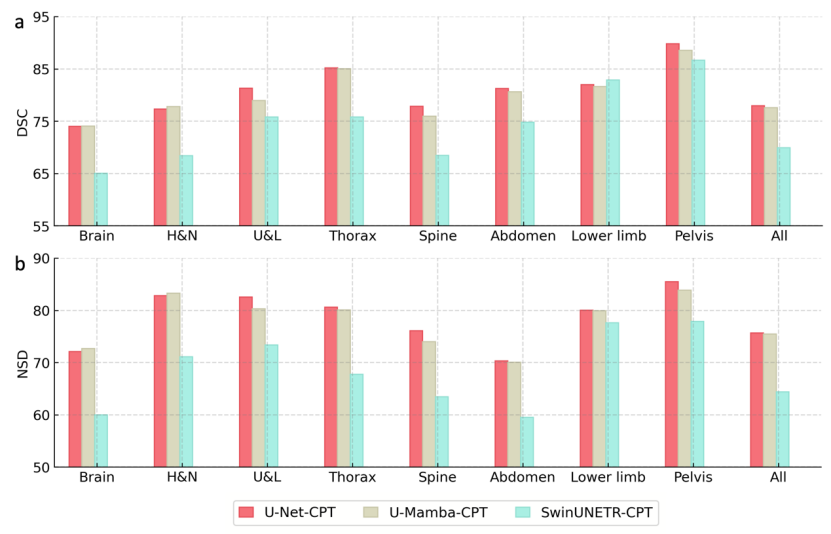
Encodeur de texte : Dans le cadre de SAT-Nano, cette étude a sélectionné trois encodeurs de texte représentatifs à des fins de comparaison : un encodeur de texte formé à l'aide de la méthode d'injection de connaissances proposée ci-dessus (notée Ours), l'état de l'art. l'encodeur de texte médical MedCPT est utilisé, ainsi que l'encodeur de texte BERT-base, qui n'est pas adapté aux données médicales, est utilisé.
Par souci d'équité, cette expérience d'ablation utilise uniformément U-Net comme réseau visuel. Les trois variantes sont respectivement appelées U-Net-Ours, U-Net-CPT et U-Net-BB. Comme le montre la figure 8, dans l'ensemble, l'utilisation de MedCPT présente une légère amélioration des performances de segmentation par rapport à l'utilisation de la base BERT, ce qui indique que la connaissance du domaine est utile pour fournir de bons conseils de segmentation, tandis que l'utilisation de l'encodeur de texte proposé dans cette étude a obtenu les meilleures performances ; obtenus dans toutes les catégories, ce qui indique que la construction d'une base de connaissances multimodale sur l'anatomie humaine et l'injection de connaissances sont très utiles pour les modèles de segmentation.

La distribution à longue traîne est une caractéristique évidente des ensembles de données segmentés. Comme le montrent les figures 9 a et b, l'équipe de recherche a étudié la répartition du nombre d'annotations de 429 catégories dans SAT-DS-Nano utilisé pour les expériences d'ablation. Si les 10 catégories avec le plus grand nombre d'annotations (2,33 %) sont définies comme classes de tête, et les 150 catégories avec le moins d'annotations (34,97 %) sont définies comme classes de queue, on peut constater que le nombre de les annotations pour les classes de queue ne représentent que 3,25 du nombre total d'annotations.
Cette étude explore plus en détail l'impact des encodeurs de texte sur les résultats de segmentation de différentes catégories dans les distributions longue traîne. Comme le montre la figure 9c, l'encodeur proposé par l'équipe de recherche a obtenu les meilleures performances dans les catégories tête, queue et milieu, l'amélioration dans la catégorie queue étant plus évidente que celle dans la catégorie tête. Dans le même temps, MedCPT a des performances légèrement inférieures à celles de la base BERT sur la classe principale, mais de meilleures performances sur la classe queue. Ces résultats montrent que la connaissance du domaine, en particulier l’injection de connaissances multimodales sur l’anatomie humaine, est considérablement utile pour la segmentation des catégories à longue traîne.

Combiné avec de grands modèles de langage
Étant donné que SAT peut être segmenté en fonction d'invites de texte, il peut être directement utilisé comme outil proxy pour les grands modèles de langage afin de fournir des capacités de segmentation. Afin de démontrer des scénarios d'application, l'équipe de recherche a sélectionné 4 données cliniques réelles diverses, a utilisé GPT4 pour extraire les cibles de segmentation du rapport et a appelé SAT pour segmentation sans tir. Les résultats sont présentés dans la figure 10.
Comme vous pouvez le voir, GPT-4 peut très bien détecter les structures anatomiques importantes dans le rapport et appeler SAT pour les segmenter très bien sur des images cliniques réelles sans aucun réglage précis des données.

Valeur de recherche
En tant que premier modèle de segmentation générale à grande échelle d'images médicales 3D basé sur des invites textuelles, la valeur de SAT se reflète dans de nombreux aspects :
SAT construit une segmentation universelle efficace et flexible : SAT-Pro utilise un seul modèle, affichant des performances comparables à 72 nnU-Nets sur un large éventail de tâches de segmentation, et dispose d'un plus petit nombre de paramètres de modèle. Cela montre que par rapport aux méthodes de segmentation médicale traditionnelles qui nécessitent la configuration, la formation et le déploiement d'une série de modèles spécialisés, SAT-Pro en tant que modèle de segmentation générale est une solution plus flexible et plus efficace. Dans le même temps, l'équipe de recherche a également prouvé que SAT-Pro offre de meilleures performances de généralisation sur les données hors région et peut mieux répondre aux besoins cliniques tels que la migration intercentres.
SAT est un modèle de base basé sur un pré-entraînement de données de segmentation à grande échelle : une fois que SAT-Pro est formé sur un ensemble de données de segmentation à grande échelle, il montre des améliorations significatives des performances lorsqu'il est transféré à un ensemble de données spécifique via un traitement fin. tuning et fonctionne globalement mieux que les nnU-Nets. Cela indique que SAT peut être considéré comme un modèle de segmentation de base puissant, capable de mieux fonctionner sur des tâches spécifiques grâce à un transfert affiné, équilibrant ainsi les besoins cliniques de la segmentation générale et de la segmentation spécialisée.
SAT obtient une segmentation précise et robuste basée sur des invites de texte : par rapport au modèle de segmentation interactif basé sur des invites de boîte, SAT basé sur des invites de texte peut obtenir des résultats de segmentation plus précis et plus robustes, et peut faire économiser beaucoup aux utilisateurs. de temps pour dessiner des boîtes, réalisant ainsi une segmentation universelle automatique et par lots.
SAT peut être utilisé comme outil proxy pour les grands modèles de langage : l'équipe de recherche a démontré sur des données cliniques réelles que SAT peut être connecté de manière transparente à de grands modèles de langage, en utilisant le texte comme pont pour fournir directement des capacités de segmentation et de positionnement à tout type de langage. grand modèle de langage. Ceci est d’une grande valeur pour promouvoir davantage le développement de l’intelligence artificielle médicale généraliste.
L'impact de la taille du modèle sur la segmentation : En entraînant deux modèles de tailles différentes : SAT-Nano et SAT-Pro, cette étude a observé que SAT-Pro a une amélioration significative par rapport à SAT-Nano dans le test in-domain . Cela implique que la loi d'échelle s'applique toujours lors de la formation de modèles de segmentation généraux sur des ensembles de données à grande échelle.
L'impact des connaissances du domaine sur la segmentation : L'équipe de recherche a proposé la première base de connaissances multimodale sur l'anatomie humaine et a exploré l'utilisation de l'amélioration des connaissances pour améliorer les performances des modèles de segmentation généraux, en particulier la segmentation des catégories à longue traîne. Étant donné que les annotations de segmentation, en particulier les annotations sur les catégories à longue traîne, sont relativement rares, cette exploration est d'une grande importance pour construire un modèle de segmentation général.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!

Outils d'IA chauds

Undresser.AI Undress
Application basée sur l'IA pour créer des photos de nu réalistes

AI Clothes Remover
Outil d'IA en ligne pour supprimer les vêtements des photos.

Undress AI Tool
Images de déshabillage gratuites

Clothoff.io
Dissolvant de vêtements AI

Video Face Swap
Échangez les visages dans n'importe quelle vidéo sans effort grâce à notre outil d'échange de visage AI entièrement gratuit !

Article chaud

Outils chauds

Bloc-notes++7.3.1
Éditeur de code facile à utiliser et gratuit

SublimeText3 version chinoise
Version chinoise, très simple à utiliser

Envoyer Studio 13.0.1
Puissant environnement de développement intégré PHP

Dreamweaver CS6
Outils de développement Web visuel

SublimeText3 version Mac
Logiciel d'édition de code au niveau de Dieu (SublimeText3)

Sujets chauds
 1670
1670
 14
14
 1428
1428
 52
52
 1329
1329
 25
25
 1276
1276
 29
29
 1256
1256
 24
24
 Comment utiliser la bibliothèque Chrono en C?
Apr 28, 2025 pm 10:18 PM
Comment utiliser la bibliothèque Chrono en C?
Apr 28, 2025 pm 10:18 PM
L'utilisation de la bibliothèque Chrono en C peut vous permettre de contrôler plus précisément les intervalles de temps et de temps. Explorons le charme de cette bibliothèque. La bibliothèque Chrono de C fait partie de la bibliothèque standard, qui fournit une façon moderne de gérer les intervalles de temps et de temps. Pour les programmeurs qui ont souffert de temps et ctime, Chrono est sans aucun doute une aubaine. Il améliore non seulement la lisibilité et la maintenabilité du code, mais offre également une précision et une flexibilité plus élevées. Commençons par les bases. La bibliothèque Chrono comprend principalement les composants clés suivants: std :: chrono :: system_clock: représente l'horloge système, utilisée pour obtenir l'heure actuelle. std :: chron
 Comment comprendre les opérations DMA en C?
Apr 28, 2025 pm 10:09 PM
Comment comprendre les opérations DMA en C?
Apr 28, 2025 pm 10:09 PM
DMA IN C fait référence à DirectMemoryAccess, une technologie d'accès à la mémoire directe, permettant aux périphériques matériels de transmettre directement les données à la mémoire sans intervention CPU. 1) L'opération DMA dépend fortement des dispositifs matériels et des pilotes, et la méthode d'implémentation varie d'un système à l'autre. 2) L'accès direct à la mémoire peut apporter des risques de sécurité et l'exactitude et la sécurité du code doivent être assurées. 3) Le DMA peut améliorer les performances, mais une mauvaise utilisation peut entraîner une dégradation des performances du système. Grâce à la pratique et à l'apprentissage, nous pouvons maîtriser les compétences de l'utilisation du DMA et maximiser son efficacité dans des scénarios tels que la transmission de données à grande vitesse et le traitement du signal en temps réel.
 Étapes pour ajouter et supprimer les champs aux tables MySQL
Apr 29, 2025 pm 04:15 PM
Étapes pour ajouter et supprimer les champs aux tables MySQL
Apr 29, 2025 pm 04:15 PM
Dans MySQL, ajoutez des champs en utilisant alterTableTable_namEaddColumnNew_Columnvarchar (255) AfterExist_Column, supprimez les champs en utilisant alterTableTable_NamedRopColumnColumn_to_drop. Lorsque vous ajoutez des champs, vous devez spécifier un emplacement pour optimiser les performances de la requête et la structure des données; Avant de supprimer les champs, vous devez confirmer que l'opération est irréversible; La modification de la structure de la table à l'aide du DDL en ligne, des données de sauvegarde, de l'environnement de test et des périodes de faible charge est l'optimisation des performances et les meilleures pratiques.
 Qu'est-ce que la programmation du système d'exploitation en temps réel en C?
Apr 28, 2025 pm 10:15 PM
Qu'est-ce que la programmation du système d'exploitation en temps réel en C?
Apr 28, 2025 pm 10:15 PM
C fonctionne bien dans la programmation du système d'exploitation en temps réel (RTOS), offrant une efficacité d'exécution efficace et une gestion du temps précise. 1) C répond aux besoins des RTO grâce à un fonctionnement direct des ressources matérielles et à une gestion efficace de la mémoire. 2) En utilisant des fonctionnalités orientées objet, C peut concevoir un système de planification de tâches flexible. 3) C prend en charge un traitement efficace d'interruption, mais l'allocation de mémoire dynamique et le traitement des exceptions doivent être évités pour assurer le temps réel. 4) La programmation des modèles et les fonctions en ligne aident à l'optimisation des performances. 5) Dans les applications pratiques, C peut être utilisé pour implémenter un système de journalisation efficace.
 Top 10 des plates-formes de trading de devises numériques: 10 premiers échanges de devises numériques sûrs et fiables
Apr 30, 2025 pm 04:30 PM
Top 10 des plates-formes de trading de devises numériques: 10 premiers échanges de devises numériques sûrs et fiables
Apr 30, 2025 pm 04:30 PM
Les 10 principales plates-formes de trading de devises virtuelles numériques sont: 1. Binance, 2. Okx, 3. Coinbase, 4. Kraken, 5. Huobi Global, 6. Bitfinex, 7. Kucoin, 8. Gemini, 9. Bitstamp, 10. Bittrex. Ces plateformes offrent toutes une haute sécurité et une variété d'options de trading, adaptées à différents besoins des utilisateurs.
 Classement d'échange quantitatif 2025 Top 10 des recommandations pour les applications de trading quantitatif de la monnaie numérique
Apr 30, 2025 pm 07:24 PM
Classement d'échange quantitatif 2025 Top 10 des recommandations pour les applications de trading quantitatif de la monnaie numérique
Apr 30, 2025 pm 07:24 PM
Les outils de quantification intégrés de l'échange comprennent: 1. Binance: fournit un module quantitatif à terme Binance Futures, des frais de manutention faible et prend en charge les transactions assistées par l'IA. 2. OKX (OUYI): prend en charge la gestion multi-comptes et le routage des ordres intelligents, et fournit un contrôle des risques au niveau institutionnel. Les plates-formes de stratégie quantitative indépendantes comprennent: 3. 3Commas: générateur de stratégie de glisser-déposer, adapté à l'arbitrage de la couverture multiplateforme. 4. Quadancy: Bibliothèque de stratégie d'algorithme de niveau professionnel, soutenant les seuils de risque personnalisés. 5. Pionex: stratégie prédéfinie intégrée, frais de transaction bas. Les outils de domaine vertical incluent: 6. CryptoPper: plate-forme quantitative basée sur le cloud, prenant en charge 150 indicateurs techniques. 7. Bitsgap:
 Comment mesurer les performances du fil en C?
Apr 28, 2025 pm 10:21 PM
Comment mesurer les performances du fil en C?
Apr 28, 2025 pm 10:21 PM
La mesure des performances du thread en C peut utiliser les outils de synchronisation, les outils d'analyse des performances et les minuteries personnalisées dans la bibliothèque standard. 1. Utilisez la bibliothèque pour mesurer le temps d'exécution. 2. Utilisez le GPROF pour l'analyse des performances. Les étapes incluent l'ajout de l'option -pg pendant la compilation, l'exécution du programme pour générer un fichier gmon.out et la génération d'un rapport de performances. 3. Utilisez le module Callgrind de Valgrind pour effectuer une analyse plus détaillée. Les étapes incluent l'exécution du programme pour générer le fichier callgrind.out et la visualisation des résultats à l'aide de Kcachegrind. 4. Les minuteries personnalisées peuvent mesurer de manière flexible le temps d'exécution d'un segment de code spécifique. Ces méthodes aident à bien comprendre les performances du thread et à optimiser le code.
 Comment le site officiel Deepseek réalise-t-il l'effet de l'événement de défilement de souris pénétrant?
Apr 30, 2025 pm 03:21 PM
Comment le site officiel Deepseek réalise-t-il l'effet de l'événement de défilement de souris pénétrant?
Apr 30, 2025 pm 03:21 PM
Comment réaliser l'effet de la pénétration des événements de défilement de la souris? Lorsque nous naviguons sur le Web, nous rencontrons souvent des conceptions d'interaction spéciales. Par exemple, sur le site officiel Deepseek, � ...



