

Les protéines sont impliquées dans de nombreuses fonctions biologiques telles que la composition cellulaire, la contraction musculaire, la digestion des aliments et l'identification des virus.
Afin de concevoir de meilleures protéines (y compris des anticorps), les scientifiques font souvent muter à plusieurs reprises les acides aminés (en organisant les unités qui composent les protéines dans un certain ordre) à différentes positions jusqu'à ce que la protéine obtienne la fonction requise.
Mais il y a plus de séquences d’acides aminés qu’il n’y a de grains de sable dans le monde, donc trouver la meilleure protéine, et donc le meilleur médicament potentiel, est souvent difficile. Face à ce défi, les scientifiques dépensent souvent des millions de dollars et testent des versions miniaturisées et simplifiées de systèmes biologiques.
« Cela nécessite beaucoup de conjectures et de vérifications. » Brian L. Hie, professeur adjoint de génie chimique à l'Université de Stanford et chercheur en innovation à l'Arc Institute, a déclaré : « L'objectif de nombreux algorithmes intelligents est d'éliminer les conjectures. "
Les scientifiques de l'Université de Stanford ont développé une nouvelle méthode basée sur l'apprentissage automatique qui peut prédire plus rapidement et avec plus de précision les changements moléculaires conduisant à de meilleurs anticorps. En combinant la structure 3D du squelette protéique avec un grand modèle de langage basé sur la séquence d’acides aminés, les chercheurs ont pu trouver des mutations rares et souhaitables en quelques minutes.
L'étude s'intitulait « Evolution non supervisée des complexes de protéines et d'anticorps avec un modèle de langage informé par la structure » et a été publiée dans « Science » le 4 juillet 2024.

Les grands modèles de langage formés uniquement sur les informations de séquence peuvent apprendre les principes de haut niveau de la conception des protéines. Cependant, outre la séquence, la structure tridimensionnelle des protéines détermine leur fonction, leur activité et leur évolutivité spécifiques.
Pour les problèmes d'ingénierie des anticorps, des chercheurs de l'Université de Stanford ont appliqué des modèles de langage protéique structurellement informés pour prédire des séquences de haute condition physique contraintes par des structures complexes d'anticorps ou d'anticorps-antigène connues.
La recherche montre qu'un modèle universel de langage protéique augmenté de coordonnées du squelette structurel des protéines peut guider l'évolution de différentes protéines sans qu'il soit nécessaire de modéliser des tâches fonctionnelles individuelles.
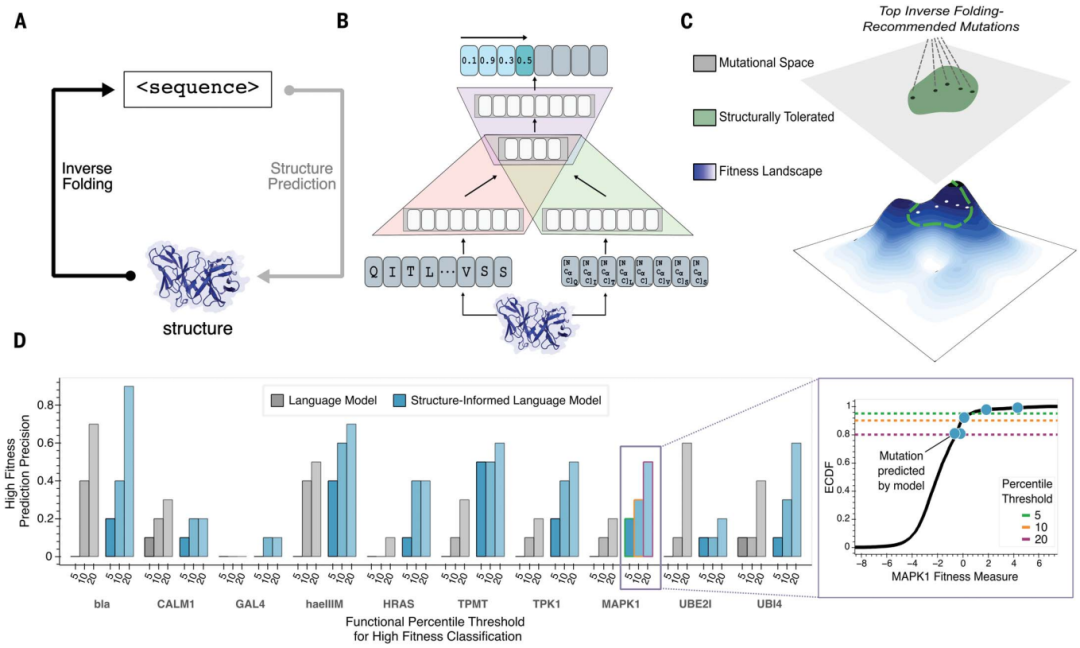
Paradigme guidé par la structure :
Large application :
Conception de complexes protéiques :
Évolution des anticorps humains :
Remplacer de grandes quantités de données :
Évolution dirigée :

Avec cette méthode, l'équipe a sélectionné environ 30 candidats pour deux anticorps cliniques thérapeutiques pour le traitement des variantes du coronavirus 2 (SRAS-CoV-2) du syndrome respiratoire aigu sévère. Dans le même temps, les chercheurs ont obtenu une neutralisation multipliée par 25 et une affinité multipliée par 37 contre les variantes du virus d’évasion d’anticorps BQ.1.1 et XBB.1.5, respectivement.
En conclusion, Cet outil aidera à répondre rapidement aux maladies nouvelles ou en développement. Cela réduit également les obstacles à la fabrication de médicaments plus efficaces. Des médicaments plus puissants nécessitent des doses plus faibles, ce qui signifie qu’un plus grand nombre de patients peuvent bénéficier d’une dose donnée.
Lien papier : https://www.science.org/doi/10.1126/science.adk8946
Rapports associés : https://phys.org/news/2024-07-ai-approach-optimizes- anticorps-drogues.html
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!