 Périphériques technologiques
Périphériques technologiques
 IA
IA
 Nouvelle norme pour l'imagerie IA, seulement 1 % des données originales peuvent atteindre les meilleures performances, modèle de base médical général publié dans la sous-journal Nature
Nouvelle norme pour l'imagerie IA, seulement 1 % des données originales peuvent atteindre les meilleures performances, modèle de base médical général publié dans la sous-journal Nature
Nouvelle norme pour l'imagerie IA, seulement 1 % des données originales peuvent atteindre les meilleures performances, modèle de base médical général publié dans la sous-journal Nature

Éditeur | Feuille de chou
Le modèle de base pré-entraîné à grande échelle a connu un grand succès dans les domaines non médicaux. Cependant, la formation de ces modèles nécessite souvent des ensembles de données volumineux et complets, contrairement aux ensembles de données plus petits et plus spécialisés courants en imagerie biomédicale.
Des chercheurs de l'Institut Fraunhofer de médecine numérique MEVIS en Allemagne ont proposé une stratégie d'apprentissage multitâche qui sépare le nombre de tâches de formation des besoins en mémoire.
Ils ont formé un modèle biomédical pré-entraîné universel (UMedPT) sur une base de données multitâche comprenant des images de tomographie, de microscopie et de rayons X et ont utilisé diverses stratégies d'étiquetage telles que la classification, la segmentation et la détection d'objets. Le modèle de base UMedPT surpasse les modèles STOA pré-entraînés ImageNet et précédents.
Dans le cadre d'une validation externe indépendante, il a été prouvé que les caractéristiques d'imagerie extraites à l'aide de l'UMedPT établissent une nouvelle norme en matière de transférabilité intercentrique.
L'étude s'intitulait « Surmonter la rareté des données en imagerie biomédicale avec un modèle multitâche fondamental » et a été publiée dans « Nature Computational Science » le 19 juillet 2024.

Le deep learning révolutionne progressivement l'analyse d'images biomédicales grâce à sa capacité à apprendre et à extraire des représentations d'images utiles.
La méthode générale consiste à pré-entraîner le modèle sur un ensemble de données d'images naturelles à grande échelle (comme ImageNet ou LAION), puis à l'affiner pour des tâches spécifiques ou à utiliser directement les fonctionnalités pré-entraînées. Mais le réglage fin nécessite davantage de ressources informatiques.
Dans le même temps, le domaine de l’imagerie biomédicale nécessite une grande quantité de données annotées pour une pré-formation efficace en apprentissage profond, mais ces données sont souvent rares.
L'apprentissage multitâche (MTL) fournit une solution à la rareté des données en entraînant un modèle pour résoudre plusieurs tâches simultanément. Il exploite de nombreux ensembles de données de petite et moyenne taille en imagerie biomédicale pour pré-entraîner des représentations d'images adaptées à toutes les tâches et convient aux domaines où les données sont rares.
MTL a été appliqué à l'analyse d'images biomédicales de diverses manières, notamment en s'entraînant à partir de plusieurs ensembles de données de petite et moyenne taille pour différentes tâches et en utilisant plusieurs types d'étiquettes sur une seule image, démontrant que les fonctionnalités partagées peuvent améliorer les performances des tâches.
Dans les dernières recherches, afin de combiner plusieurs ensembles de données avec différents types d'étiquettes pour une pré-formation à grande échelle, des chercheurs de l'Institut MEVIS ont introduit une stratégie de formation multitâche et une architecture de modèle correspondante, notamment grâce à l'apprentissage de représentations polyvalentes selon différentes modalités. , maladies et types d'étiquettes pour remédier à la rareté des données en imagerie biomédicale.
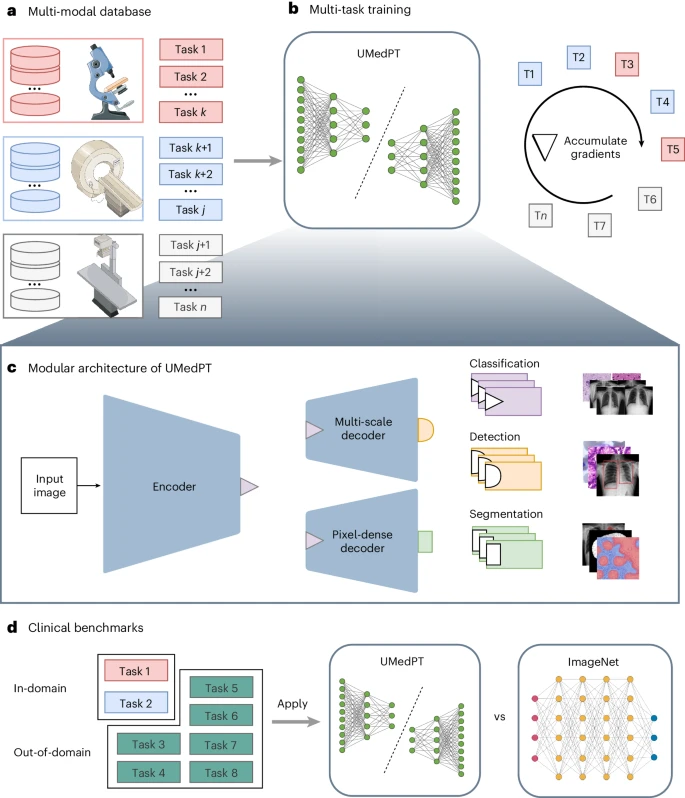
Pour faire face aux contraintes de mémoire rencontrées dans l'apprentissage multitâche à grande échelle, cette méthode adopte une boucle d'entraînement basée sur l'accumulation de gradient, dont l'expansion est presque illimitée par le nombre de tâches d'entraînement.
Sur cette base, les chercheurs ont formé un modèle de base d'imagerie biomédicale entièrement supervisé appelé UMedPT en utilisant 17 tâches et leurs annotations originales.
L'image ci-dessous montre l'architecture du réseau neuronal de l'équipe, qui se compose de blocs partagés comprenant un encodeur, un décodeur de segmentation et un décodeur de localisation, ainsi que des têtes spécifiques à des tâches. Les blocs partagés sont formés pour être applicables à toutes les tâches de pré-formation, aidant ainsi à extraire les caractéristiques communes, tandis que les superviseurs spécifiques aux tâches gèrent les calculs et les prévisions de pertes spécifiques aux étiquettes.
Les tâches définies comprennent trois types d'étiquettes supervisées : détection d'objets, segmentation et classification. Par exemple, les tâches de classification peuvent modéliser des biomarqueurs binaires, les tâches de segmentation peuvent extraire des informations spatiales et les tâches de détection d'objets peuvent être utilisées pour former des biomarqueurs basés sur le nombre de cellules.
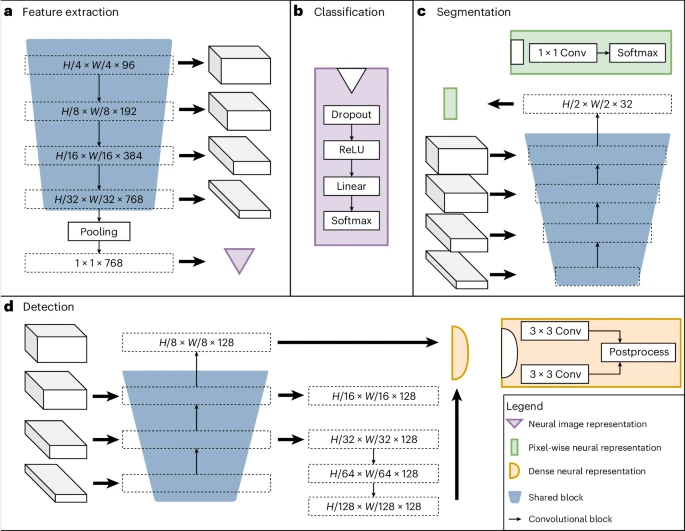
Illustration : L’architecture de l’UMedPT. (Source : article)
UMedPT correspond ou surpasse systématiquement les réseaux ImageNet pré-entraînés sur les tâches dans le domaine et hors domaine, tout en conservant de solides performances en utilisant moins de données d'entraînement lors de l'application directe de la représentation d'image (gel) et du réglage fin des paramètres.
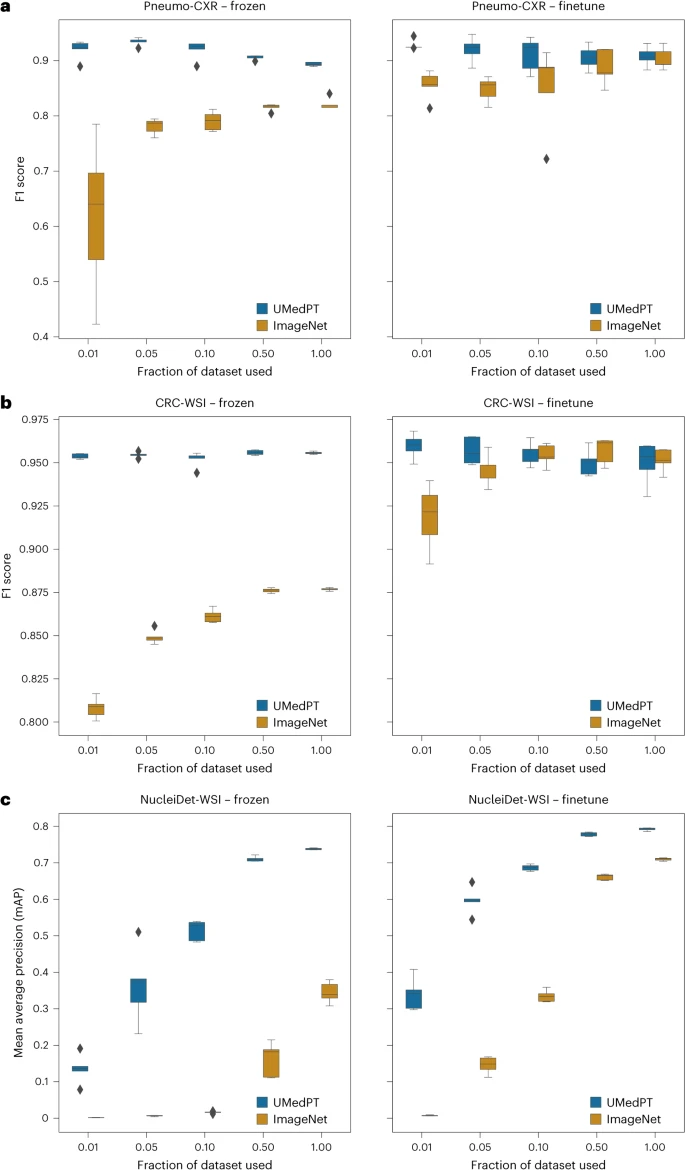
Illustration : Résultats des tâches au sein du domaine. (Source : article)
Pour les tâches de classification associées aux bases de données pré-entraînées, UMedPT est capable d'obtenir les meilleures performances de la base ImageNet sur toutes les configurations en utilisant seulement 1 % des données d'entraînement d'origine. Ce modèle atteint des performances supérieures en utilisant des encodeurs gelés par rapport au modèle utilisant un réglage fin.
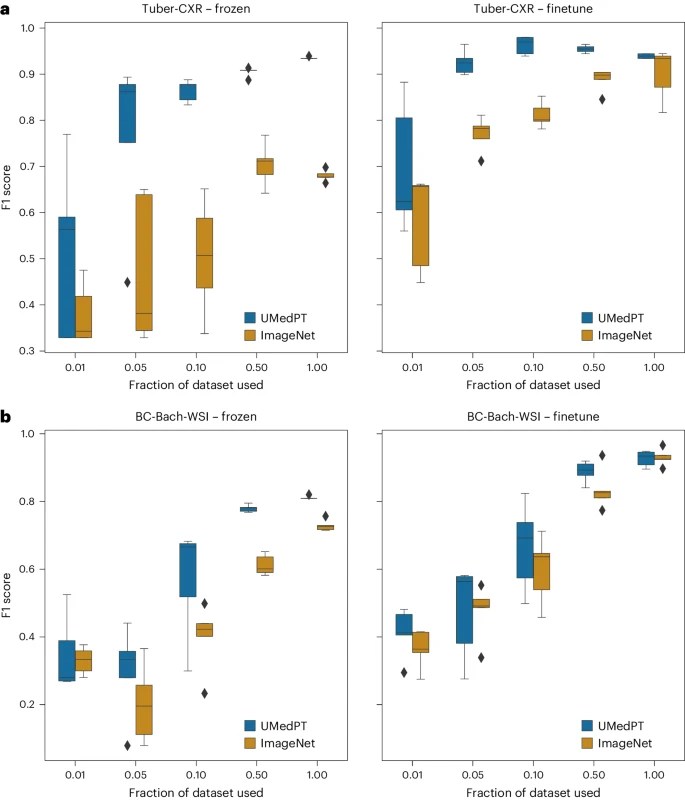
Illustration : Résultats pour les tâches hors domaine (source : papier)
Pour les tâches hors domaine, UMedPT est capable d'égaler les performances d'ImageNet en utilisant seulement 50 % ou moins de données, même avec des données fines. réglage appliqué.
De plus, les chercheurs ont comparé les performances de l'UMedPT avec les résultats rapportés dans la littérature. Lors de l'utilisation de la configuration d'encodeur gelée, UMedPT a dépassé les résultats de référence externe dans la plupart des tâches. Dans ce contexte, il surpasse également l'aire moyenne sous la courbe (AUC) de la base de données MedMNIST 16 .
Il est à noter que les tâches pour lesquelles l'application gelée de l'UMedPT n'a pas surpassé les résultats de référence étaient hors du domaine (BC-Bach-WSI pour la classification du cancer du sein et CNS-MRI pour le diagnostic des tumeurs du SNC). Avec un réglage fin, la pré-formation avec UMedPT surpasse les résultats de référence externe dans toutes les tâches.

Illustration : La quantité de données requise pour que l'UMedPT puisse atteindre des performances de pointe sur des tâches dans différents domaines d'imagerie. (Source : article)
En tant que base des développements futurs dans les domaines où les données sont rares, l'UMedPT ouvre la perspective d'applications d'apprentissage profond dans des domaines médicaux où la collecte de grandes quantités de données est particulièrement difficile, comme les maladies rares et l'imagerie pédiatrique.
Lien papier :https://www.nature.com/articles/s43588-024-00662-z
Contenu associé :https://www.nature.com/articles/s43588-024-00658- 9
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!

Outils d'IA chauds

Undresser.AI Undress
Application basée sur l'IA pour créer des photos de nu réalistes

AI Clothes Remover
Outil d'IA en ligne pour supprimer les vêtements des photos.

Undress AI Tool
Images de déshabillage gratuites

Clothoff.io
Dissolvant de vêtements AI

AI Hentai Generator
Générez AI Hentai gratuitement.

Article chaud

Outils chauds

Bloc-notes++7.3.1
Éditeur de code facile à utiliser et gratuit

SublimeText3 version chinoise
Version chinoise, très simple à utiliser

Envoyer Studio 13.0.1
Puissant environnement de développement intégré PHP

Dreamweaver CS6
Outils de développement Web visuel

SublimeText3 version Mac
Logiciel d'édition de code au niveau de Dieu (SublimeText3)

Sujets chauds
 Bytedance Cutting lance le super abonnement SVIP : 499 yuans pour un abonnement annuel continu, offrant une variété de fonctions d'IA
Jun 28, 2024 am 03:51 AM
Bytedance Cutting lance le super abonnement SVIP : 499 yuans pour un abonnement annuel continu, offrant une variété de fonctions d'IA
Jun 28, 2024 am 03:51 AM
Bytedance Cutting lance le super abonnement SVIP : 499 yuans pour un abonnement annuel continu, offrant une variété de fonctions d'IA
 Repoussant les limites de la détection de défauts traditionnelle, « Defect Spectrum » permet pour la première fois une détection de défauts industriels d'une ultra haute précision et d'une sémantique riche.
Jul 26, 2024 pm 05:38 PM
Repoussant les limites de la détection de défauts traditionnelle, « Defect Spectrum » permet pour la première fois une détection de défauts industriels d'une ultra haute précision et d'une sémantique riche.
Jul 26, 2024 pm 05:38 PM
Repoussant les limites de la détection de défauts traditionnelle, « Defect Spectrum » permet pour la première fois une détection de défauts industriels d'une ultra haute précision et d'une sémantique riche.
 Le modèle de dialogue NVIDIA ChatQA a évolué vers la version 2.0, avec la longueur du contexte mentionnée à 128 Ko
Jul 26, 2024 am 08:40 AM
Le modèle de dialogue NVIDIA ChatQA a évolué vers la version 2.0, avec la longueur du contexte mentionnée à 128 Ko
Jul 26, 2024 am 08:40 AM
Le modèle de dialogue NVIDIA ChatQA a évolué vers la version 2.0, avec la longueur du contexte mentionnée à 128 Ko
 Google AI a remporté la médaille d'argent de l'Olympiade mathématique de l'OMI, le modèle de raisonnement mathématique AlphaProof a été lancé et l'apprentissage par renforcement est de retour.
Jul 26, 2024 pm 02:40 PM
Google AI a remporté la médaille d'argent de l'Olympiade mathématique de l'OMI, le modèle de raisonnement mathématique AlphaProof a été lancé et l'apprentissage par renforcement est de retour.
Jul 26, 2024 pm 02:40 PM
Google AI a remporté la médaille d'argent de l'Olympiade mathématique de l'OMI, le modèle de raisonnement mathématique AlphaProof a été lancé et l'apprentissage par renforcement est de retour.
 Afin de fournir un nouveau système de référence et d'évaluation de questions-réponses scientifiques et complexes pour les grands modèles, l'UNSW, Argonne, l'Université de Chicago et d'autres institutions ont lancé conjointement le cadre SciQAG.
Jul 25, 2024 am 06:42 AM
Afin de fournir un nouveau système de référence et d'évaluation de questions-réponses scientifiques et complexes pour les grands modèles, l'UNSW, Argonne, l'Université de Chicago et d'autres institutions ont lancé conjointement le cadre SciQAG.
Jul 25, 2024 am 06:42 AM
Afin de fournir un nouveau système de référence et d'évaluation de questions-réponses scientifiques et complexes pour les grands modèles, l'UNSW, Argonne, l'Université de Chicago et d'autres institutions ont lancé conjointement le cadre SciQAG.
 Le point de vue de la nature : les tests de l'intelligence artificielle en médecine sont dans le chaos. Que faut-il faire ?
Aug 22, 2024 pm 04:37 PM
Le point de vue de la nature : les tests de l'intelligence artificielle en médecine sont dans le chaos. Que faut-il faire ?
Aug 22, 2024 pm 04:37 PM
Le point de vue de la nature : les tests de l'intelligence artificielle en médecine sont dans le chaos. Que faut-il faire ?
 Formation avec des millions de données cristallines pour résoudre le problème de la phase cristallographique, la méthode d'apprentissage profond PhAI est publiée dans Science
Aug 08, 2024 pm 09:22 PM
Formation avec des millions de données cristallines pour résoudre le problème de la phase cristallographique, la méthode d'apprentissage profond PhAI est publiée dans Science
Aug 08, 2024 pm 09:22 PM
Formation avec des millions de données cristallines pour résoudre le problème de la phase cristallographique, la méthode d'apprentissage profond PhAI est publiée dans Science
 Les performances de SOTA, la méthode d'IA de prédiction d'affinité protéine-ligand multimodale de Xiamen, combinent pour la première fois des informations sur la surface moléculaire
Jul 17, 2024 pm 06:37 PM
Les performances de SOTA, la méthode d'IA de prédiction d'affinité protéine-ligand multimodale de Xiamen, combinent pour la première fois des informations sur la surface moléculaire
Jul 17, 2024 pm 06:37 PM
Les performances de SOTA, la méthode d'IA de prédiction d'affinité protéine-ligand multimodale de Xiamen, combinent pour la première fois des informations sur la surface moléculaire





