

Éditeur | ScienceAI
En utilisant des modèles de séquence avancés tels que Transformer, le problème de prédiction de rétrosynthèse en une seule étape est transformé en une tâche de traduction de la représentation SMILES du produit vers la représentation SMILES du réactif, qui est devenue une stratégie largement utilisée avec des résultats remarquables.
Cependant, cette méthode ignore souvent un point clé : entre les réactifs et les produits, il existe un grand nombre de sous-structures identiques qui peuvent être directement utilisées. Une utilisation inadéquate de ces sous-structures limite l’efficacité et la précision des prédictions du modèle.
En juillet 2024, l'équipe de recherche de Jin Yaohui et Xu Yanyan de l'Institut d'intelligence artificielle de l'Université Jiao Tong de Shanghai a publié un article « Ualign : repousser les limites de la prédiction de rétrosynthèse sans modèle avec un alignement SMILES non supervisé » dans le "Journal de Chemininformatique".
Dans l'étude, l'auteur a proposé un processus de prédiction rétrosynthétique en une seule étape, qui intégrait une technologie d'alignement de séquences SMILES non supervisée, visant à améliorer la précision et l'efficacité de la prédiction des réactions chimiques. Les résultats expérimentaux démontrent l'efficacité du modèle pour prédire les voies rétrosynthétiques et suggèrent que le modèle a le potentiel de devenir un outil précieux pour la découverte de médicaments.
Architecture modèle de graphe en séquence
Si les atomes sont considérés comme des nœuds, En traitant les liaisons chimiques comme des arêtes, la structure moléculaire peut être naturellement transformée en structure graphique. Par rapport aux modèles de séquence, les réseaux de neurones graphiques peuvent mieux capturer les informations sur la structure topologique à l'intérieur des molécules, obtenant ainsi une caractérisation moléculaire plus précise. De plus, par rapport à d'autres structures graphiques, les liaisons chimiques dans les molécules chimiques contiennent de riches informations sur les propriétés chimiques. Sur la base de ces avantages, l'auteur propose une variante basée sur Graph Attention Network pour remplacer la partie encodeur dans le modèle Transformer, visant à fournir des capacités de représentation moléculaire plus puissantes pour les applications en aval.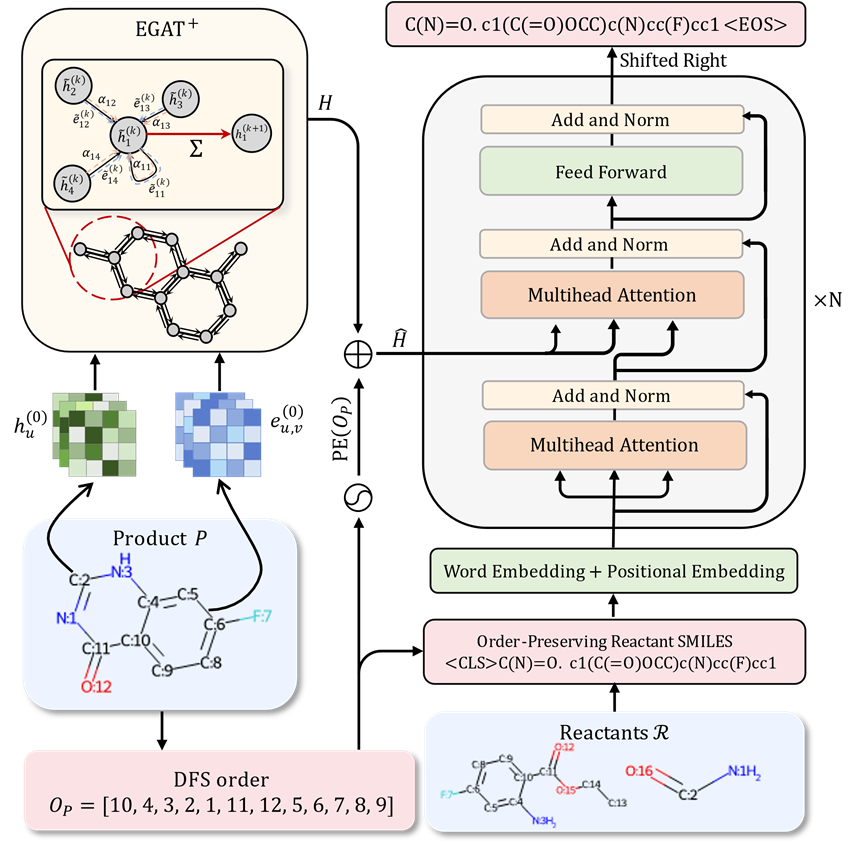
Mécanisme d'alignement SMILES non supervisé
Dans la prédiction rétrosynthétique en une seule étape, l'utilisation de méthodes de modélisation de séquence signifie généralement que la structure des réactifs doit être construite à partir de zéro, et ne peut pas apporter de modifications directes basées sur des produits existants pour utiliser efficacement des sous-structures identiques entre les réactifs et les produits. Cette approche limite dans une certaine mesure la précision des résultats générés. Considérant que la représentation moléculaire SMILES couramment utilisée dans la modélisation de séquences organise en fait les atomes et les liaisons chimiques dans la molécule dans l'ordre de recherche en profondeur d'abord, si les informations de position de chaque atome produit apparaissant dans la représentation SMILES du réactif peuvent être fournies à le modèle, cela aidera le modèle à identifier et à réutiliser les sous-structures qui n'ont pas changé pendant la réaction. Cela réduira considérablement la difficulté pour le modèle de prédire les réactifs et améliorera la précision des prédictions. Du point de vue de la modélisation de séquences, la caractérisation moléculaire SMILES couramment utilisée organise essentiellement les atomes et les liaisons chimiques dans la molécule selon l'ordre de recherche en profondeur d'abord (DFS). Si les informations de position de chaque atome dans le produit dans la représentation SMILES des réactifs peuvent être fournies au modèle, cela facilitera grandement l'identification et la réutilisation des sous-structures inchangées par le modèle, réduisant ainsi considérablement la difficulté de prédire les réactifs et améliorant la précision des prédictions. . Cependant, fournir directement ces informations de correspondance peut introduire un risque de fuite d'informations lors de la formation du modèle. Pour éviter ce problème, les chercheurs ont proposé une stratégie innovante pour optimiser la capacité du modèle à comprendre et prédire la structure moléculaire des réactifs sans divulguer les informations sur les étiquettes. Considérant que la caractérisation de la séquence SMILES est dérivée d'une recherche en profondeur sur des graphiques moléculaires et que la plupart des sous-structures entre les réactifs et les produits sont hautement cohérentes, pour une séquence DFS donnée de tout produit, il doit y en avoir une correspondante. L'ordre DFS sur la séquence moléculaire Le diagramme des réactifs est tel que les atomes correspondants sur les réactifs et les produits apparaissent presque dans le même ordre. Sur la base de cette stratégie, les chercheurs ont non seulement incorporé la structure moléculaire du produit dans l'entrée du modèle, mais ont également introduit l'ordre DFS des molécules réactives dans le cadre de l'entrée. De plus, selon la stratégie ci-dessus, les chercheurs ont généré une séquence DFS de molécule de produit hautement cohérente avec la séquence DFS d'un réactif donné, et ont utilisé cette séquence pour générer une représentation SMILES du réactif comme cible de la formation du modèle. . Cette conception permet à des sous-structures similaires entre les réactifs et les produits d'être disposées presque dans le même ordre dans l'entrée et la sortie du modèle, simplifiant ainsi le processus d'apprentissage du modèle par la même correspondance structurelle entre les réactifs et les produits et aidant à identifier les groupes. qui change au cours de la réaction.Même lorsque la structure du réactif est construite à partir de zéro, cette méthode peut réutiliser efficacement les informations sur la structure du produit et améliorer considérablement la précision de la prédiction.
Il est particulièrement important que, puisque l'ordre DFS du produit est uniquement basé sur ses informations de structure moléculaire et ne repose sur aucune information sur les réactifs comme annotations, cette méthode évite efficacement le problème de fuite d'étiquette pendant le processus de formation du modèle.
Dans le même temps, cette méthode d'alignement SMILES non supervisée ne nécessite pas l'introduction de signaux de supervision supplémentaires pendant le processus de formation, évitant ainsi des problèmes complexes d'annotation et d'optimisation de données dans l'apprentissage multitâche, et fournit une nouvelle méthode pour le domaine de l'analyse moléculaire. prédiction de la rétrosynthèse et méthodes de recherche efficaces.
Affichage des résultats expérimentaux
Dans cette étude, l'auteur a mené une évaluation systématique de plusieurs ensembles de données de prédiction de rétrosynthèse moléculaire, couvrant l'ensemble de données USPTO-50K largement utilisé, ainsi que l'ensemble de données USPTO-50K avec une plus grande quantité de données. MIT et USPTO-FULL.
Lors de l'évaluation des performances du modèle, la précision top-k est utilisée comme principal indice d'évaluation. Sur l'ensemble de données USPTO-50K, l'auteur a non seulement examiné la légalité de la séquence SMILES générée par le modèle, mais a également effectué une vérification en boucle de la faisabilité pratique du schéma de synthèse produit par le modèle via un pré-entraînement à grande échelle. modèle de prédiction de réaction directe.
Tableau 1 : Précision Top-k des prédictions rétrosynthétiques de l'USPTO-50K
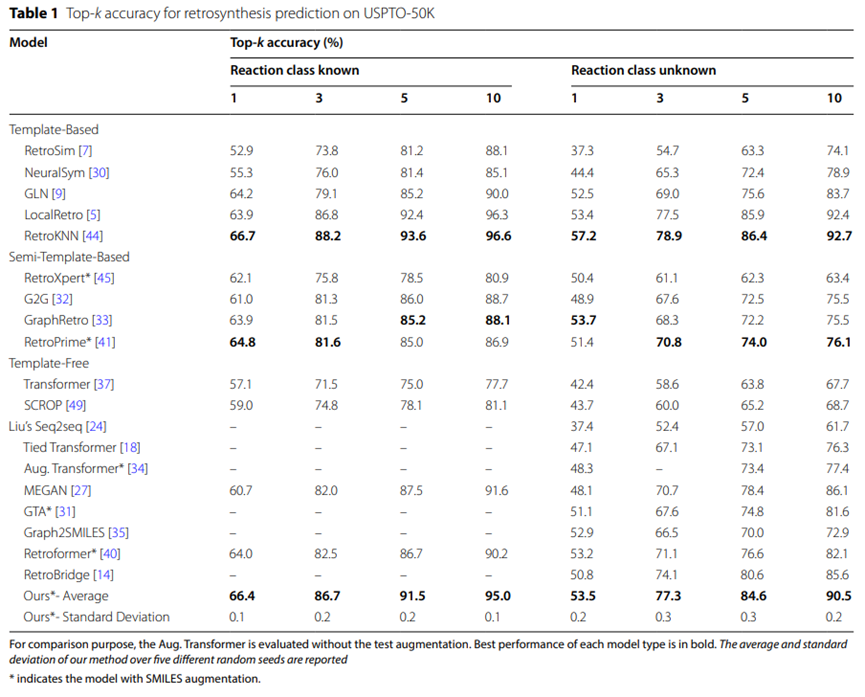
Les résultats expérimentaux de l'ensemble de données USPTO-50K sont résumés dans le tableau 1, montrant que le modèle UAlign fonctionne mieux dans l'USPTO lorsque le type de réaction spécifique n'est pas spécifié. La précision du top 5 sur l'ensemble de données -50K atteint 84,6 %, ce qui est nettement meilleur que les autres modèles de référence sans modèle.
Tableau 2 : Précision Top-k de la prédiction rétrosynthétique USPTO-MIT
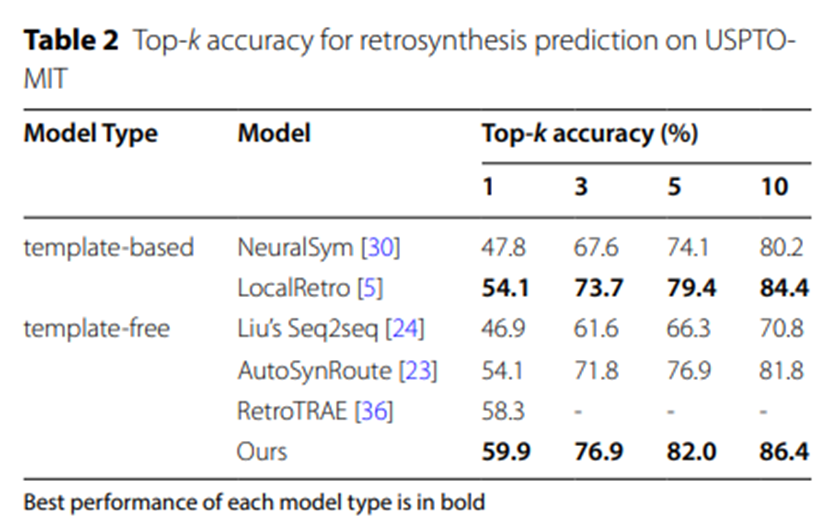
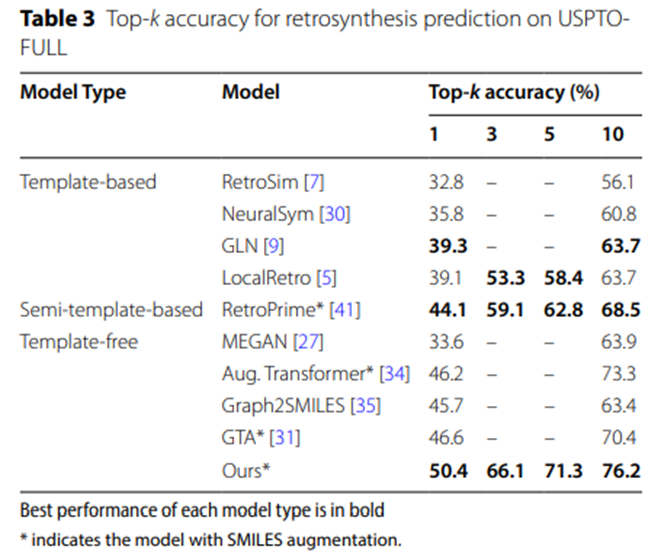
Les données expérimentales des tableaux 2 et 3 confirment en outre que sur les ensembles de données à plus grande échelle USPTO-MIT et USPTO-FULL, UAlign Le modèle surpasse les autres modèles de base par des avantages significatifs.
Tableau 3 : Précision Top-k de la prédiction rétrosynthétique sur USPTO-FULL
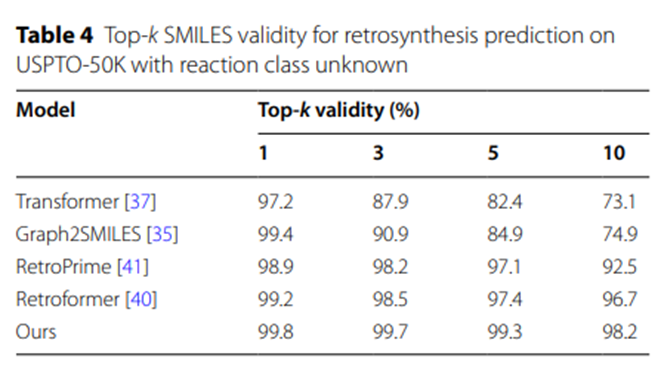
De plus, les résultats expérimentaux du tableau 4 montrent que, par rapport à d'autres modèles de prédiction rétrosynthétiques basés sur SMILES, les réactifs générés par le modèle UAlign. La séquence SMILES a une légitimité plus élevée.
Tableau 4 : Efficacité Top-k SMILES pour les prédictions rétrosynthétiques de classes de réactions inconnues sur USPTO-50K
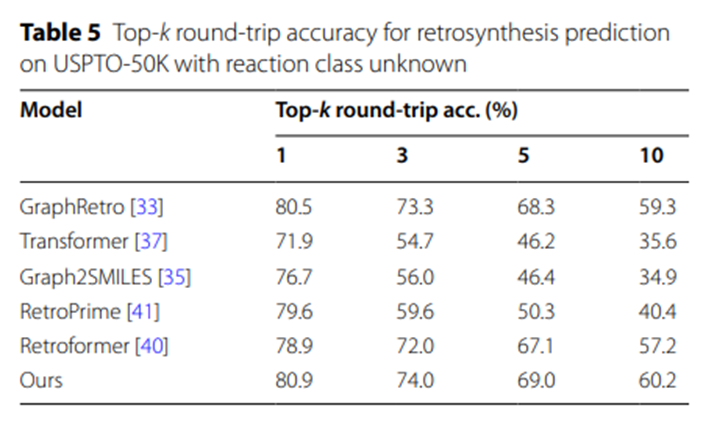
Les données expérimentales du tableau 5 mettent en évidence la capacité du modèle UAlign à générer des solutions de synthèse raisonnables et réalisables. La raison en est qu'une proportion relativement élevée des schémas de synthèse proposés par UAlign peuvent réussir la vérification du modèle de prédiction de réaction directe, c'est-à-dire que ces schémas peuvent être efficacement convertis en produits cibles donnés après les réactions chimiques correspondantes.
Tableau 5 : Précision aller-retour Top-k pour la prédiction de la rétrosynthèse avec des catégories de réactions inconnues sur USPTO-50K
Ces résultats expérimentaux vérifient non seulement l'efficacité et la précision du modèle UAlign dans la tâche de prédiction de la rétrosynthèse moléculaire, mais Il met également en évidence ses excellentes performances lors du traitement d'ensembles de données à grande échelle et ses avantages significatifs dans la génération de solutions de synthèse de haute qualité.
Afin de vérifier le potentiel d'application du modèle UAlign en production réelle, l'auteur a sélectionné de nouveaux médicaments approuvés par la Food and Drug Administration (FDA) des États-Unis au cours des deux dernières années comme cibles de synthèse. la synthèse a été obtenue avec succès. Les prédictions du modèle concernant les voies de synthèse de ces deux médicaments sont très cohérentes avec les voies enregistrées dans la littérature.
De plus, pour le troisième médicament, la voie de synthèse prédite par le modèle a également été reconnue réalisable par les experts du domaine de la chimie. Ces voies de synthèse couvrent non seulement une variété de types de réactions, mais incluent également des situations complexes telles que la synthèse de composés cycliques et les prédictions rétrosynthétiques en une seule étape impliquant plusieurs centres de réaction.
Les résultats expérimentaux ci-dessus prouvent pleinement que le modèle UAlign peut non seulement faire face à divers types de réactions, mais qu'il a également une grande valeur d'application dans la production réelle. Cela montre que le modèle UAlign présente une grande praticabilité et flexibilité dans le domaine de la prédiction de la rétrosynthèse moléculaire et peut fournir des solutions efficaces pour la synthèse de médicaments.

Perspectives futures
Grâce à ses excellentes performances et sa flexibilité, le modèle UAlign est tout à fait capable de servir de pierre angulaire à la construction d'un système rétrosynthétique en plusieurs étapes. Il peut être combiné avec divers algorithmes de recherche et technologies d’optimisation multi-objectifs pour former un système de planification de chemin rétrosynthétique efficace et intelligent.
En outre, l'auteur explore également activement l'intégration de l'algorithme UAlign avec un équipement matériel avancé pour créer un laboratoire automatisé sans pilote afin de promouvoir l'automatisation des processus de découverte et de synthèse de médicaments, apportant des changements révolutionnaires dans les domaines de la recherche chimique et du développement de médicaments. changement.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
 Utilisation de la fonction instr dans Oracle
Utilisation de la fonction instr dans Oracle
 cycle de vie des servlets
cycle de vie des servlets
 Comment récupérer l'historique des discussions WeChat supprimé
Comment récupérer l'historique des discussions WeChat supprimé
 Que faire en cas d'erreur de connexion
Que faire en cas d'erreur de connexion
 bootmgr est manquant et ne peut pas démarrer
bootmgr est manquant et ne peut pas démarrer
 amd240
amd240
 comment ouvrir le fichier php
comment ouvrir le fichier php
 Méthode de réparation des doutes sur la base de données
Méthode de réparation des doutes sur la base de données