Périphériques technologiques
Périphériques technologiques
 IA
IA
 36 fois supérieure à la résolution originale, des équipes de l'Université Beihang et de l'Université Tsinghua ont utilisé l'IA pour caractériser les tissus à haute résolution sur une plateforme omique multi-espace, publiée dans la sous-journal Nature
36 fois supérieure à la résolution originale, des équipes de l'Université Beihang et de l'Université Tsinghua ont utilisé l'IA pour caractériser les tissus à haute résolution sur une plateforme omique multi-espace, publiée dans la sous-journal Nature
36 fois supérieure à la résolution originale, des équipes de l'Université Beihang et de l'Université Tsinghua ont utilisé l'IA pour caractériser les tissus à haute résolution sur une plateforme omique multi-espace, publiée dans la sous-journal Nature

1. Introduction
L'omique spatiale a élargi la portée de l'analyse des classes moléculaires, mais de nombreuses techniques sont limitées par la résolution spatiale. Les méthodes informatiques existantes sont principalement ciblées sur les données transcriptomiques et manquent d’adaptabilité aux technologies émergentes d’omique spatiale.
2. Cadre soScope
Des chercheurs de l'Université Beihang et de l'Université Tsinghua ont proposé soScope, un cadre de génération unifié conçu pour améliorer la qualité et la résolution des données omiques spatiales.
3. Principe technique
soScope résume les informations tissulaires multimodales à partir des omiques, des relations spatiales et des images. Produisez des spectres omiques avec une résolution améliorée grâce à l'inférence conjointe des priorités de distribution et à une modélisation spécifique aux omiques.
4. Évaluation des performances
Les résultats de l'évaluation de soScope sur Visium, Xenium, spatial-CUT&Tag, slide-DNA/RNA-seq et d'autres plateformes montrent que :
- améliore les performances d'identification des structures intestinales et rénales
- Révélation de la structure fine du cœur embryonnaire
- Corrigé pour les biais d'échantillon et techniques
5 Applications étendues
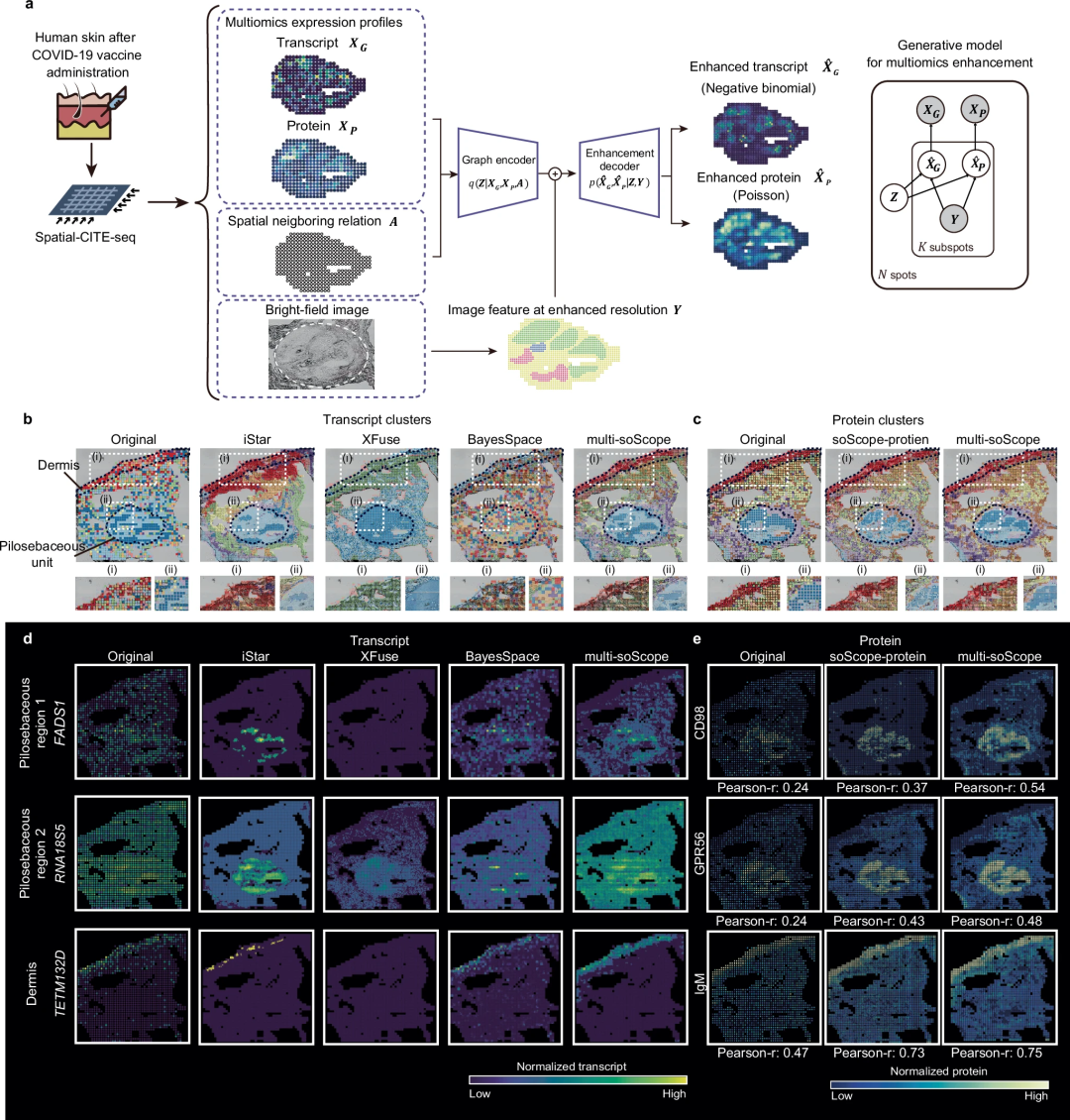
soScope a été étendu au séq spatial CITE et au séq spatial ATAC-ARN, tirant parti de l'omique croisée. références pour l’amélioration multi-Omic.
6. Conclusion
soScope fournit un outil polyvalent qui améliore l'utilisation de la technologie et des ressources spatiales omiques.
7. Référence
Cette recherche a été publiée dans « Nature Communications » le 2 août 2024 sous le titre « Caractérisation des tissus à une résolution améliorée sur les plateformes omiques spatiales avec modèle génératif profond ».

Les tissus sont composés de cellules avec différents états moléculaires et organisations spatiales. La technologie spatiale omique a fait des progrès significatifs ces dernières années, permettant l’analyse spatiale de diverses classes moléculaires tout en conservant le contexte spatial.
Défis et solutions
Malgré les premiers succès, la technologie de l'omique spatiale est toujours confrontée à deux défis majeurs :
- Les tissus congelés ou fixés au formol peuvent affecter l'état moléculaire et réduire la précision du séquençage.
- La plupart des techniques ont une résolution spatiale limitée, ce qui rend difficile la révélation de subtiles hétérogénéités dans la structure des tissus.
La technologie informatique peut améliorer la résolution des données omiques spatiales, mais la plupart des méthodes actuelles ne ciblent qu'une seule modalité tissulaire, ce qui rend difficile l'utilisation complète des informations multimodales.
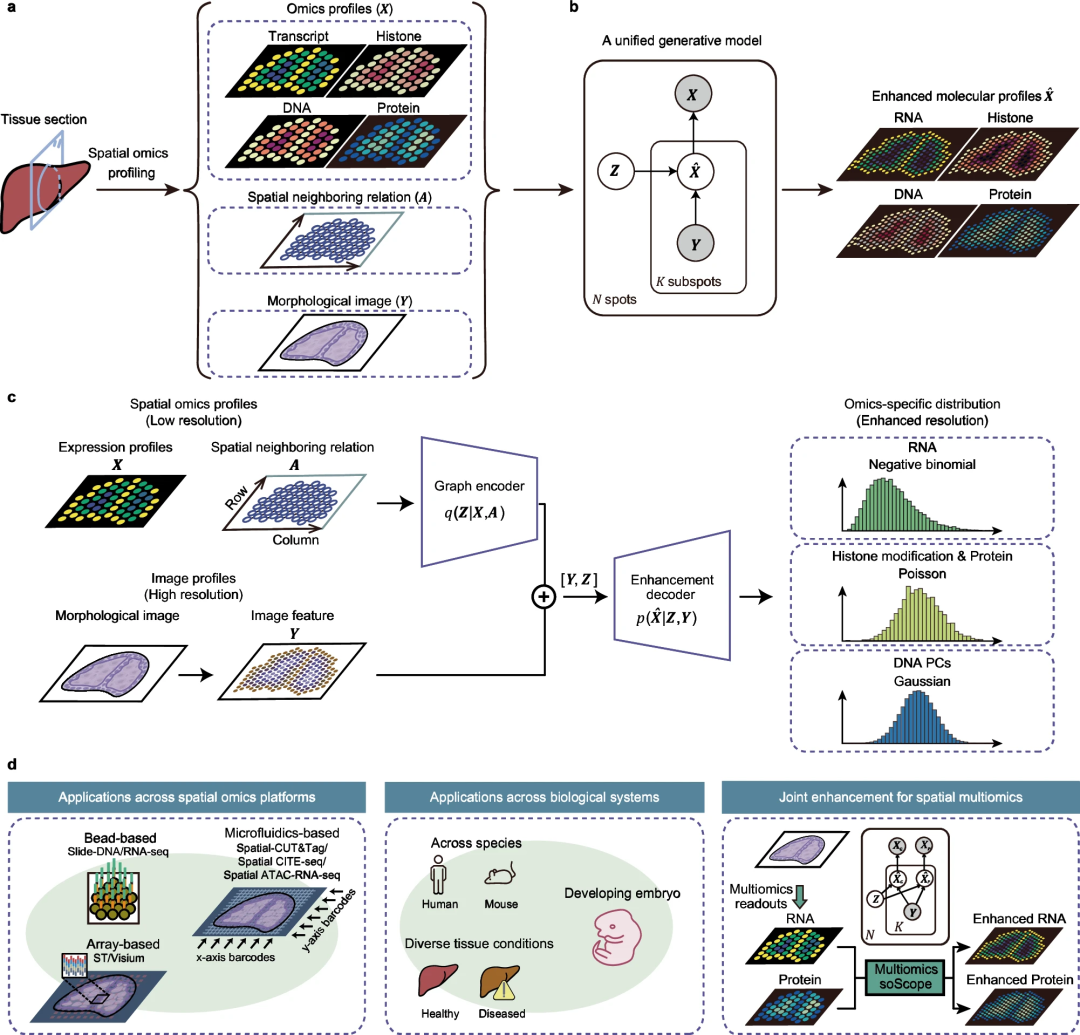
soScope : Améliorer la résolution spatiale et la qualité des données
Les équipes de recherche de l'Université Beihang et de l'Université Tsinghua présentent Spatiomic Scope (soScope), un cadre entièrement génératif qui simule des données au niveau du point provenant de différentes technologies d'omique spatiale. Le processus de génération de profil vise à améliorer leur résolution spatiale et la qualité des données.
soScope traite chaque point comme un ensemble de « sous-points » avec une résolution spatiale améliorée, dont les caractéristiques omiques sont liées à la localisation spatiale et aux modèles morphologiques. SoScope utilise ensuite un cadre d'apprentissage profond multimodal pour intégrer des profils omiques ponctuels, des relations spatiales et des images morphologiques à haute résolution et déduire conjointement des profils omiques à une résolution sub-spot. En sélectionnant des distributions spécifiques aux omiques, soScope peut modéliser et réduire avec précision la variation des différentes données omiques spatiales.
Caractéristiques de soScope :
- Outil unifié, combiné à des cartes tissulaires multimodales
- Cartes omiques améliorées (différentes classes moléculaires)
- Améliorer la résolution spatiale
- Réduire les changements inutiles
- Caractérisation Tissu complexe structures (indétectables à la résolution native)
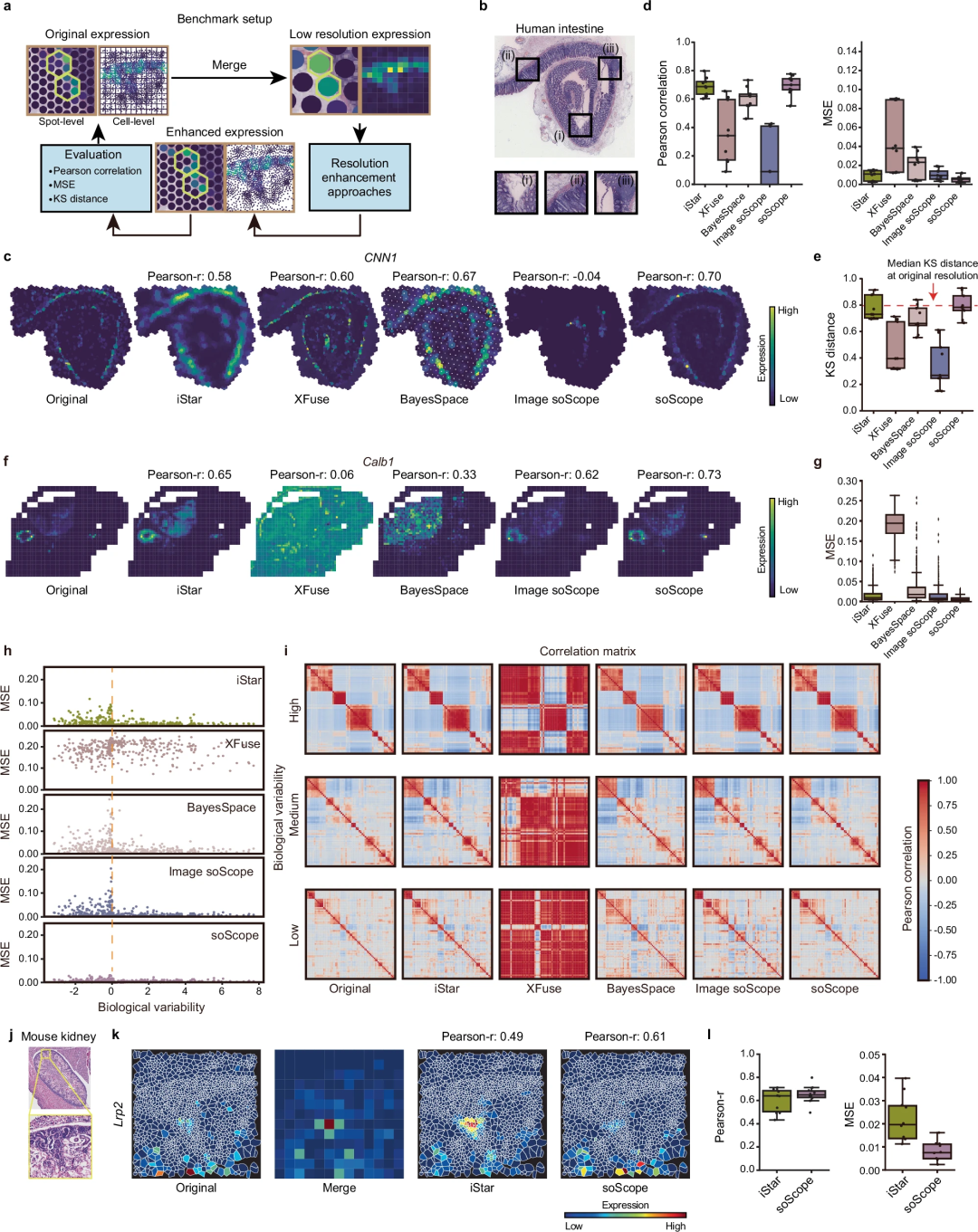
Illustration : Évaluation de soScope sur des ensembles de données transcriptomiques spatiales provenant de plusieurs tissus et plateformes. (Source : article)
L'équipe a évalué de manière approfondie l'efficacité et la généralisabilité de soScope pour plusieurs types moléculaires analysés par plusieurs techniques spatiales, notamment Visium, Xenium, spatial-CUT&Tag, slide-DNA-seq, slide-RNA-seq, spatial-CITE. -seq et ATAC-RNA-seq spatial.
Dans les tissus sains et malades, soScope améliore l'identification du domaine tissulaire, augmente la différentiabilité des marqueurs connus et corrige les données et les biais techniques. La méthode est capable de révéler des structures tissulaires plus fines jusqu’à 36 fois supérieures à la résolution originale. Il peut adapter efficacement les données spatiales multi-omiques pour améliorer simultanément les profils multi-omiques.
Les chercheurs notent qu'il existe plusieurs technologies omiques spatiales basées sur l'imagerie, telles que seqFISH, STARmap et MERFISH, qui peuvent directement réaliser une analyse spatiale à une résolution unicellulaire, mais au détriment d'un débit omique plus faible et de zones tissulaires plus petites. Bien que soScope fournisse des profils améliorés pour des sous-points ou des emplacements cellulaires prédéfinis, il peut ne pas atteindre une résolution subcellulaire.
Améliorez encore la résolution :
- Modifiez soScope pour inclure des données omiques unicellulaires appariées provenant du même tissu, fournissant des informations de résolution plus élevée pour l'inférence de sous-points.
- Intégrez des images H&E en entrée, qui peuvent être facilement annotées par des experts humains dans certaines études cliniques.
- Modifiez soScope pour intégrer des étiquettes humaines et guider l'inférence postérieure de manière semi-supervisée, améliorant ainsi la représentation latente et l'apprentissage du profil.
Réduire les coûts de calcul :
Pour des ensembles de données plus volumineux contenant plusieurs tranches contiguës du même organe, soScope peut :
- S'entraîner sur des données partielles.
- Appliquer sur les sections de tissus restantes.
Potentiel :
Avec l'expansion continue des ressources de données spatiales omiques et l'émergence de nouvelles technologies spatiales, les chercheurs pensent que soScope a le potentiel suivant :
- Un outil polyvalent.
- Exploitez pleinement les données spatiales omiques.
- Améliorez la compréhension des scientifiques des structures tissulaires complexes et des processus biologiques.
Lien papier :
https://www.nature.com/articles/s41467-024-50837-5
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!

Outils d'IA chauds

Undresser.AI Undress
Application basée sur l'IA pour créer des photos de nu réalistes

AI Clothes Remover
Outil d'IA en ligne pour supprimer les vêtements des photos.

Undress AI Tool
Images de déshabillage gratuites

Clothoff.io
Dissolvant de vêtements AI

AI Hentai Generator
Générez AI Hentai gratuitement.

Article chaud

Outils chauds

Bloc-notes++7.3.1
Éditeur de code facile à utiliser et gratuit

SublimeText3 version chinoise
Version chinoise, très simple à utiliser

Envoyer Studio 13.0.1
Puissant environnement de développement intégré PHP

Dreamweaver CS6
Outils de développement Web visuel

SublimeText3 version Mac
Logiciel d'édition de code au niveau de Dieu (SublimeText3)
 Repoussant les limites de la détection de défauts traditionnelle, « Defect Spectrum » permet pour la première fois une détection de défauts industriels d'une ultra haute précision et d'une sémantique riche.
Jul 26, 2024 pm 05:38 PM
Repoussant les limites de la détection de défauts traditionnelle, « Defect Spectrum » permet pour la première fois une détection de défauts industriels d'une ultra haute précision et d'une sémantique riche.
Jul 26, 2024 pm 05:38 PM
Dans la fabrication moderne, une détection précise des défauts est non seulement la clé pour garantir la qualité des produits, mais également la clé de l’amélioration de l’efficacité de la production. Cependant, les ensembles de données de détection de défauts existants manquent souvent de précision et de richesse sémantique requises pour les applications pratiques, ce qui rend les modèles incapables d'identifier des catégories ou des emplacements de défauts spécifiques. Afin de résoudre ce problème, une équipe de recherche de premier plan composée de l'Université des sciences et technologies de Hong Kong, Guangzhou et de Simou Technology a développé de manière innovante l'ensemble de données « DefectSpectrum », qui fournit une annotation à grande échelle détaillée et sémantiquement riche des défauts industriels. Comme le montre le tableau 1, par rapport à d'autres ensembles de données industrielles, l'ensemble de données « DefectSpectrum » fournit le plus grand nombre d'annotations de défauts (5 438 échantillons de défauts) et la classification de défauts la plus détaillée (125 catégories de défauts).
 L'Université de Tianjin et l'Université de Beihang sont profondément impliquées dans le projet « Cangjie » de Huawei et ont lancé le premier cadre de programmation d'agents d'IA « Cangqiong » basé sur des langages de programmation nationaux.
Jun 23, 2024 am 08:37 AM
L'Université de Tianjin et l'Université de Beihang sont profondément impliquées dans le projet « Cangjie » de Huawei et ont lancé le premier cadre de programmation d'agents d'IA « Cangqiong » basé sur des langages de programmation nationaux.
Jun 23, 2024 am 08:37 AM
Selon les informations de ce site du 22 juin, Huawei a présenté hier le langage de programmation auto-développé par Huawei, Cangjie, aux développeurs du monde entier. Il s'agit de la première apparition publique du langage de programmation Cangjie. Selon des enquêtes sur ce site, l'Université de Tianjin et l'Université d'aéronautique et d'astronautique de Pékin ont été profondément impliquées dans la recherche et le développement du « Cangjie » de Huawei. Université de Tianjin : compilateur de langage de programmation Cangjie L'équipe d'ingénierie logicielle du département d'intelligence et d'informatique de l'université de Tianjin s'est associée à l'équipe de Huawei Cangjie pour participer en profondeur à la recherche sur l'assurance qualité du compilateur de langage de programmation Cangjie. Selon certaines informations, le compilateur Cangjie est le logiciel de base en symbiose avec le langage de programmation Cangjie. Lors de la phase préparatoire du langage de programmation Cangjie, un compilateur de haute qualité qui lui correspond est devenu l'un des objectifs principaux. À mesure que le langage de programmation Cangjie évolue, le compilateur Cangjie est constamment mis à niveau et amélioré. Au cours des cinq dernières années, l'Université de Tianjin
 Le modèle de dialogue NVIDIA ChatQA a évolué vers la version 2.0, avec la longueur du contexte mentionnée à 128 Ko
Jul 26, 2024 am 08:40 AM
Le modèle de dialogue NVIDIA ChatQA a évolué vers la version 2.0, avec la longueur du contexte mentionnée à 128 Ko
Jul 26, 2024 am 08:40 AM
La communauté ouverte LLM est une époque où une centaine de fleurs fleurissent et s'affrontent. Vous pouvez voir Llama-3-70B-Instruct, QWen2-72B-Instruct, Nemotron-4-340B-Instruct, Mixtral-8x22BInstruct-v0.1 et bien d'autres. excellents interprètes. Cependant, par rapport aux grands modèles propriétaires représentés par le GPT-4-Turbo, les modèles ouverts présentent encore des lacunes importantes dans de nombreux domaines. En plus des modèles généraux, certains modèles ouverts spécialisés dans des domaines clés ont été développés, tels que DeepSeek-Coder-V2 pour la programmation et les mathématiques, et InternVL pour les tâches de langage visuel.
 Formation avec des millions de données cristallines pour résoudre le problème de la phase cristallographique, la méthode d'apprentissage profond PhAI est publiée dans Science
Aug 08, 2024 pm 09:22 PM
Formation avec des millions de données cristallines pour résoudre le problème de la phase cristallographique, la méthode d'apprentissage profond PhAI est publiée dans Science
Aug 08, 2024 pm 09:22 PM
Editeur | KX À ce jour, les détails structurels et la précision déterminés par cristallographie, des métaux simples aux grandes protéines membranaires, sont inégalés par aucune autre méthode. Cependant, le plus grand défi, appelé problème de phase, reste la récupération des informations de phase à partir d'amplitudes déterminées expérimentalement. Des chercheurs de l'Université de Copenhague au Danemark ont développé une méthode d'apprentissage en profondeur appelée PhAI pour résoudre les problèmes de phase cristalline. Un réseau neuronal d'apprentissage en profondeur formé à l'aide de millions de structures cristallines artificielles et de leurs données de diffraction synthétique correspondantes peut générer des cartes précises de densité électronique. L'étude montre que cette méthode de solution structurelle ab initio basée sur l'apprentissage profond peut résoudre le problème de phase avec une résolution de seulement 2 Angströms, ce qui équivaut à seulement 10 à 20 % des données disponibles à la résolution atomique, alors que le calcul ab initio traditionnel
 Google AI a remporté la médaille d'argent de l'Olympiade mathématique de l'OMI, le modèle de raisonnement mathématique AlphaProof a été lancé et l'apprentissage par renforcement est de retour.
Jul 26, 2024 pm 02:40 PM
Google AI a remporté la médaille d'argent de l'Olympiade mathématique de l'OMI, le modèle de raisonnement mathématique AlphaProof a été lancé et l'apprentissage par renforcement est de retour.
Jul 26, 2024 pm 02:40 PM
Pour l’IA, l’Olympiade mathématique n’est plus un problème. Jeudi, l'intelligence artificielle de Google DeepMind a réalisé un exploit : utiliser l'IA pour résoudre la vraie question de l'Olympiade mathématique internationale de cette année, l'OMI, et elle n'était qu'à un pas de remporter la médaille d'or. Le concours de l'OMI qui vient de se terminer la semaine dernière comportait six questions portant sur l'algèbre, la combinatoire, la géométrie et la théorie des nombres. Le système d'IA hybride proposé par Google a répondu correctement à quatre questions et a marqué 28 points, atteignant le niveau de la médaille d'argent. Plus tôt ce mois-ci, le professeur titulaire de l'UCLA, Terence Tao, venait de promouvoir l'Olympiade mathématique de l'IA (AIMO Progress Award) avec un prix d'un million de dollars. De manière inattendue, le niveau de résolution de problèmes d'IA s'était amélioré à ce niveau avant juillet. Posez les questions simultanément sur l'OMI. La chose la plus difficile à faire correctement est l'OMI, qui a la plus longue histoire, la plus grande échelle et la plus négative.
 Le point de vue de la nature : les tests de l'intelligence artificielle en médecine sont dans le chaos. Que faut-il faire ?
Aug 22, 2024 pm 04:37 PM
Le point de vue de la nature : les tests de l'intelligence artificielle en médecine sont dans le chaos. Que faut-il faire ?
Aug 22, 2024 pm 04:37 PM
Editeur | ScienceAI Sur la base de données cliniques limitées, des centaines d'algorithmes médicaux ont été approuvés. Les scientifiques se demandent qui devrait tester les outils et comment le faire au mieux. Devin Singh a vu un patient pédiatrique aux urgences subir un arrêt cardiaque alors qu'il attendait un traitement pendant une longue période, ce qui l'a incité à explorer l'application de l'IA pour réduire les temps d'attente. À l’aide des données de triage des salles d’urgence de SickKids, Singh et ses collègues ont construit une série de modèles d’IA pour fournir des diagnostics potentiels et recommander des tests. Une étude a montré que ces modèles peuvent accélérer les visites chez le médecin de 22,3 %, accélérant ainsi le traitement des résultats de près de 3 heures par patient nécessitant un examen médical. Cependant, le succès des algorithmes d’intelligence artificielle dans la recherche ne fait que le vérifier.
 Afin de fournir un nouveau système de référence et d'évaluation de questions-réponses scientifiques et complexes pour les grands modèles, l'UNSW, Argonne, l'Université de Chicago et d'autres institutions ont lancé conjointement le cadre SciQAG.
Jul 25, 2024 am 06:42 AM
Afin de fournir un nouveau système de référence et d'évaluation de questions-réponses scientifiques et complexes pour les grands modèles, l'UNSW, Argonne, l'Université de Chicago et d'autres institutions ont lancé conjointement le cadre SciQAG.
Jul 25, 2024 am 06:42 AM
L'ensemble de données ScienceAI Question Answering (QA) joue un rôle essentiel dans la promotion de la recherche sur le traitement du langage naturel (NLP). Des ensembles de données d'assurance qualité de haute qualité peuvent non seulement être utilisés pour affiner les modèles, mais également évaluer efficacement les capacités des grands modèles linguistiques (LLM), en particulier la capacité à comprendre et à raisonner sur les connaissances scientifiques. Bien qu’il existe actuellement de nombreux ensembles de données scientifiques d’assurance qualité couvrant la médecine, la chimie, la biologie et d’autres domaines, ces ensembles de données présentent encore certaines lacunes. Premièrement, le formulaire de données est relativement simple, et la plupart sont des questions à choix multiples. Elles sont faciles à évaluer, mais limitent la plage de sélection des réponses du modèle et ne peuvent pas tester pleinement la capacité du modèle à répondre aux questions scientifiques. En revanche, les questions et réponses ouvertes
 PRO | Pourquoi les grands modèles basés sur le MoE méritent-ils davantage d'attention ?
Aug 07, 2024 pm 07:08 PM
PRO | Pourquoi les grands modèles basés sur le MoE méritent-ils davantage d'attention ?
Aug 07, 2024 pm 07:08 PM
En 2023, presque tous les domaines de l’IA évoluent à une vitesse sans précédent. Dans le même temps, l’IA repousse constamment les limites technologiques de domaines clés tels que l’intelligence embarquée et la conduite autonome. Sous la tendance multimodale, le statut de Transformer en tant qu'architecture dominante des grands modèles d'IA sera-t-il ébranlé ? Pourquoi l'exploration de grands modèles basés sur l'architecture MoE (Mixture of Experts) est-elle devenue une nouvelle tendance dans l'industrie ? Les modèles de grande vision (LVM) peuvent-ils constituer une nouvelle avancée dans la vision générale ? ...Dans la newsletter des membres PRO 2023 de ce site publiée au cours des six derniers mois, nous avons sélectionné 10 interprétations spéciales qui fournissent une analyse approfondie des tendances technologiques et des changements industriels dans les domaines ci-dessus pour vous aider à atteindre vos objectifs dans le nouveau année. Cette interprétation provient de la Week50 2023