 Périphériques technologiques
Périphériques technologiques
 IA
IA
 En concevant des anticorps à partir de zéro, les équipes de Tencent et de l'Université de Pékin ont pré-entraîné de grands modèles de langage et les ont publiés dans la sous-journal Nature
En concevant des anticorps à partir de zéro, les équipes de Tencent et de l'Université de Pékin ont pré-entraîné de grands modèles de langage et les ont publiés dans la sous-journal Nature
En concevant des anticorps à partir de zéro, les équipes de Tencent et de l'Université de Pékin ont pré-entraîné de grands modèles de langage et les ont publiés dans la sous-journal Nature
 Éditeur | KX
Éditeur | KX
La technologie IA a fait de grands progrès dans l'aide à la conception d'anticorps. Cependant, la conception d’anticorps repose encore largement sur l’isolement des anticorps spécifiques de l’antigène à partir du sérum, ce qui constitue un processus long et gourmand en ressources.
Afin de résoudre ce problème, l'équipe de recherche du Tencent AI Lab, de la Peking University Shenzhen Graduate School et du Xijing Digestive Disease Hospital a proposé un grand modèle linguistique pré-entraîné de génération d'anticorps (PALM-H3) pour la génération de novo d'anticorps avec le spécificité de liaison à l'antigène requise L'anticorps artificiel unique CDRH3 réduit la dépendance aux anticorps naturels.
De plus, un modèle de prédiction de liaison antigène-anticorps de haute précision, A2binder, a été conçu pour faire correspondre la séquence de l'épitope de l'antigène avec la séquence de l'anticorps afin de prédire la spécificité et l'affinité de liaison.
En résumé, cette étude établit un cadre d’intelligence artificielle pour la génération et l’évaluation d’anticorps, qui a le potentiel d’accélérer considérablement le développement de médicaments à base d’anticorps.
Une recherche connexe intitulée « Génération de novo de l'anticorps CDRH3 du SRAS-CoV-2 avec un grand modèle de langage génératif pré-entraîné » a été publiée dans « Nature Communications » le 10 août.

Les médicaments anticorps, également connus sous le nom d'anticorps monoclonaux, jouent un rôle essentiel dans l'effet de la thérapie biologique. En imitant les actions du système immunitaire, ces médicaments peuvent cibler de manière sélective des agents pathogènes tels que les virus et les cellules cancéreuses. Les médicaments à base d'anticorps constituent une approche plus spécifique et plus efficace que les traitements traditionnels. Les médicaments à base d’anticorps ont donné des résultats positifs dans le traitement de diverses maladies.
Le développement de médicaments à base d'anticorps est un processus complexe qui consiste à isoler l'anticorps de sources animales, à l'humaniser et à optimiser son affinité. Mais le développement de médicaments à base d’anticorps repose encore largement sur les anticorps naturels.
Les données de séquence d'une protéine peuvent être considérées comme un langage, c'est pourquoi des modèles pré-entraînés à grande échelle dans le domaine du traitement du langage naturel (NLP) ont été utilisés pour apprendre les modèles de représentation des protéines. Une variété de modèles de langage protéique ont été développés. Cependant, générer des anticorps ayant une affinité élevée pour des épitopes spécifiques reste une tâche difficile en raison de la grande diversité des anticorps et de la rareté des données disponibles sur l’appariement antigène-anticorps.
Afin de relever les défis ci-dessus, l'équipe du Tencent AI Lab a proposé le modèle linguistique à grande échelle PALM-H3, pré-entraîné, pour la génération d'anticorps, afin d'optimiser et de générer la région 3 déterminant la complémentarité des chaînes lourdes (CDRH3), qui joue un rôle important. dans la spécificité et la diversité des anticorps joue un rôle essentiel.
Pour évaluer l'affinité des anticorps produits par PALM-H3 pour l'antigène, les chercheurs ont utilisé une combinaison de méthodes d'amarrage antigène-anticorps et basées sur l'IA.
Les chercheurs ont également développé A2binder pour évaluer l’affinité anticorps-antigène. A2binder permet des prédictions d’affinité précises et généralisables, même pour des antigènes inconnus.
Le cadre de PALM-H3 et A2Binder
Le flux de travail et le cadre de modèle de PALM-H3 et A2binder sont présentés dans la figure ci-dessous.
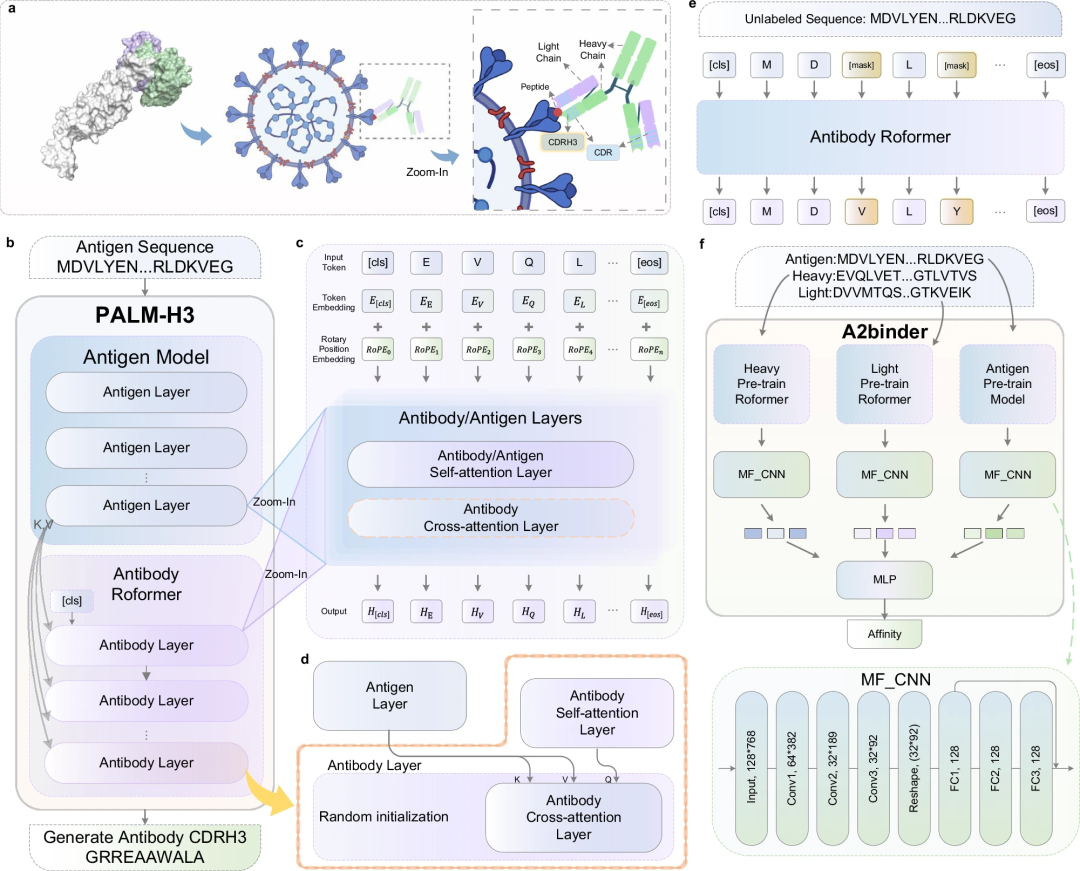
PALM-H3 est conçu pour générer de novo des séquences CDRH3 dans les anticorps. La région CDRH3 joue le rôle le plus important dans la détermination de la spécificité de liaison des anticorps pour des séquences antigéniques spécifiques. PALM-H3 est un modèle de type transformateur qui utilise un modèle d'antigène basé sur ESM2 comme codeur et un anticorps Roformer comme décodeur. L’étude a également construit A2binder pour prédire l’affinité de liaison des anticorps générés artificiellement.
La construction de PALM-H3 et d'A2binder comprenait trois étapes : Tout d'abord, les chercheurs ont pré-entraîné deux modèles Roformer sur des séquences de chaînes lourdes et de chaînes légères d'anticorps non appariées, respectivement. Ensuite, A2binder a été construit sur la base d’ESM2 pré-entraîné, de Roformer à chaîne lourde d’anticorps et de Roformer à chaîne légère d’anticorps, et formé à l’aide de données d’affinité appariées. Enfin, PALM-H3 a été construit à l’aide d’ESM2 pré-entraîné et de Roformer de chaîne lourde d’anticorps et formé sur des données antigène-CDRH3 appariées pour générer CDRH3 de novo.
A2binder peut prédire avec précision la probabilité de liaison antigène-anticorps et l'affinité
Les performances d'A2binder ont été évaluées en comparant sa capacité à prédire l'affinité avec plusieurs méthodes de base.
A2binder fonctionne bien sur l'ensemble de données d'affinité, en partie grâce au pré-entraînement sur les séquences d'anticorps, qui permet à A2binder d'apprendre les modèles uniques présents dans ces séquences.
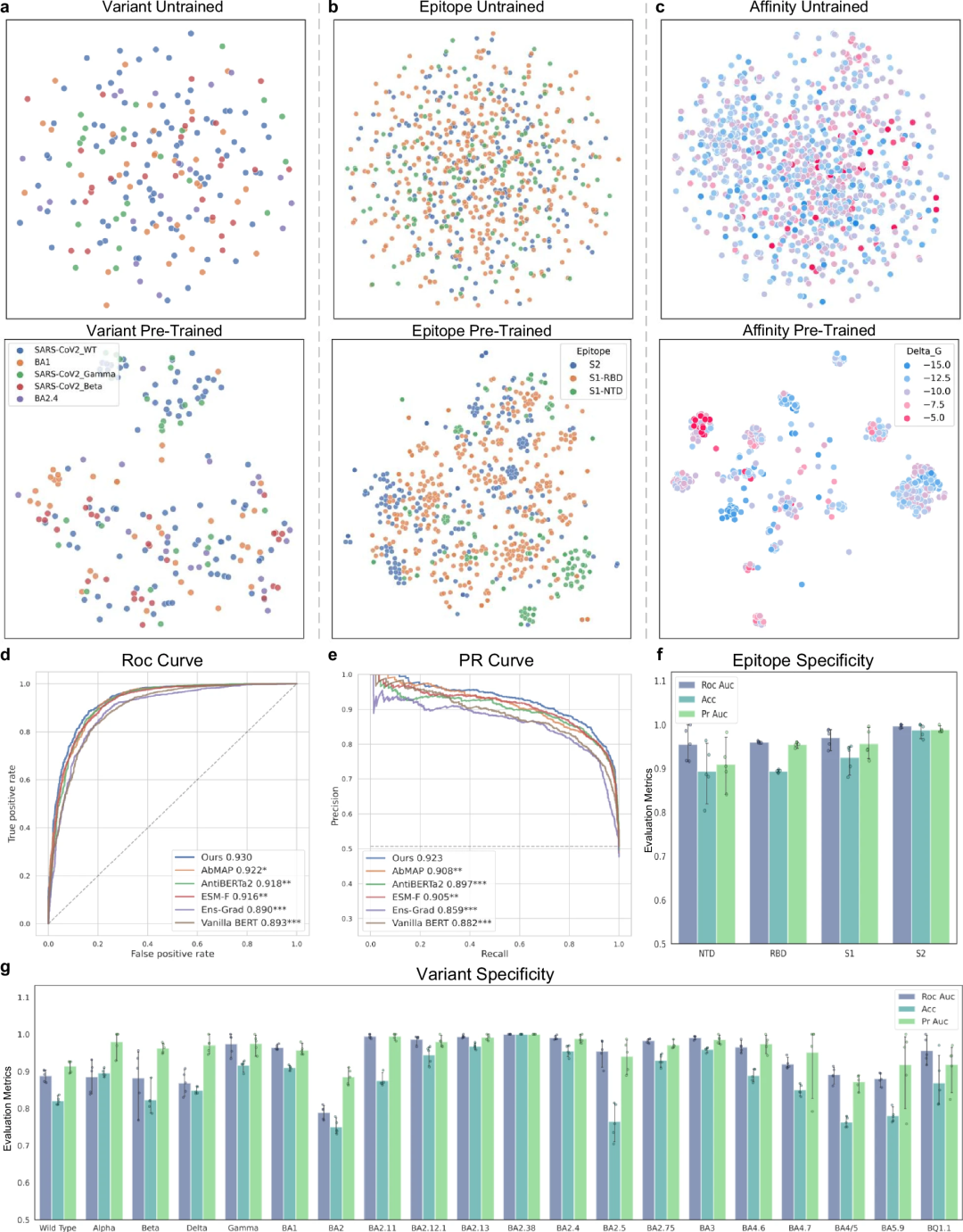
Die Ergebnisse zeigen, dass A2binder bei allen Antigen-Antikörper-Affinitätsvorhersagedatensätzen eine bessere Leistung erbringt als das Basismodell ESM-F (letzteres hat das gleiche Framework, aber das vorab trainierte Modell wird durch ESM2 ersetzt). zeigt, dass ein Vortraining mit Antikörpersequenzen für verwandte nachgelagerte Aufgaben von Vorteil sein kann.
Um die Leistung des Modells bei der Vorhersage von Affinitätswerten zu bewerten, verwendeten die Forscher auch zwei Datensätze, 14H und 14L, die Affinitätswertbezeichnungen enthalten.
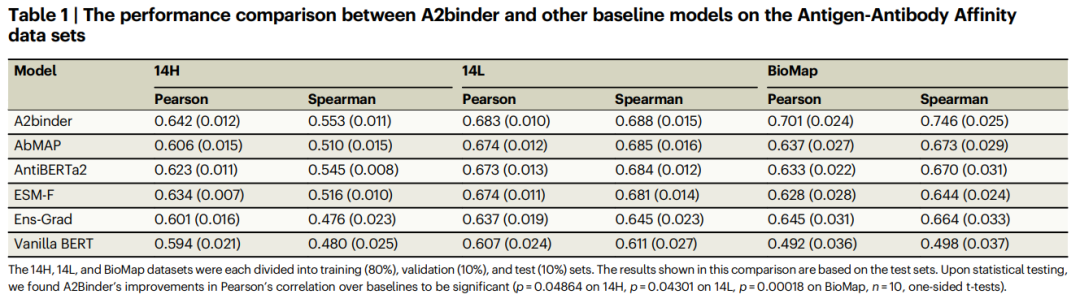
A2binder übertrifft alle Basismodelle sowohl hinsichtlich der Pearson-Korrelations- als auch der Spearman-Korrelationsmetriken. A2binder erreicht eine Pearson-Korrelation von 0,642 für den 14H-Datensatz (eine Verbesserung von 3 %) und 0,683 für den 14L-Datensatz (eine Verbesserung von 1 %).
Allerdings sank die Leistung von A2binder und anderen Basismodellen bei den 14H- und 14L-Datensätzen im Vergleich zu anderen Datensätzen leicht. Diese Beobachtung steht im Einklang mit früheren Studien.
PALM-H3 zeichnet sich durch die Erzeugung von Antikörpern mit hoher Bindungswahrscheinlichkeit aus.
Forscher untersuchten die Unterschiede zwischen den von PALM-H3 produzierten Antikörpern und natürlichen Antikörpern. Es wurde festgestellt, dass sich ihre Sequenzen erheblich unterscheiden, die Bindungswahrscheinlichkeiten der produzierten Antikörper wurden jedoch durch diese Unterschiede nicht wesentlich beeinflusst. Gleichzeitig führen ihre strukturellen Unterschiede zu einer Verringerung der Bindungsaffinität. Diese Ergebnisse stehen im Einklang mit früheren Studien zur Netzwerkanalyse von Antikörperbibliotheken und zur Erzeugung funktioneller Proteinsequenzen.
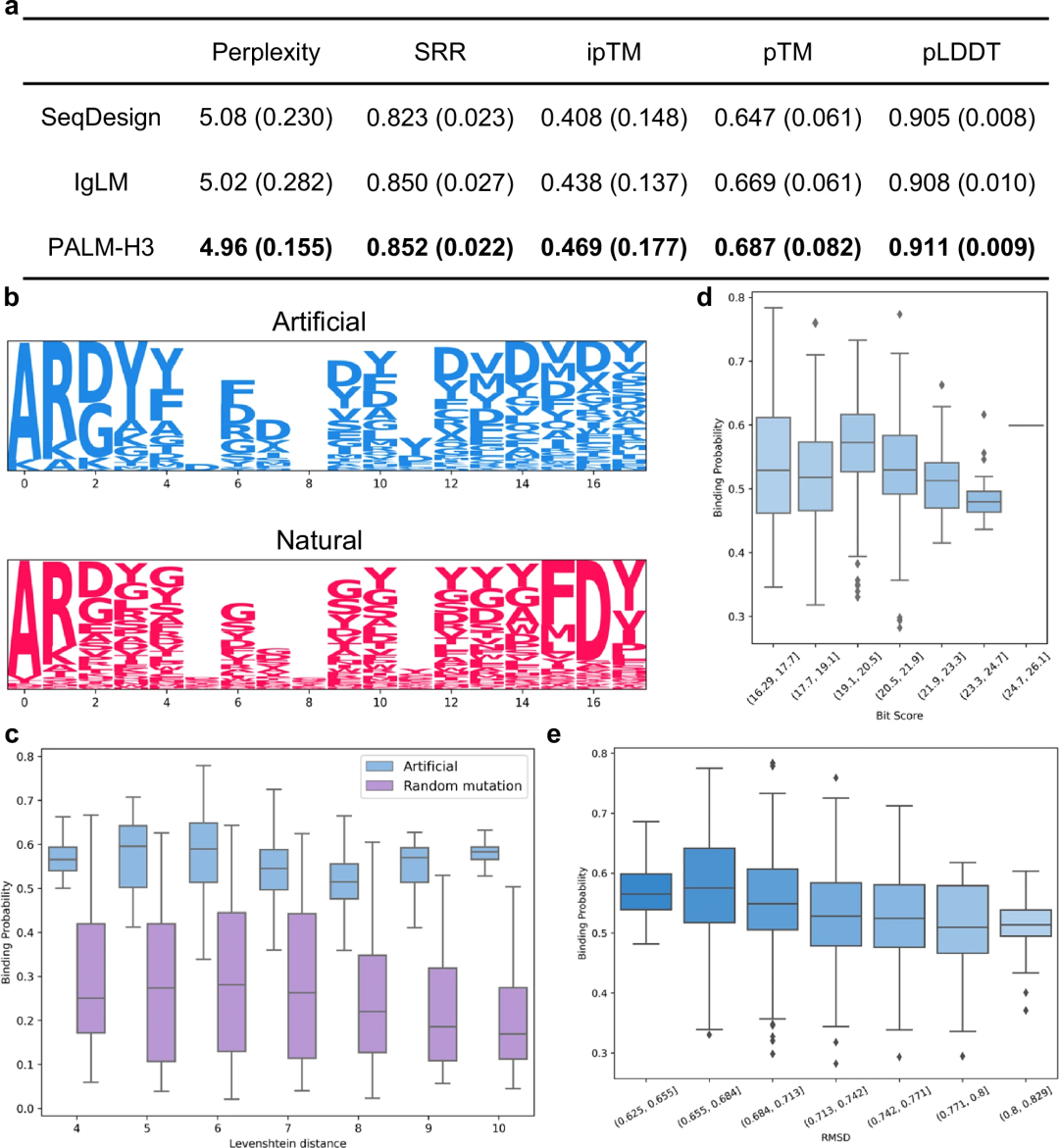
Insgesamt zeigen die Ergebnisse, dass PALM-H3 im Gegensatz zu natürlichen Antikörpern in der Lage ist, vielfältige Antikörpersequenzen mit hohen Bindungsaffinitäten zu erzeugen.
Darüber hinaus überprüften Forscher die Leistung von PALM-H3 durch ClusPro und SnugDock. PALM-H3 ist in der Lage, Antikörper gegen die CDRH3-Sequenz des SARS-CoV-2 HR2-Region-stabilisierenden Peptids zu erzeugen. Es wurde eine neuartige CDRH3-Sequenz generiert und bestätigt, dass die generierte Sequenz GRREAAWALA im Vergleich zur nativen CDHR3-Sequenz GKAAGTFDS ein verbessertes Targeting antigenstabilisierender Peptide aufweist.
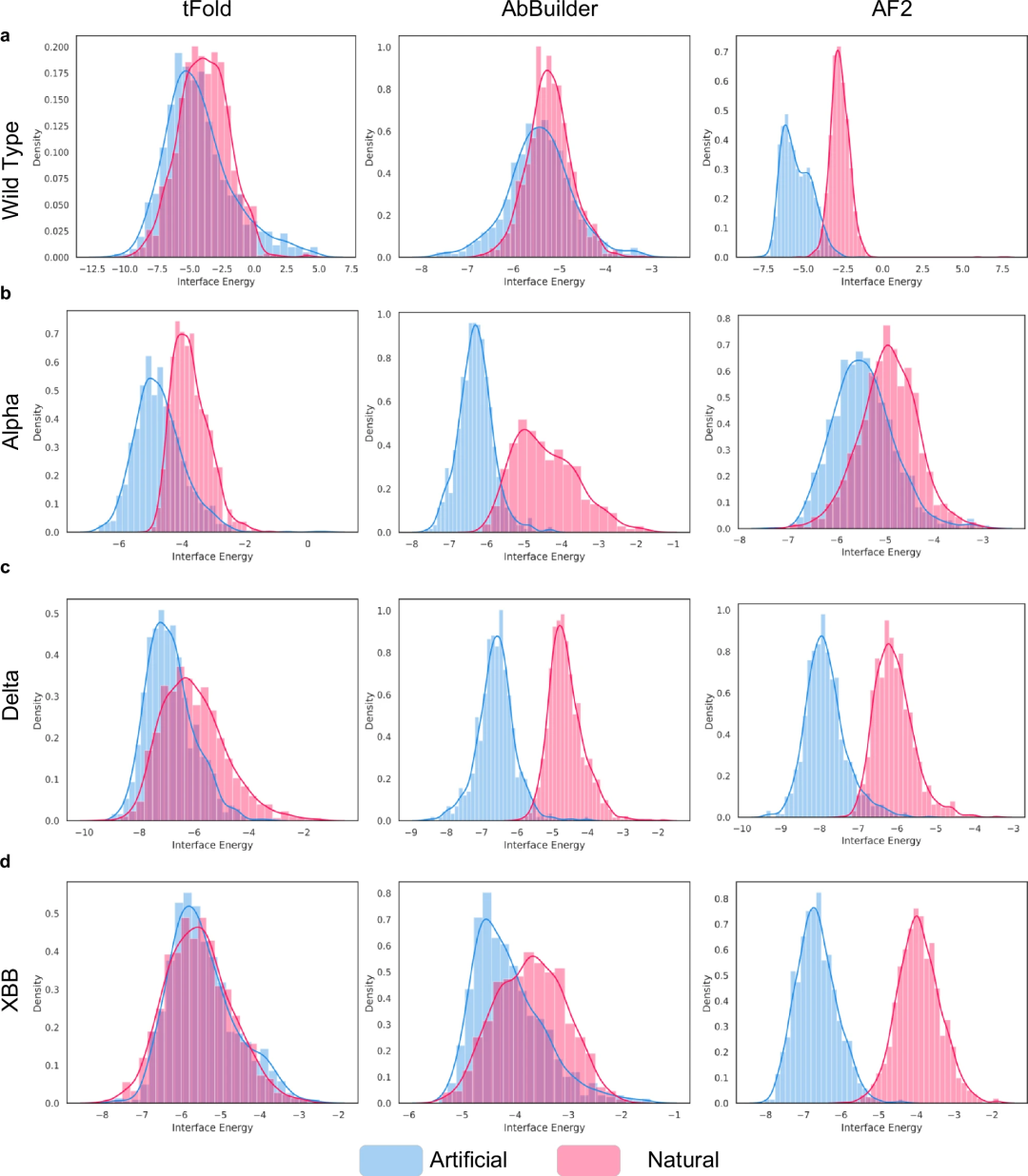
Darüber hinaus ist PALM-H3 in der Lage, Antikörper mit höherer Affinität gegen die neu auftretende SARS-CoV-2-Variante XBB CDRH3-Sequenz zu erzeugen. Die resultierende Sequenz AKDSRTSPLRLDYS hat eine stärkere Affinität zu XBB als ihre Quelle ASEVLDNLRDGYNF.
Darüber hinaus überwindet PALM-H3 nicht nur die lokalen optimalen Fallstricke, mit denen traditionelle sequentielle Mutationsstrategien konfrontiert sind, sondern erzeugt im Vergleich zum E-EVO-Ansatz auch Antikörper mit höherer Antigenbindungsaffinität. Dies unterstreicht die Vorteile von PALM-H3 beim Antikörperdesign, das eine effizientere Erkundung des Sequenzraums und die Erzeugung hochaffiner Binder ermöglicht, die auf spezifische Epitope abzielen.
In-vitro-Experimente
Darüber hinaus führten die Forscher auch In-vitro-Experimente durch, darunter Western Blot, Oberflächenplasmonresonanzanalyse und Pseudovirus-Neutralisationstests, die einen wichtigen Nachweis für die Wirksamkeit der von PALM-H3 entwickelten Antikörper lieferten.
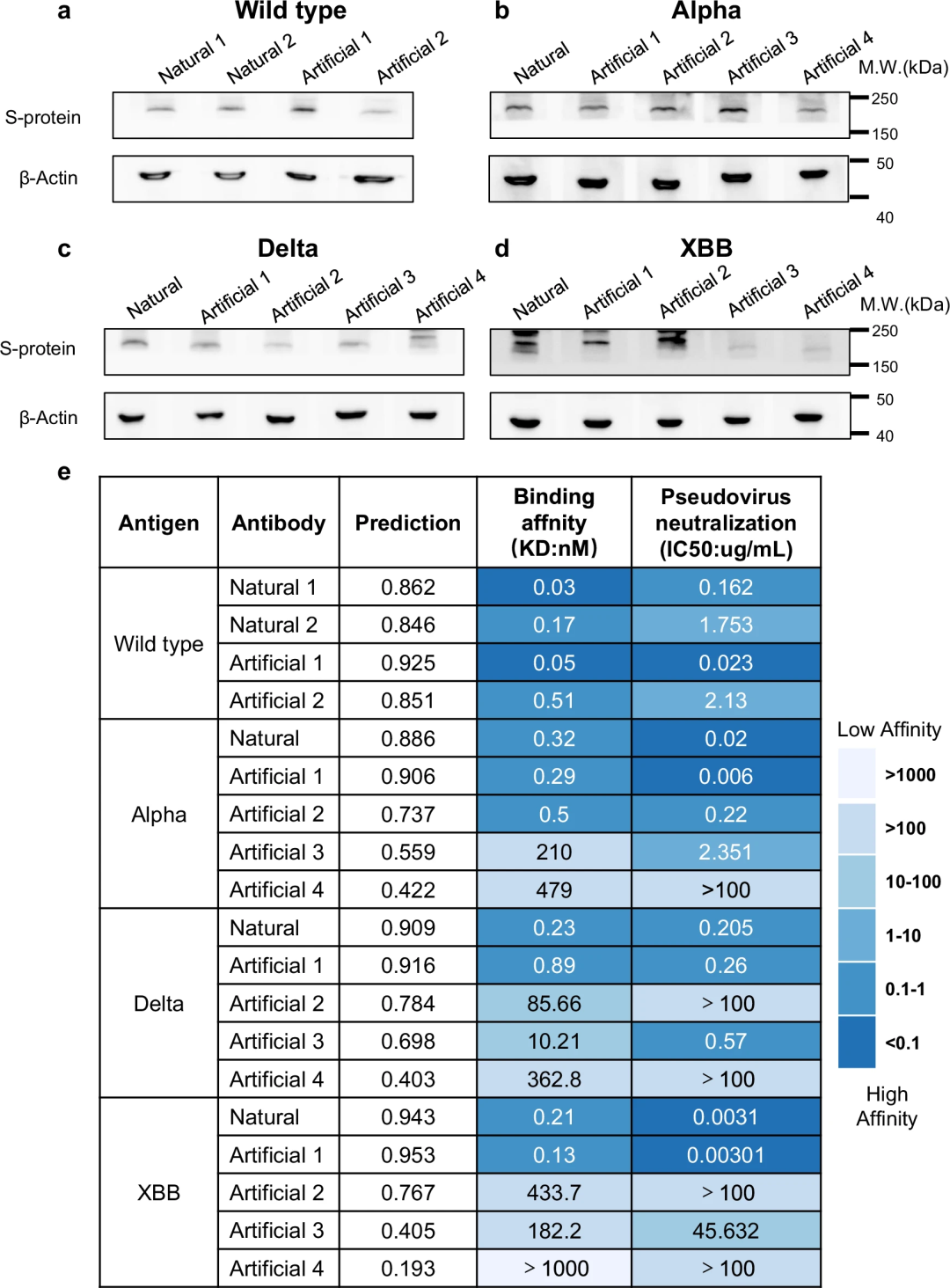
Zwei von PALM-H3 erzeugte Antikörper gegen die Spike-Proteine der SARS-CoV-2-Wildtyp-, Alpha-, Delta- und XBB-Varianten erzielten in diesen Experimenten höhere Bindungsaffinitäten und eine neutralisierende Wirkung als native Antikörper. Die robusten empirischen Ergebnisse dieser Nasslaborexperimente ergänzen rechnerische Vorhersagen und Analysen und bestätigen die Fähigkeit von PALM-H3 und A2binder, wirksame Antikörper mit hoher Spezifität und Affinität für bekannte und neue Antigene zu erzeugen und auszuwählen.
Zusammenfassend lässt sich sagen, dass das vorgeschlagene PALM-H3 die Fähigkeit eines groß angelegten Antikörper-Vortrainings und die Wirksamkeit der globalen Merkmalsfusion integriert, was zu einer hervorragenden Affinitätsvorhersageleistung und der Fähigkeit zur Entwicklung hochaffiner Antikörper führt. Darüber hinaus machen die direkte Sequenzgenerierung und die interpretierbare Gewichtsvisualisierung es zu einem effizienten und interpretierbaren Werkzeug für die Entwicklung hochaffiner Antikörper.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!

Outils d'IA chauds

Undresser.AI Undress
Application basée sur l'IA pour créer des photos de nu réalistes

AI Clothes Remover
Outil d'IA en ligne pour supprimer les vêtements des photos.

Undress AI Tool
Images de déshabillage gratuites

Clothoff.io
Dissolvant de vêtements AI

Video Face Swap
Échangez les visages dans n'importe quelle vidéo sans effort grâce à notre outil d'échange de visage AI entièrement gratuit !

Article chaud

Outils chauds

Bloc-notes++7.3.1
Éditeur de code facile à utiliser et gratuit

SublimeText3 version chinoise
Version chinoise, très simple à utiliser

Envoyer Studio 13.0.1
Puissant environnement de développement intégré PHP

Dreamweaver CS6
Outils de développement Web visuel

SublimeText3 version Mac
Logiciel d'édition de code au niveau de Dieu (SublimeText3)

Sujets chauds
 1673
1673
 14
14
 1428
1428
 52
52
 1333
1333
 25
25
 1278
1278
 29
29
 1257
1257
 24
24
 Repoussant les limites de la détection de défauts traditionnelle, « Defect Spectrum » permet pour la première fois une détection de défauts industriels d'une ultra haute précision et d'une sémantique riche.
Jul 26, 2024 pm 05:38 PM
Repoussant les limites de la détection de défauts traditionnelle, « Defect Spectrum » permet pour la première fois une détection de défauts industriels d'une ultra haute précision et d'une sémantique riche.
Jul 26, 2024 pm 05:38 PM
Dans la fabrication moderne, une détection précise des défauts est non seulement la clé pour garantir la qualité des produits, mais également la clé de l’amélioration de l’efficacité de la production. Cependant, les ensembles de données de détection de défauts existants manquent souvent de précision et de richesse sémantique requises pour les applications pratiques, ce qui rend les modèles incapables d'identifier des catégories ou des emplacements de défauts spécifiques. Afin de résoudre ce problème, une équipe de recherche de premier plan composée de l'Université des sciences et technologies de Hong Kong, Guangzhou et de Simou Technology a développé de manière innovante l'ensemble de données « DefectSpectrum », qui fournit une annotation à grande échelle détaillée et sémantiquement riche des défauts industriels. Comme le montre le tableau 1, par rapport à d'autres ensembles de données industrielles, l'ensemble de données « DefectSpectrum » fournit le plus grand nombre d'annotations de défauts (5 438 échantillons de défauts) et la classification de défauts la plus détaillée (125 catégories de défauts).
 Formation avec des millions de données cristallines pour résoudre le problème de la phase cristallographique, la méthode d'apprentissage profond PhAI est publiée dans Science
Aug 08, 2024 pm 09:22 PM
Formation avec des millions de données cristallines pour résoudre le problème de la phase cristallographique, la méthode d'apprentissage profond PhAI est publiée dans Science
Aug 08, 2024 pm 09:22 PM
Editeur | KX À ce jour, les détails structurels et la précision déterminés par cristallographie, des métaux simples aux grandes protéines membranaires, sont inégalés par aucune autre méthode. Cependant, le plus grand défi, appelé problème de phase, reste la récupération des informations de phase à partir d'amplitudes déterminées expérimentalement. Des chercheurs de l'Université de Copenhague au Danemark ont développé une méthode d'apprentissage en profondeur appelée PhAI pour résoudre les problèmes de phase cristalline. Un réseau neuronal d'apprentissage en profondeur formé à l'aide de millions de structures cristallines artificielles et de leurs données de diffraction synthétique correspondantes peut générer des cartes précises de densité électronique. L'étude montre que cette méthode de solution structurelle ab initio basée sur l'apprentissage profond peut résoudre le problème de phase avec une résolution de seulement 2 Angströms, ce qui équivaut à seulement 10 à 20 % des données disponibles à la résolution atomique, alors que le calcul ab initio traditionnel
 Le modèle de dialogue NVIDIA ChatQA a évolué vers la version 2.0, avec la longueur du contexte mentionnée à 128 Ko
Jul 26, 2024 am 08:40 AM
Le modèle de dialogue NVIDIA ChatQA a évolué vers la version 2.0, avec la longueur du contexte mentionnée à 128 Ko
Jul 26, 2024 am 08:40 AM
La communauté ouverte LLM est une époque où une centaine de fleurs fleurissent et s'affrontent. Vous pouvez voir Llama-3-70B-Instruct, QWen2-72B-Instruct, Nemotron-4-340B-Instruct, Mixtral-8x22BInstruct-v0.1 et bien d'autres. excellents interprètes. Cependant, par rapport aux grands modèles propriétaires représentés par le GPT-4-Turbo, les modèles ouverts présentent encore des lacunes importantes dans de nombreux domaines. En plus des modèles généraux, certains modèles ouverts spécialisés dans des domaines clés ont été développés, tels que DeepSeek-Coder-V2 pour la programmation et les mathématiques, et InternVL pour les tâches de langage visuel.
 Google AI a remporté la médaille d'argent de l'Olympiade mathématique de l'OMI, le modèle de raisonnement mathématique AlphaProof a été lancé et l'apprentissage par renforcement est de retour.
Jul 26, 2024 pm 02:40 PM
Google AI a remporté la médaille d'argent de l'Olympiade mathématique de l'OMI, le modèle de raisonnement mathématique AlphaProof a été lancé et l'apprentissage par renforcement est de retour.
Jul 26, 2024 pm 02:40 PM
Pour l’IA, l’Olympiade mathématique n’est plus un problème. Jeudi, l'intelligence artificielle de Google DeepMind a réalisé un exploit : utiliser l'IA pour résoudre la vraie question de l'Olympiade mathématique internationale de cette année, l'OMI, et elle n'était qu'à un pas de remporter la médaille d'or. Le concours de l'OMI qui vient de se terminer la semaine dernière comportait six questions portant sur l'algèbre, la combinatoire, la géométrie et la théorie des nombres. Le système d'IA hybride proposé par Google a répondu correctement à quatre questions et a marqué 28 points, atteignant le niveau de la médaille d'argent. Plus tôt ce mois-ci, le professeur titulaire de l'UCLA, Terence Tao, venait de promouvoir l'Olympiade mathématique de l'IA (AIMO Progress Award) avec un prix d'un million de dollars. De manière inattendue, le niveau de résolution de problèmes d'IA s'était amélioré à ce niveau avant juillet. Posez les questions simultanément sur l'OMI. La chose la plus difficile à faire correctement est l'OMI, qui a la plus longue histoire, la plus grande échelle et la plus négative.
 PRO | Pourquoi les grands modèles basés sur le MoE méritent-ils davantage d'attention ?
Aug 07, 2024 pm 07:08 PM
PRO | Pourquoi les grands modèles basés sur le MoE méritent-ils davantage d'attention ?
Aug 07, 2024 pm 07:08 PM
En 2023, presque tous les domaines de l’IA évoluent à une vitesse sans précédent. Dans le même temps, l’IA repousse constamment les limites technologiques de domaines clés tels que l’intelligence embarquée et la conduite autonome. Sous la tendance multimodale, le statut de Transformer en tant qu'architecture dominante des grands modèles d'IA sera-t-il ébranlé ? Pourquoi l'exploration de grands modèles basés sur l'architecture MoE (Mixture of Experts) est-elle devenue une nouvelle tendance dans l'industrie ? Les modèles de grande vision (LVM) peuvent-ils constituer une nouvelle avancée dans la vision générale ? ...Dans la newsletter des membres PRO 2023 de ce site publiée au cours des six derniers mois, nous avons sélectionné 10 interprétations spéciales qui fournissent une analyse approfondie des tendances technologiques et des changements industriels dans les domaines ci-dessus pour vous aider à atteindre vos objectifs dans le nouveau année. Cette interprétation provient de la Week50 2023
 Afin de fournir un nouveau système de référence et d'évaluation de questions-réponses scientifiques et complexes pour les grands modèles, l'UNSW, Argonne, l'Université de Chicago et d'autres institutions ont lancé conjointement le cadre SciQAG.
Jul 25, 2024 am 06:42 AM
Afin de fournir un nouveau système de référence et d'évaluation de questions-réponses scientifiques et complexes pour les grands modèles, l'UNSW, Argonne, l'Université de Chicago et d'autres institutions ont lancé conjointement le cadre SciQAG.
Jul 25, 2024 am 06:42 AM
L'ensemble de données ScienceAI Question Answering (QA) joue un rôle essentiel dans la promotion de la recherche sur le traitement du langage naturel (NLP). Des ensembles de données d'assurance qualité de haute qualité peuvent non seulement être utilisés pour affiner les modèles, mais également évaluer efficacement les capacités des grands modèles linguistiques (LLM), en particulier la capacité à comprendre et à raisonner sur les connaissances scientifiques. Bien qu’il existe actuellement de nombreux ensembles de données scientifiques d’assurance qualité couvrant la médecine, la chimie, la biologie et d’autres domaines, ces ensembles de données présentent encore certaines lacunes. Premièrement, le formulaire de données est relativement simple, et la plupart sont des questions à choix multiples. Elles sont faciles à évaluer, mais limitent la plage de sélection des réponses du modèle et ne peuvent pas tester pleinement la capacité du modèle à répondre aux questions scientifiques. En revanche, les questions et réponses ouvertes
 Le taux de précision atteint 60,8 %. Le modèle de prédiction de rétrosynthèse chimique de l'Université du Zhejiang basé sur Transformer a été publié dans la sous-journal Nature.
Aug 06, 2024 pm 07:34 PM
Le taux de précision atteint 60,8 %. Le modèle de prédiction de rétrosynthèse chimique de l'Université du Zhejiang basé sur Transformer a été publié dans la sous-journal Nature.
Aug 06, 2024 pm 07:34 PM
Editeur | KX La rétrosynthèse est une tâche essentielle dans la découverte de médicaments et la synthèse organique, et l'IA est de plus en plus utilisée pour accélérer le processus. Les méthodes d’IA existantes ont des performances insatisfaisantes et une diversité limitée. En pratique, les réactions chimiques provoquent souvent des modifications moléculaires locales, avec un chevauchement considérable entre les réactifs et les produits. Inspirée par cela, l'équipe de Hou Tingjun de l'Université du Zhejiang a proposé de redéfinir la prédiction rétrosynthétique en une seule étape en tant que tâche d'édition de chaînes moléculaires, en affinant de manière itérative la chaîne moléculaire cible pour générer des composés précurseurs. Et un modèle rétrosynthétique basé sur l'édition, EditRetro, est proposé, qui permet d'obtenir des prédictions diverses et de haute qualité. Des expériences approfondies montrent que le modèle atteint d'excellentes performances sur l'ensemble de données de référence standard USPTO-50 K, avec une précision top 1 de 60,8 %.
 Le point de vue de la nature : les tests de l'intelligence artificielle en médecine sont dans le chaos. Que faut-il faire ?
Aug 22, 2024 pm 04:37 PM
Le point de vue de la nature : les tests de l'intelligence artificielle en médecine sont dans le chaos. Que faut-il faire ?
Aug 22, 2024 pm 04:37 PM
Editeur | ScienceAI Sur la base de données cliniques limitées, des centaines d'algorithmes médicaux ont été approuvés. Les scientifiques se demandent qui devrait tester les outils et comment le faire au mieux. Devin Singh a vu un patient pédiatrique aux urgences subir un arrêt cardiaque alors qu'il attendait un traitement pendant une longue période, ce qui l'a incité à explorer l'application de l'IA pour réduire les temps d'attente. À l’aide des données de triage des salles d’urgence de SickKids, Singh et ses collègues ont construit une série de modèles d’IA pour fournir des diagnostics potentiels et recommander des tests. Une étude a montré que ces modèles peuvent accélérer les visites chez le médecin de 22,3 %, accélérant ainsi le traitement des résultats de près de 3 heures par patient nécessitant un examen médical. Cependant, le succès des algorithmes d’intelligence artificielle dans la recherche ne fait que le vérifier.



