

Dans la vie, c'est facile d'allumer une lampe ou de régler les lumières. Mais les systèmes permettant un contrôle similaire des fonctions biomoléculaires sont complexes et mal compris.
En biologie, les fonctions des protéines sont activées et désactivées de manière complexe, et la régulation allostérique est l'un des mécanismes de régulation biologique importants, crucial pour un métabolisme sain et une signalisation cellulaire. Mais la création d’allostérie dans des systèmes protéiques synthétiques a toujours présenté des défis importants.
Récemment, l’équipe de David Baker de l’Université de Washington a conçu une protéine capable de basculer entre l’assemblage et le démontage de manière fiable et précise grâce à un contrôle allostérique. En utilisant l’IA pour concevoir de nouvelles protéines qui n’existent pas dans la nature, les chercheurs ont mis au point plusieurs arrangements dynamiques de protéines.
David Baker a déclaré : "En concevant des protéines qui peuvent être assemblées et désassemblées sur commande, nous ouvrons la voie à de futures biotechnologies qui pourraient rivaliser avec la complexité de la nature."
Arvind Pillai, premier auteur et auteur correspondant de l'article. L'une des principales innovations de cette recherche est la conception d'assemblages protéiques capables de basculer entre différents états oligomères, tels que des dimères, des anneaux et des cages, en réponse à des molécules effectrices", a déclaré le Dr. La capacité de contrôler à distance la structure des protéines ouvre la voie à la possibilité de développer des biomatériaux adaptatifs et des systèmes d'administration de médicaments. "Nature" qualifie cela de "percée étonnante dans la conception des protéines".
La recherche pertinente s'intitulait "Conception de novo d'assemblages de protéines allostériquement commutables" et a été publiée dans "Nature" le 14 août.
Allostère et conception de novo
.
Conception de novoà partir de zéro en couplant des modules de charnière commutables et des interfaces protéiques avec des corps rigides.
Stratégie de conception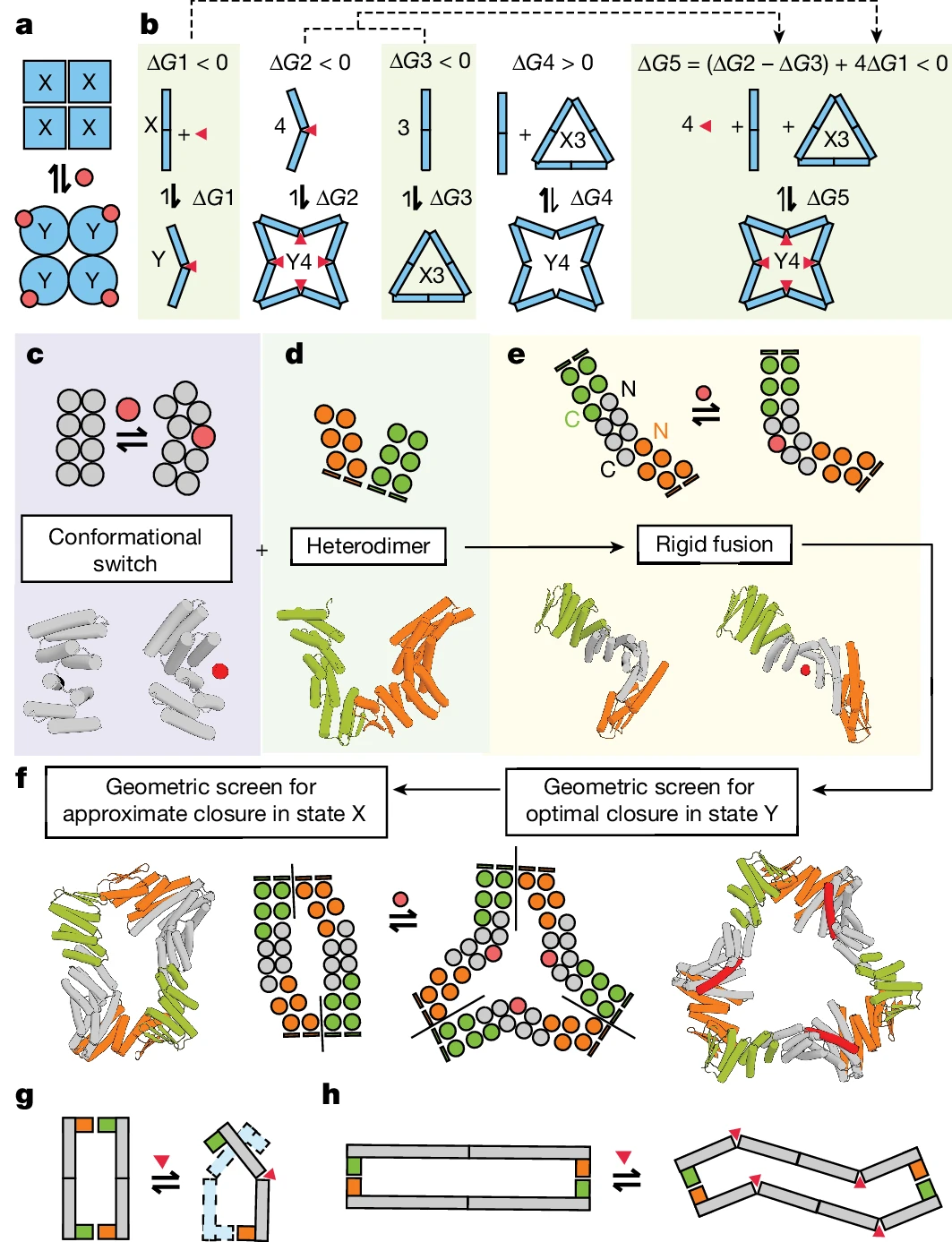 Illustration :
Illustration :
Les chercheurs démontrent l'application de RFdiffusion, ProteinMPNN et d'autres outils de conception pour créer une série d'assemblages de protéines dynamiques et conformationnellement commutables. En combinant des charnières à deux états et des modules d’interaction protéine-protéine personnalisés, les assemblages résultants sont très différents de tous ceux observés auparavant, élargissant ainsi les possibilités de la biologie synthétique.
Innovation clé :Une innovation clé de cette recherche est la conception d'assemblages de protéines. En plus de la polyvalence structurelle, l’équipe a également obtenu une liaison de haute affinité entre la nouvelle protéine et ses effecteurs, garantissant ainsi un contrôle allostérique programmé fiable.
Sélection des effecteurs :« Pour ce projet, nous avons utilisé des peptides spécifiques comme effecteurs, mais n'importe quel type de molécule peut produire une allostérie protéique dans de bonnes conditions », co-auteur Abbas Idris, étudiant diplômé à l'Université de Washington. ajouté.
Illustration : Conception d'un assemblage cyclique à commande allostérique. (Source : article) 
De plus, le nombre d'effecteurs liés à une protéine suit le modèle MWC : tous les sites de liaison sont remplis, voire aucun. En d’autres termes, la liaison des effecteurs homologues est hautement coopérative et l’assemblage résultant ne contient pas de mélange de protomères R et T.
Illustration : Une protéine bascule entre les états d’assemblage par conception. (Source : Nature)
Pour aller plus loin, les chercheurs ont conçu des protéines contenant des doubles charnières (deux charnières reliées par des boucles courtes) afin de créer une protéine qui ne change pas le nombre de protomères dans le Assemblage protéique. Protéine qui modifie sa structure 3D en réponse à la liaison effectrice. Effectivement, ces protéines ont fonctionné comme prévu, reproduisant le comportement dominant des protéines allostériques naturelles telles que l’hémoglobine. Enfin, les chercheurs ont également conçu des protomères qui s’assemblent ou se désassemblent lorsqu’ils sont liés à des molécules effectrices.
Les assemblages de protéines de novo spécifiques conçus dans l'étude comprennent un anneau formé par la dimérisation de deux monomères, qui, une fois assemblés, déclenche une production de lumière pour les applications de biodétection, et une structure en forme de cage, qui est contrôlée. pour libérer la charge utile pour la livraison de médicaments. Ces dynamiques protéiques ont été vérifiées expérimentalement in vitro par chromatographie d'exclusion stérique, spectrométrie de masse et microscopie électronique.
Pillai a souligné que les structures en anneaux présentent des propriétés précises supplémentaires telles que la coopérativité, un phénomène présenté par les systèmes naturels (par exemple les protéines sanguines, l'hémoglobine). Dans les systèmes synergiques, la liaison d’une molécule améliore la liaison d’autres molécules, créant des réactions marche-arrêt rapides qui sont essentielles à un contrôle précis, comme la capture de l’oxygène dans les poumons et sa libération dans les tissus.
"Historiquement, en laboratoire, nous avons fait beaucoup de travail pour contrôler l'affinité de lier une substance, comme par exemple la lier de plus en plus fort. Mais ce n'est pas le cas des systèmes biologiques. Le seul aspect pertinent », a déclaré Pillai. "Parfois, on veut pouvoir se lier sur une plage de concentrations très étroite."
Pour valider la conception, les chercheurs ont caractérisé plus de 20 assemblages de protéines par coloration négative et microscopie cryoélectronique. "Cela nous a permis de confirmer quelles conceptions se sont formées comme prévu et d'observer comment ces assemblages ont modifié leur structure lorsque des molécules effectrices ont été introduites", explique le Dr Andrew Borst, responsable du noyau de recherche en microscopie électronique de l'IPD.

L'étude a observé un couplage allostérique entre le site de liaison effecteur et l'interface d'assemblage, une distance supérieure à un nanomètre, ce qui représente une étendue énorme d'un point de vue atomique. Ce couplage étendu est essentiel pour créer des comportements protéiques complexes qui imitent et peut-être même dépassent la nature.
Applications potentielles
Les composants conçus comprennent des conteneurs à l'échelle nanométrique qui peuvent être ouverts et fermés à distance. De tels systèmes pourraient conduire à de nouveaux véhicules d’administration de médicaments dotés de mécanismes de contrôle avancés, notamment des dispositifs séquestrant les médicaments destructeurs de cellules jusqu’à ce qu’ils rencontrent des tumeurs.
Cette recherche ouvre la voie à la conception de fonctions contrôlées allostériquement au-delà de l'assemblage et du désassemblage des protéines, telles que la régulation de l'activité enzymatique pour réaliser des fonctions métaboliques et des nanomachines capables de convertir l'énergie en travail mécanique, similaires à celles des protéines actine et myosine responsable du mouvement cellulaire.
"La prochaine étape consiste à déterminer si nous pouvons former des interactions avec de petites molécules et catalyser avec précision des réactions, ce qui constitue une frontière plus difficile pour l'ensemble du domaine", a déclaré Pillai.
À l’avenir, l’équipe de recherche cherche à évaluer la dynamique de ces protéines modifiées dans un contexte biologique plus large. Les travaux futurs comprennent l’installation de ces fonctionnalités techniques sur les surfaces cellulaires dans la culture tissulaire, fournissant ainsi des outils précieux pour le contrôle par rétroaction dans des thérapies telles que la thérapie cellulaire adoptive.
Contenu de référence :
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
 La différence entre indexof et include
La différence entre indexof et include
 Quelles sont les méthodes pour redémarrer les applications sous Android ?
Quelles sont les méthodes pour redémarrer les applications sous Android ?
 Comment supprimer des pages vierges dans Word
Comment supprimer des pages vierges dans Word
 Comment ouvrir le format TIF sous Windows
Comment ouvrir le format TIF sous Windows
 Solution au problème selon lequel les fichiers exe ne peuvent pas être ouverts dans le système win10
Solution au problème selon lequel les fichiers exe ne peuvent pas être ouverts dans le système win10
 Quel fichier est Windows.old ?
Quel fichier est Windows.old ?
 Comment ouvrir le fichier apk
Comment ouvrir le fichier apk
 Comment définir le vlanid
Comment définir le vlanid