

Les interactions médicamenteuses inattendues (DDI) sont un problème important dans la recherche sur les médicaments et leurs applications cliniques, car elles sont très susceptibles de provoquer de graves effets indésirables des médicaments. réaction ou sevrage médicamenteux.
Bien que de nombreux modèles d'apprentissage profond aient obtenu de bons résultats en matière de prédiction du DDI, l'interprétabilité du modèle pour révéler les causes sous-jacentes du DDI n'a pas été largement explorée.
Des chercheurs de l'Université de Fuzhou, du premier hôpital affilié à l'Université de médecine du Fujian et de Yuanxing Intelligent Medicine ont proposé MeTDDI - un cadre d'apprentissage en profondeur avec une auto-attention locale-globale et une attention conjointe pour l'apprentissage basé sur l'intrigue de prédiction DDI du sujet.
Concernant l'interprétabilité, les chercheurs ont mené une évaluation approfondie de 73 médicaments (13 786 DDI), et MeTDDI peut expliquer avec précision les mécanismes structurels de 5 602 DDI impliquant 58 médicaments. De plus, MeTDDI montre le potentiel d’expliquer les mécanismes complexes de DDI et de réduire le risque de DDI.
MeTDDI offre une nouvelle perspective pour explorer les mécanismes DDI, qui faciliteront la découverte de médicaments et la polypharmacie, offrant ainsi des traitements plus sûrs aux patients.
L'étude s'intitulait « Apprentissage de graphiques basés sur des motifs pour la prédiction des interactions médicamenteuses via l'auto-attention locale-globale » et a été publiée dans « Nature Machine Intelligence » le 27 août 2024.

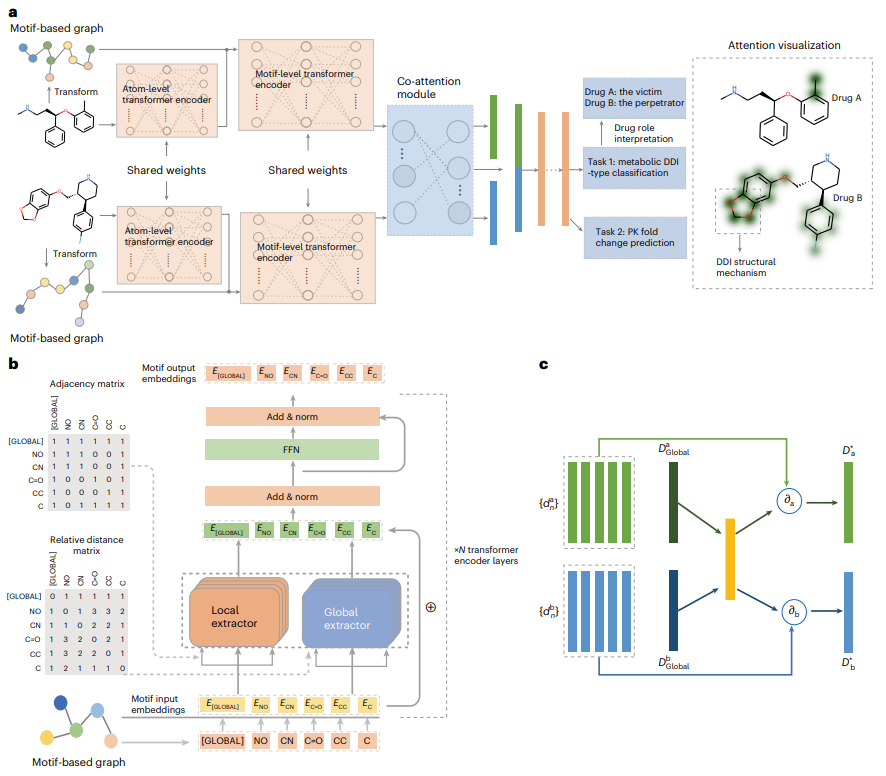
Bénéficiant de l'auto-attention locale-globale et des structures d'attention conjointe, MeTDDI peut apprendre efficacement les interactions de sous-structures intra- et intermoléculaires au sein/entre des graphiques basés sur des motifs, effectuant ainsi un raisonnement DDI.
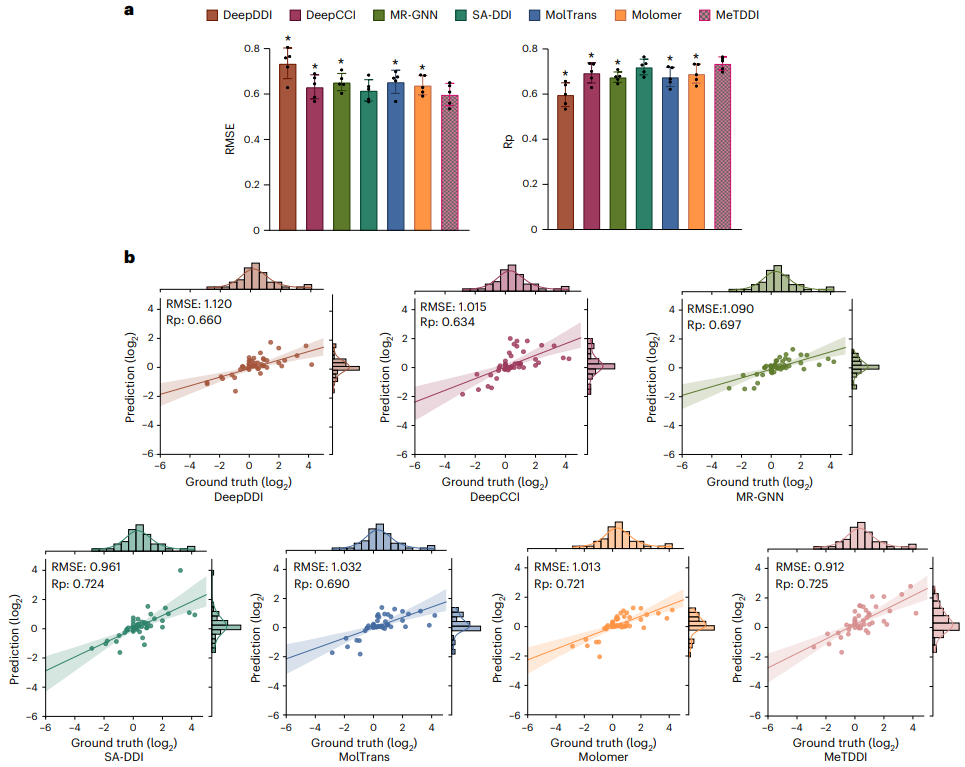
Les résultats de l'évaluation montrent qu'il atteint des performances compétitives par rapport aux références dans les tâches de classification et de régression. MeTDDI peut également identifier avec précision le rôle mécaniste d'un médicament (auteur ou victime) dans le DDI et quantifier l'impact de l'auteur sur la pharmacocinétique de la victime, ce qui est très bénéfique à la fois pour la recherche sur les médicaments et les applications cliniques.
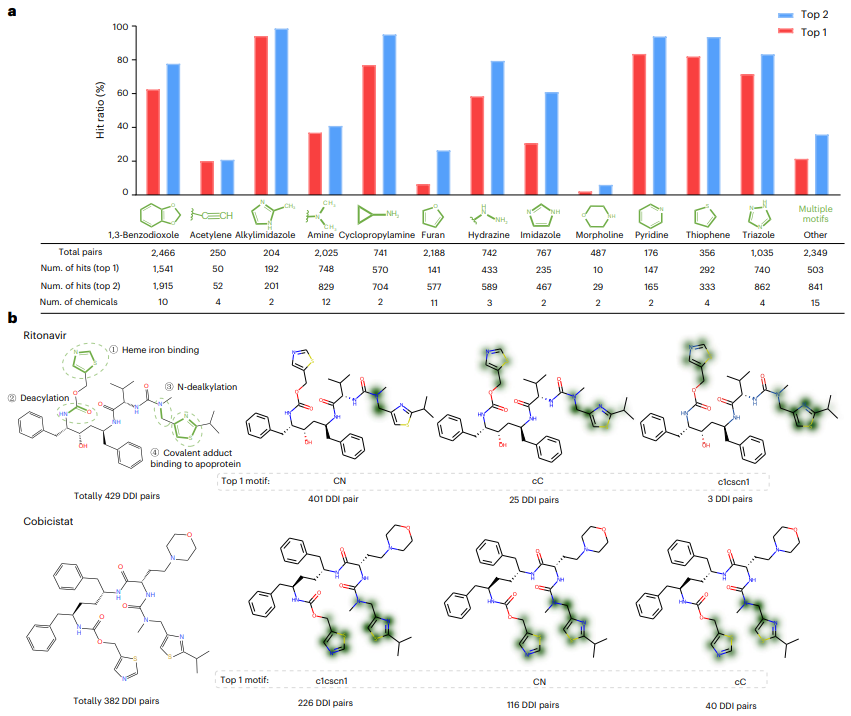
Concernant l'interprétabilité du modèle, MeTDDI démontre la capacité à identifier les sous-structures mécanistiques clés pertinentes pour le DDI.
Premièrement, les sous-structures clés visualisées par MeTDDI correspondent à peu près à celles rapportées dans la littérature à partir de l'analyse de 73 composés représentatifs (avec 13 786 paires DDI).
Deuxièmement, les chercheurs ont évalué l'interprétabilité du modèle MeTDDI et de deux modèles de pointe, à savoir CIGIN et CGIB. Les résultats montrent que MeTDDI présente également d’excellentes performances en termes d’interprétabilité du modèle.
De plus, MeTDDI peut mettre en évidence les sites métaboliques de produits chimiques associés à l'inhibition enzymatique.
Avantages du MeTDDI
Les méthodes traditionnelles n'expliquent le mécanisme du DDI qu'en testant l'inhibition de l'enzyme métabolique de l'agresseur in vitro, sans prendre pleinement en compte la victime. Ceci est problématique car la puissance de l'inhibition enzymatique par l'agresseur peut varier en fonction de l'identité chimique de la victime.
La victime peut modifier le modèle de liaison ou d'interaction des agresseurs avec les enzymes métaboliques (en particulier le CYP), ce qui entraîne divers mécanismes d'inhibition enzymatique. Cela peut expliquer pourquoi certains produits chimiques, tels que l'éthinylestradiol et le gestodène, qui sont de puissants inhibiteurs des enzymes métaboliques lorsqu'ils sont utilisés seuls in vitro, sont moins efficaces lorsqu'ils sont associés à leurs victimes. Cela peut expliquer pourquoi seules deux réactions ont été observées dans l'étude avec l'éthinylestradiol, qui serait un mécanisme d'inactivation du CYP3A4 in vitro.
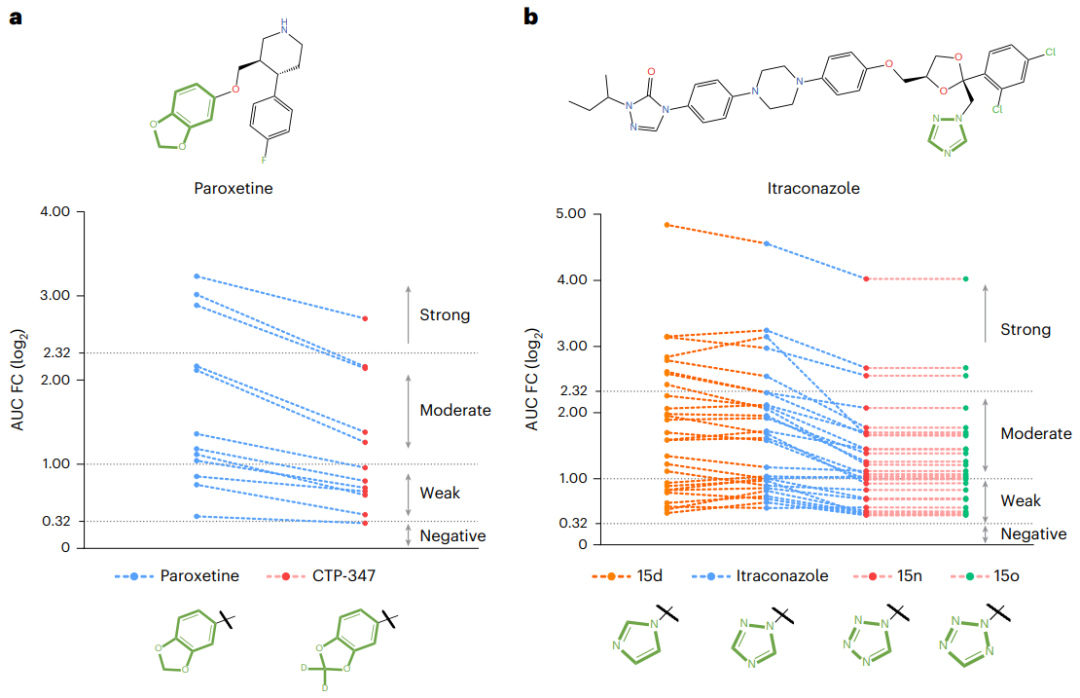
De plus, des études de cas sur la paroxétine et l'itraconazole montrent que le MeTDDI prédit correctement les changements de motifs dans les produits chimiques et correspond aux résultats des expériences biologiques, démontrant ainsi son potentiel pour aider les chercheurs à modifier la structure des médicaments pour atténuer les risques liés au MMDDI.
En résumé, MeTDDI améliore les capacités de prédiction du DDI et offre une nouvelle perspective pour comprendre et explorer les mécanismes du DDI, ce qui facilitera le développement de médicaments et la polypharmacie, offrant ainsi des traitements plus sûrs aux patients.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
 Comment flasher le téléphone Xiaomi
Comment flasher le téléphone Xiaomi
 Comment centrer un div en CSS
Comment centrer un div en CSS
 Comment ouvrir un fichier rar
Comment ouvrir un fichier rar
 Méthodes de lecture et d'écriture de fichiers Java DBF
Méthodes de lecture et d'écriture de fichiers Java DBF
 Comment résoudre le problème de l'absence du fichier msxml6.dll
Comment résoudre le problème de l'absence du fichier msxml6.dll
 Formules de permutation et de combinaison couramment utilisées
Formules de permutation et de combinaison couramment utilisées
 Numéro de téléphone mobile virtuel pour recevoir le code de vérification
Numéro de téléphone mobile virtuel pour recevoir le code de vérification
 album photo dynamique
album photo dynamique