
La segmentation d'image, l'une des procédures les plus élémentaires de la vision par ordinateur, permet à un système de décomposer et d'analyser diverses régions d'une image. Qu'il s'agisse de reconnaissance d'objets, d'imagerie médicale ou de conduite autonome, la segmentation est ce qui décompose les images en parties significatives.
Bien que les modèles d'apprentissage profond continuent d'être de plus en plus populaires dans cette tâche, les techniques traditionnelles de traitement d'images numériques restent puissantes et pratiques. Les approches examinées dans cet article incluent le seuillage, la détection des contours, la région et le clustering en mettant en œuvre un ensemble de données bien reconnu pour l'analyse des images cellulaires, l'ensemble de données d'images MIVIA HEp-2.
L'ensemble de données d'images MIVIA HEp-2 est un ensemble d'images des cellules utilisées pour analyser le profil des anticorps antinucléaires (ANA) à travers les cellules HEp-2. Il s’agit d’images 2D prises par microscopie à fluorescence. Cela le rend très approprié pour les tâches de segmentation, notamment celles liées à l'analyse d'images médicales, où la détection de régions cellulaires est la plus importante.
Passons maintenant aux techniques de segmentation utilisées pour traiter ces images, en comparant leurs performances en fonction des scores F1.
Le seuil est le processus par lequel les images en niveaux de gris sont converties en images binaires en fonction de l'intensité des pixels. Dans l’ensemble de données MIVIA HEp-2, ce processus est utile pour l’extraction de cellules en arrière-plan. C'est simple et efficace à un niveau relativement élevé, surtout avec la méthode d'Otsu, car elle calcule automatiquement le seuil optimal.
LaMéthode d'Otsu est une méthode de seuillage automatique, dans laquelle elle essaie de trouver la meilleure valeur de seuil pour produire la variance intra-classe minimale, séparant ainsi les deux classes : le premier plan (cellules) et l'arrière-plan. La méthode examine l'histogramme de l'image et calcule le seuil parfait, où la somme des variances d'intensité des pixels dans chaque classe est minimisée.
# Thresholding Segmentation
def thresholding(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Otsu's thresholding
_, thresh = cv.threshold(gray, 0, 255, cv.THRESH_BINARY + cv.THRESH_OTSU)
return thresh
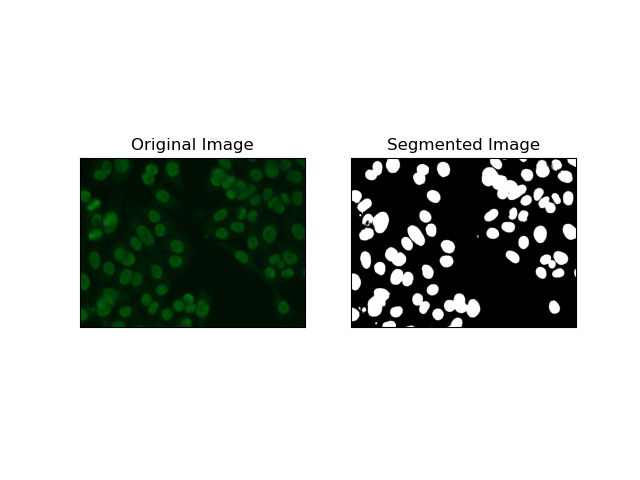
La détection des contours concerne l'identification des limites d'objets ou de régions, telles que les bords des cellules dans l'ensemble de données MIVIA HEp-2. Parmi les nombreuses méthodes disponibles pour détecter les changements brusques d'intensité, le Canny Edge Detector est la méthode la meilleure et donc la plus appropriée à utiliser pour détecter les limites cellulaires.
Canny Edge Detector est un algorithme à plusieurs étapes qui peut détecter les bords en détectant des zones de forts gradients d'intensité. Le processus comprend le lissage avec un filtre gaussien, le calcul des gradients d'intensité, l'application d'une suppression non maximale pour éliminer les réponses parasites et une opération finale de double seuillage pour la conservation des seuls bords saillants.
# Edge Detection Segmentation
def edge_detection(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Gaussian blur
gray = cv.GaussianBlur(gray, (3, 3), 0)
# Calculate lower and upper thresholds for Canny edge detection
sigma = 0.33
v = np.median(gray)
lower = int(max(0, (1.0 - sigma) * v))
upper = int(min(255, (1.0 + sigma) * v))
# Apply Canny edge detection
edges = cv.Canny(gray, lower, upper)
# Dilate the edges to fill gaps
kernel = np.ones((5, 5), np.uint8)
dilated_edges = cv.dilate(edges, kernel, iterations=2)
# Clean the edges using morphological opening
cleaned_edges = cv.morphologyEx(dilated_edges, cv.MORPH_OPEN, kernel, iterations=1)
# Find connected components and filter out small components
num_labels, labels, stats, _ = cv.connectedComponentsWithStats(
cleaned_edges, connectivity=8
)
min_size = 500
filtered_mask = np.zeros_like(cleaned_edges)
for i in range(1, num_labels):
if stats[i, cv.CC_STAT_AREA] >= min_size:
filtered_mask[labels == i] = 255
# Find contours of the filtered mask
contours, _ = cv.findContours(
filtered_mask, cv.RETR_EXTERNAL, cv.CHAIN_APPROX_SIMPLE
)
# Create a filled mask using the contours
filled_mask = np.zeros_like(gray)
cv.drawContours(filled_mask, contours, -1, (255), thickness=cv.FILLED)
# Perform morphological closing to fill holes
final_filled_image = cv.morphologyEx(
filled_mask, cv.MORPH_CLOSE, kernel, iterations=2
)
# Dilate the final filled image to smooth the edges
final_filled_image = cv.dilate(final_filled_image, kernel, iterations=1)
return final_filled_image
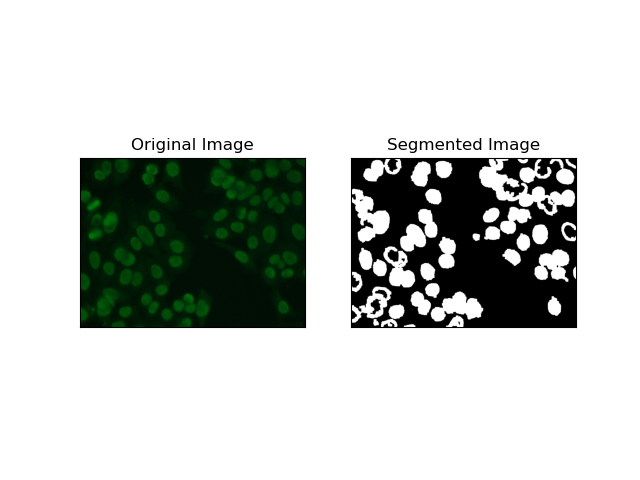
La segmentation basée sur les régions regroupe les pixels similaires en régions, en fonction de certains critères tels que l'intensité ou la couleur. La technique de Segmentation des bassins versants peut être utilisée pour aider à segmenter les images de cellules HEp-2 afin de pouvoir détecter les régions qui représentent les cellules ; il considère les intensités de pixels comme une surface topographique et décrit les régions distinctives.
LaLa segmentation des bassins versants traite les intensités des pixels comme une surface topographique. L'algorithme identifie des « bassins » dans lesquels il identifie des minima locaux puis inonde progressivement ces bassins pour agrandir des régions distinctes. Cette technique est très utile lorsqu'on veut séparer des objets en contact, comme dans le cas de cellules au sein d'images microscopiques, mais elle peut être sensible au bruit. Le processus peut être guidé par des marqueurs et la sur-segmentation peut souvent être réduite.
# Region-Based Segmentation
def region_based(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Otsu's thresholding
_, thresh = cv.threshold(gray, 0, 255, cv.THRESH_BINARY_INV + cv.THRESH_OTSU)
# Apply morphological opening to remove noise
kernel = np.ones((3, 3), np.uint8)
opening = cv.morphologyEx(thresh, cv.MORPH_OPEN, kernel, iterations=2)
# Dilate the opening to get the background
sure_bg = cv.dilate(opening, kernel, iterations=3)
# Calculate the distance transform
dist_transform = cv.distanceTransform(opening, cv.DIST_L2, 5)
# Threshold the distance transform to get the foreground
_, sure_fg = cv.threshold(dist_transform, 0.2 * dist_transform.max(), 255, 0)
sure_fg = np.uint8(sure_fg)
# Find the unknown region
unknown = cv.subtract(sure_bg, sure_fg)
# Label the markers for watershed algorithm
_, markers = cv.connectedComponents(sure_fg)
markers = markers + 1
markers[unknown == 255] = 0
# Apply watershed algorithm
markers = cv.watershed(img, markers)
# Create a mask for the segmented region
mask = np.zeros_like(gray, dtype=np.uint8)
mask[markers == 1] = 255
return mask
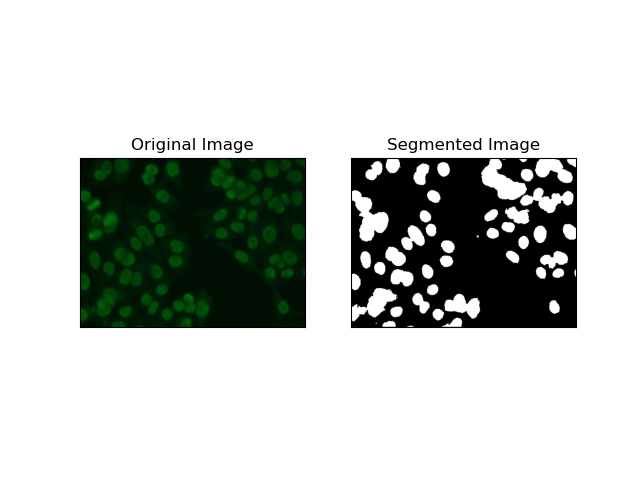
Les techniques de clustering telles que K-Means ont tendance à regrouper les pixels en groupes similaires, ce qui fonctionne bien lorsque l'on souhaite segmenter des cellules dans des environnements multicolores ou complexes, comme le montrent les images de cellules HEp-2. Fondamentalement, cela pourrait représenter différentes classes, comme une région cellulaire par rapport à un arrière-plan.
K-means is an unsupervised learning algorithm for clustering images based on the pixel similarity of color or intensity. The algorithm randomly selects K centroids, assigns each pixel to the nearest centroid, and updates the centroid iteratively until it converges. It is particularly effective in segmenting an image that has multiple regions of interest that are very different from one another.
# Clustering Segmentation
def clustering(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Reshape the image
Z = gray.reshape((-1, 3))
Z = np.float32(Z)
# Define the criteria for k-means clustering
criteria = (cv.TERM_CRITERIA_EPS + cv.TERM_CRITERIA_MAX_ITER, 10, 1.0)
# Set the number of clusters
K = 2
# Perform k-means clustering
_, label, center = cv.kmeans(Z, K, None, criteria, 10, cv.KMEANS_RANDOM_CENTERS)
# Convert the center values to uint8
center = np.uint8(center)
# Reshape the result
res = center[label.flatten()]
res = res.reshape((gray.shape))
# Apply thresholding to the result
_, res = cv.threshold(res, 0, 255, cv.THRESH_BINARY + cv.THRESH_OTSU)
return res

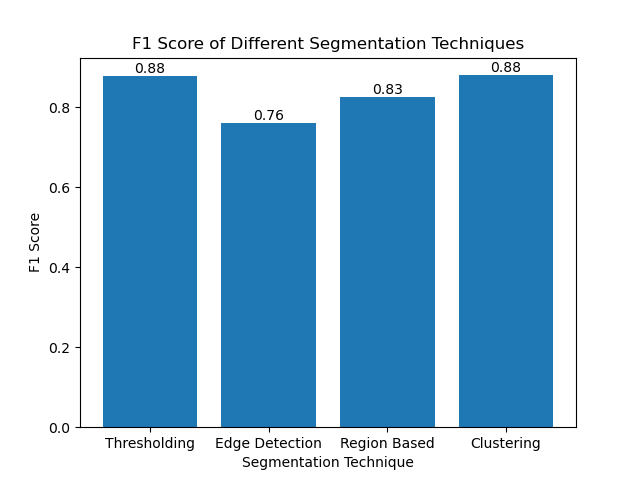
The F1 score is a measure that combines precision and recall together to compare the predicted segmentation image with the ground truth image. It is the harmonic mean of precision and recall, which is useful in cases of high data imbalance, such as in medical imaging datasets.
We calculated the F1 score for each segmentation method by flattening both the ground truth and the segmented image and calculating the weighted F1 score.
def calculate_f1_score(ground_image, segmented_image):
ground_image = ground_image.flatten()
segmented_image = segmented_image.flatten()
return f1_score(ground_image, segmented_image, average="weighted")
We then visualized the F1 scores of different methods using a simple bar chart:
Although many recent approaches for image segmentation are emerging, traditional segmentation techniques such as thresholding, edge detection, region-based methods, and clustering can be very useful when applied to datasets such as the MIVIA HEp-2 image dataset.
Each method has its strength:
By evaluating these methods using F1 scores, we understand the trade-offs each of these models has. These methods may not be as sophisticated as what is developed in the newest models of deep learning, but they are still fast, interpretable, and serviceable in a broad range of applications.
Thanks for reading! I hope this exploration of traditional image segmentation techniques inspires your next project. Feel free to share your thoughts and experiences in the comments below!
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
 La différence entre l'hibernation et le sommeil de Windows
La différence entre l'hibernation et le sommeil de Windows
 Comment configurer Douyin pour empêcher tout le monde de voir l'œuvre
Comment configurer Douyin pour empêcher tout le monde de voir l'œuvre
 Qu'est-ce que Bitcoin ? Est-ce légal ?
Qu'est-ce que Bitcoin ? Est-ce légal ?
 Le rôle des serveurs de noms de domaine
Le rôle des serveurs de noms de domaine
 Résumé des connaissances de base de Java
Résumé des connaissances de base de Java
 Utilisation des tâches C#
Utilisation des tâches C#
 Quelles versions du système Linux existe-t-il ?
Quelles versions du système Linux existe-t-il ?
 Utilisation des fonctions aléatoires du langage C
Utilisation des fonctions aléatoires du langage C