 Périphériques technologiques
Périphériques technologiques
 IA
IA
 Du travail manuel à la révolution industrielle ! Article Nature : Cinq domaines de l'analyse d'images biologiques révolutionnés par l'apprentissage profond
Du travail manuel à la révolution industrielle ! Article Nature : Cinq domaines de l'analyse d'images biologiques révolutionnés par l'apprentissage profond
Du travail manuel à la révolution industrielle ! Article Nature : Cinq domaines de l'analyse d'images biologiques révolutionnés par l'apprentissage profond
Un millimètre cube ne paraît pas gros, c'est la taille d'une graine de sésame, mais dans le cerveau humain, ce petit espace peut accueillir environ 50 000 lignes nerveuses reliées par 134 millions de synapses (fils neuronaux).
Pour générer les données brutes, les bioscientifiques devaient utiliser la microscopie électronique à coupe ultrafine en série pour imager des milliers de fragments de tissus en 11 mois.
Et la quantité de données finalement obtenues a atteint un étonnant 1,4 PetaBytes (soit 1400 To, équivalent à la capacité d'environ 2 millions de CD-ROM), ce qui est tout simplement un chiffre astronomique pour les chercheurs.
Jeff Lichtman, biologiste moléculaire et cellulaire à l'Université Harvard, a déclaré que si cela est fait uniquement à la main, il est tout simplement impossible pour les humains de tracer manuellement toutes les lignes nerveuses. Il n'y a même pas assez de personnes sur terre. qui peut vraiment faire le travail efficacement.
Les progrès de la technologie de la microscopie ont apporté une grande quantité de données d'imagerie, mais la quantité de données est trop importante et la main-d'œuvre est insuffisante. C'est également le cas pour la Connectomics (Connectomics, une discipline qui étudie la structure. et connexions fonctionnelles du cerveau), et d'autres Phénomènes courants biologiques chez les sujets de terrain.
Mais la mission de l'informatique est précisément de résoudre ce genre de problème de ressources humaines insuffisantes, en particulier l'algorithme d'apprentissage en profondeur optimisé, qui peut exploiter des modèles de données à partir d'ensembles de données à grande échelle.
Beth Cimini, biologiste computationnelle au Broad Institute du MIT et à l'Université Harvard à Cambridge, a déclaré qu'au cours des dernières années, l'apprentissage profond a joué un rôle énorme dans la promotion du domaine de la biologie et a développé de nombreux outils de recherche. .
Voici les cinq domaines de l'analyse d'images biologiques résumés par les éditeurs de Nature dans lesquels l'apprentissage profond a apporté des changements.
Connectomique à grande échelle
Le deep learning permet aux chercheurs de générer des connectomes de plus en plus complexes à partir de mouches des fruits, de souris et même d'humains.
Ces données peuvent aider les neuroscientifiques à comprendre comment fonctionne le cerveau et comment la structure du cerveau change au cours du développement et de la maladie, mais les connexions neuronales ne sont pas faciles à cartographier.
En 2018, Lichtman s'est associé à Viren Jain, responsable de la connectomique chez Google à Mountain View, en Californie, pour trouver des solutions pour les algorithmes d'intelligence artificielle dont l'équipe avait besoin.
La tâche d'analyse d'images en connectomique est en réalité très difficile, il faut être capable de tracer ces fines lignes, les axones et les dendrites des cellules, et sur de longues distances, Les méthodes traditionnelles de traitement d'images sont Il y aura beaucoup de des erreurs dans cette tâche et cela sera fondamentalement inutile pour cette tâche .
Ces brins nerveux peuvent être plus fins qu’un micron, s’étendant sur des centaines de microns ou même sur des tissus de la taille d’un millimètre.
Et l'algorithme d'apprentissage en profondeurpeut non seulement analyser automatiquement les données connectomiques, mais également maintenir une haute précision.
Les chercheurs peuvent utiliser des ensembles de données annotés contenant des caractéristiques d'intérêt pour former des modèles informatiques complexes capables d'identifier rapidement les mêmes caractéristiques dans d'autres données.
Anna Kreshuk, informaticienne au Laboratoire européen de biologie moléculaire, estime que le processus d'utilisation d'algorithmes d'apprentissage profond est similaire à "donner un exemple". Tant qu'il y a suffisamment d'exemples, vous pouvez résoudre tous les problèmes.
Mais même en utilisant l’apprentissage profond, l’équipe de Lichtman et Jain avait une tâche difficile : cartographier des segments du cortex cérébral humain.
Dans la phase de collecte de données, il a fallu 326 jours rien que pour prélever plus de 5 000 coupes de tissus ultra-minces.
Deux chercheurs ont passé environ 100 heures à annoter manuellement des images et à suivre les neurones pour créer un ensemble de données de vérité terrain pour entraîner l'algorithme.
L'algorithme formé à l'aide de données standard peut automatiquement assembler les images, identifier les neurones et les synapses et générer le connectome final.
L'équipe de Jain a également investi beaucoup de ressources informatiques pour résoudre ce problème, notamment des milliers d'unités de traitement tenseur (TPU) , et a passé mois à prétraiter 1 million d'heures TPU requises.
Bien que les chercheurs aient obtenu le plus grand ensemble de données actuellement collectées et puissent le reconstruire à un niveau très fin, cette quantité de données ne représente qu'environ 0,0001% du cerveau humain
Grâce aux améliorations des algorithmes et du matériel. , les chercheurs devraient être capables de cartographier des zones plus vastes du cerveau et de résoudre davantage de caractéristiques cellulaires, telles que les organites et même les protéines.
Au moins, l'apprentissage en profondeur offre une possibilité.
Histologie virtuelleL'histologie est un outil important en médecine pour diagnostiquer des maladies sur la base d'une coloration chimique ou moléculaire.
Mais l'ensemble du processus prend du temps et est laborieux, et prend généralement des jours, voire des semaines.
La biopsie est d'abord découpée en coupes minces, colorées pour montrer les caractéristiques cellulaires et subcellulaires, puis le pathologiste lit et interprète les résultats.
Aydogan Ozcan, ingénieur informatique à l'Université de Californie à Los Angeles, estime que l'ensemble du processus peut être accéléré grâce à l'apprentissage en profondeur.
Il a formé un modèle d'apprentissage profond personnalisé, simulé la coloration d'une coupe de tissu via un ordinateur, a nourri le modèle avec des dizaines de milliers d'échantillons non colorés et colorés sur la même section et a laissé le modèle calculer la différence entre eux .
En plus de l'avantage de temps de la coloration virtuelle (elle peut être réalisée en un instant), les pathologistes ont découvert par observation qu'il n'y a presque aucune différence entre la coloration virtuelle et la coloration traditionnelle, et les professionnels ne peuvent pas faire la différence. Les résultats expérimentaux montrent que l'algorithme peut reproduire la coloration moléculaire du biomarqueur du cancer du sein HER2 en
secondes, un processus qui prend généralement au moins 24 heures dans un laboratoire d'histologie. Un groupe d'experts composé de trois pathologistes du sein a évalué ces images et les a jugées d'une qualité et d'une précision comparables à la coloration immunohistochimique traditionnelle.
Ozcan voit le potentiel de commercialiser la coloration virtuelle dans le développement de médicaments, mais il espère éliminer le besoin de colorants toxiques et d'équipements de coloration coûteux en histologie.
Recherche de cellules
Si vous souhaitez extraire des données à partir d'images cellulaires, vous devez connaître l'emplacement réel des cellules dans l'image. Ce processus est également appelé segmentation cellulaire.
Les chercheurs doivent observer
les cellules au microscope ou décrire les cellules une par unedans le logiciel. Morgan Schwartz, biologiste informatique au California Institute of Technology, cherche des moyens d'automatiser le traitement. À mesure que les ensembles de données d'imagerie deviennent de plus en plus volumineux, les méthodes manuelles traditionnelles se heurtent également à des goulots d'étranglement et certaines expériences
sont impossibles sans automatisation. .Analysez. Le conseiller diplômé de Schwartz, le bio-ingénieur David Van Valen, a créé un ensemble de modèles d'intelligence artificielle et les a publiés sur le site Web deepcell.org, qui peuvent être utilisés pour calculer et analyser les cellules et d'autres caractéristiques des cellules vivantes et des images de tissus enregistrées. .
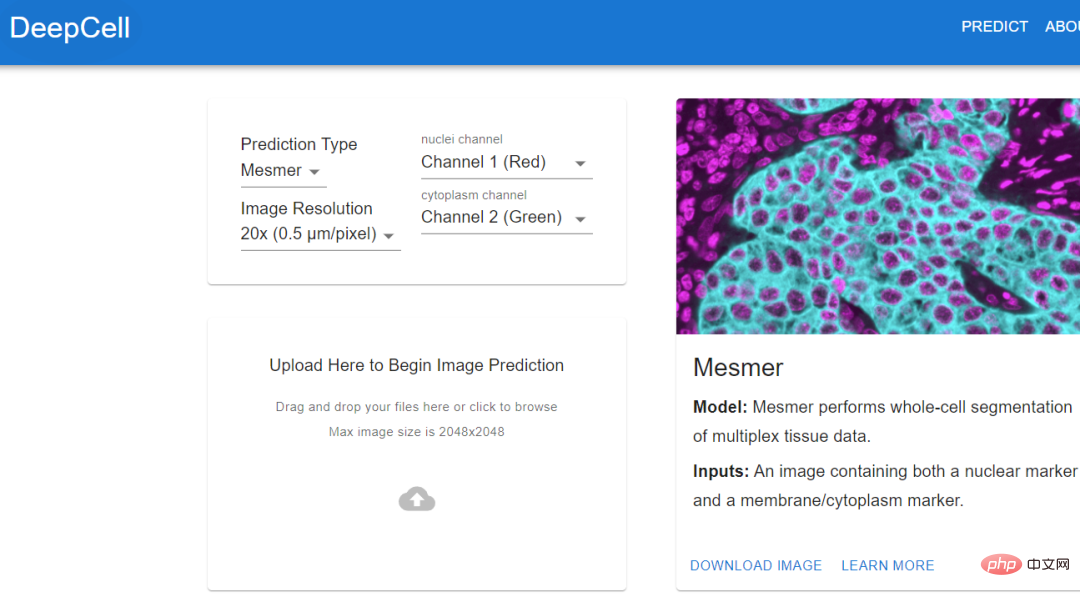 Van Valen, en collaboration avec des collaborateurs tels que Noah Greenwald, biologiste du cancer à l'Université de Stanford, a également développé Mesmer, un modèle d'apprentissage profond qui peut
Van Valen, en collaboration avec des collaborateurs tels que Noah Greenwald, biologiste du cancer à l'Université de Stanford, a également développé Mesmer, un modèle d'apprentissage profond qui peut
. Les chercheurs pourraient utiliser ces informations pour distinguer les tissus cancéreux des tissus non cancéreux et rechercher des différences avant et après le traitement, ou pour mieux comprendre pourquoi certains patients répondent ou ne répondent pas en fonction des changements d'imagerie, selon Greenwald. et déterminer les sous-types de tumeurs. Le projet Human Protein Atlas tire parti d'une autre application du deep learning : la localisation intracellulaire. Emma Lundberg, bio-ingénieure à l'Université de Stanford, a déclaré qu'au cours des dernières décennies, le projet a généré des millions d'images illustrant l'expression de protéines dans les cellules et les tissus humains. Au début, les participants au projet devaient annoter manuellement ces images, mais cette méthode n'était pas viable et Lundberg a commencé à demander l'aide d'algorithmes d'intelligence artificielle. Au cours des dernières années, elle a commencé à lancer des solutions de crowdsourcing dans le cadre du Kaggle Challenge. Des scientifiques et des passionnés d'intelligence artificielle réaliseront diverses tâches informatiques pour les prix. Les prix pour les deux projets s'élèvent respectivement à 37 000 dollars américains. et 25 000$. Les participants concevront des modèles d'apprentissage automatique supervisé et annoteront des images de cartes protéiques. Les résultats du défi Kaggle ont également surpris les membres du projet. Les performances du modèle gagnant sont environ 20 % supérieures à celles de la précédente classification multi-étiquettes des modèles de localisation des protéines de Lundberg, et peuvent être généralisées aux lignées cellulaires (cellules). line), a également réalisé de nouvelles percées industrielles dans la classification précise des protéines présentes dans plusieurs emplacements cellulaires. Avec le modèle, les expériences biologiques peuvent continuer à progresser. La localisation des protéines humaines est importante car la même protéine se comporte différemment à différents endroits. Savoir si une protéine se trouve dans le noyau ou dans les mitochondries, cela aide. pour comprendre sa fonctionnalité. Mackenzie Mathis, neuroscientifique au Centre de biotechnologie du campus de l'École Polytechnique Fédérale de Lausanne en Suisse, s'intéresse depuis longtemps à la manière dont le cerveau détermine le comportement. À cette fin, elle a développé un programme appelé DeepLabCut qui permet aux neuroscientifiques de suivre les postures et les mouvements fins des animaux à partir de vidéos et de convertir les « vidéos de chats » et autres enregistrements d'animaux en données. DeepLabcut fournit une interface utilisateur graphique qui permet aux chercheurs de télécharger et d'annoter des vidéos et de former des modèles d'apprentissage en profondeur en un seul clic. En avril de cette année, l'équipe de Mathis a étendu le logiciel pour estimer les poses de plusieurs animaux simultanément, ce qui constitue un nouveau défi pour les humains et l'intelligence artificielle. En appliquant le modèle formé par DeepLabCut aux ouistitis, les chercheurs ont découvert que lorsque les animaux sont rapprochés, leurs corps s'alignent et regardent dans des directions similaires, et lorsqu'ils se séparent, ils ont tendance à se faire face. Les biologistes identifient les postures des animaux pour comprendre comment deux animaux interagissent, regardent ou observent le monde. Localisation des protéines
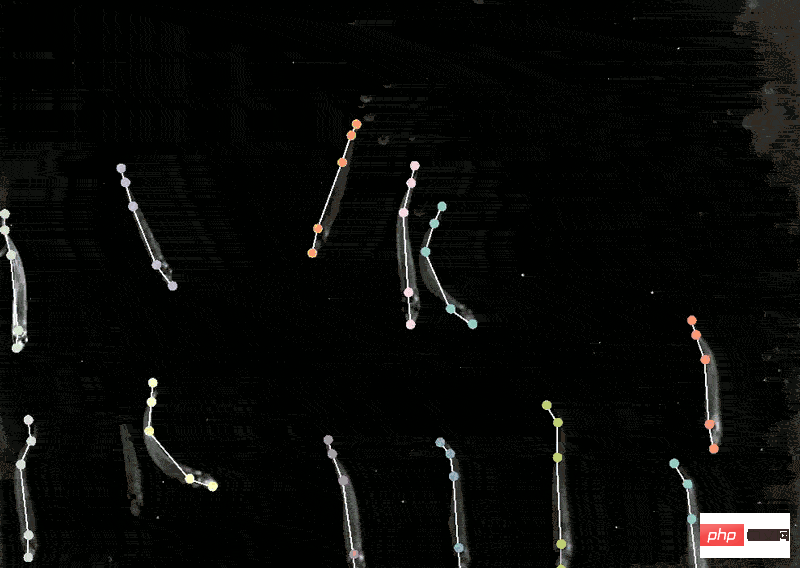
Suivi du comportement animal
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!

Outils d'IA chauds

Undresser.AI Undress
Application basée sur l'IA pour créer des photos de nu réalistes

AI Clothes Remover
Outil d'IA en ligne pour supprimer les vêtements des photos.

Undress AI Tool
Images de déshabillage gratuites

Clothoff.io
Dissolvant de vêtements AI

AI Hentai Generator
Générez AI Hentai gratuitement.

Article chaud

Outils chauds

Bloc-notes++7.3.1
Éditeur de code facile à utiliser et gratuit

SublimeText3 version chinoise
Version chinoise, très simple à utiliser

Envoyer Studio 13.0.1
Puissant environnement de développement intégré PHP

Dreamweaver CS6
Outils de développement Web visuel

SublimeText3 version Mac
Logiciel d'édition de code au niveau de Dieu (SublimeText3)
 Méthodes et étapes d'utilisation de BERT pour l'analyse des sentiments en Python
Jan 22, 2024 pm 04:24 PM
Méthodes et étapes d'utilisation de BERT pour l'analyse des sentiments en Python
Jan 22, 2024 pm 04:24 PM
BERT est un modèle de langage d'apprentissage profond pré-entraîné proposé par Google en 2018. Le nom complet est BidirectionnelEncoderRepresentationsfromTransformers, qui est basé sur l'architecture Transformer et présente les caractéristiques d'un codage bidirectionnel. Par rapport aux modèles de codage unidirectionnels traditionnels, BERT peut prendre en compte les informations contextuelles en même temps lors du traitement du texte, de sorte qu'il fonctionne bien dans les tâches de traitement du langage naturel. Sa bidirectionnalité permet à BERT de mieux comprendre les relations sémantiques dans les phrases, améliorant ainsi la capacité expressive du modèle. Grâce à des méthodes de pré-formation et de réglage fin, BERT peut être utilisé pour diverses tâches de traitement du langage naturel, telles que l'analyse des sentiments, la dénomination
 Intégration d'espace latent : explication et démonstration
Jan 22, 2024 pm 05:30 PM
Intégration d'espace latent : explication et démonstration
Jan 22, 2024 pm 05:30 PM
L'intégration d'espace latent (LatentSpaceEmbedding) est le processus de mappage de données de grande dimension vers un espace de faible dimension. Dans le domaine de l'apprentissage automatique et de l'apprentissage profond, l'intégration d'espace latent est généralement un modèle de réseau neuronal qui mappe les données d'entrée de grande dimension dans un ensemble de représentations vectorielles de basse dimension. Cet ensemble de vecteurs est souvent appelé « vecteurs latents » ou « latents ». encodages". Le but de l’intégration de l’espace latent est de capturer les caractéristiques importantes des données et de les représenter sous une forme plus concise et compréhensible. Grâce à l'intégration de l'espace latent, nous pouvons effectuer des opérations telles que la visualisation, la classification et le regroupement de données dans un espace de faible dimension pour mieux comprendre et utiliser les données. L'intégration d'espace latent a de nombreuses applications dans de nombreux domaines, tels que la génération d'images, l'extraction de caractéristiques, la réduction de dimensionnalité, etc. L'intégration de l'espace latent est le principal
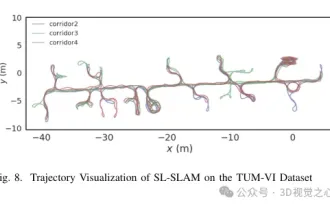 Au-delà d'ORB-SLAM3 ! SL-SLAM : les scènes de faible luminosité, de gigue importante et de texture faible sont toutes gérées
May 30, 2024 am 09:35 AM
Au-delà d'ORB-SLAM3 ! SL-SLAM : les scènes de faible luminosité, de gigue importante et de texture faible sont toutes gérées
May 30, 2024 am 09:35 AM
Écrit précédemment, nous discutons aujourd'hui de la manière dont la technologie d'apprentissage profond peut améliorer les performances du SLAM (localisation et cartographie simultanées) basé sur la vision dans des environnements complexes. En combinant des méthodes d'extraction de caractéristiques approfondies et de correspondance de profondeur, nous introduisons ici un système SLAM visuel hybride polyvalent conçu pour améliorer l'adaptation dans des scénarios difficiles tels que des conditions de faible luminosité, un éclairage dynamique, des zones faiblement texturées et une gigue importante. Notre système prend en charge plusieurs modes, notamment les configurations étendues monoculaire, stéréo, monoculaire-inertielle et stéréo-inertielle. En outre, il analyse également comment combiner le SLAM visuel avec des méthodes d’apprentissage profond pour inspirer d’autres recherches. Grâce à des expériences approfondies sur des ensembles de données publiques et des données auto-échantillonnées, nous démontrons la supériorité du SL-SLAM en termes de précision de positionnement et de robustesse du suivi.
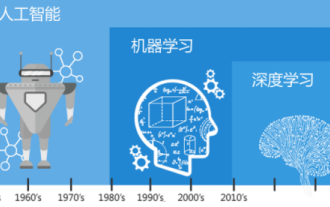 Comprendre en un seul article : les liens et les différences entre l'IA, le machine learning et le deep learning
Mar 02, 2024 am 11:19 AM
Comprendre en un seul article : les liens et les différences entre l'IA, le machine learning et le deep learning
Mar 02, 2024 am 11:19 AM
Dans la vague actuelle de changements technologiques rapides, l'intelligence artificielle (IA), l'apprentissage automatique (ML) et l'apprentissage profond (DL) sont comme des étoiles brillantes, à la tête de la nouvelle vague des technologies de l'information. Ces trois mots apparaissent fréquemment dans diverses discussions de pointe et applications pratiques, mais pour de nombreux explorateurs novices dans ce domaine, leurs significations spécifiques et leurs connexions internes peuvent encore être entourées de mystère. Alors regardons d'abord cette photo. On constate qu’il existe une corrélation étroite et une relation progressive entre l’apprentissage profond, l’apprentissage automatique et l’intelligence artificielle. Le deep learning est un domaine spécifique du machine learning, et le machine learning
 Super fort! Top 10 des algorithmes de deep learning !
Mar 15, 2024 pm 03:46 PM
Super fort! Top 10 des algorithmes de deep learning !
Mar 15, 2024 pm 03:46 PM
Près de 20 ans se sont écoulés depuis que le concept d'apprentissage profond a été proposé en 2006. L'apprentissage profond, en tant que révolution dans le domaine de l'intelligence artificielle, a donné naissance à de nombreux algorithmes influents. Alors, selon vous, quels sont les 10 meilleurs algorithmes pour l’apprentissage profond ? Voici les meilleurs algorithmes d’apprentissage profond, à mon avis. Ils occupent tous une position importante en termes d’innovation, de valeur d’application et d’influence. 1. Contexte du réseau neuronal profond (DNN) : Le réseau neuronal profond (DNN), également appelé perceptron multicouche, est l'algorithme d'apprentissage profond le plus courant lorsqu'il a été inventé pour la première fois, jusqu'à récemment en raison du goulot d'étranglement de la puissance de calcul. années, puissance de calcul, La percée est venue avec l'explosion des données. DNN est un modèle de réseau neuronal qui contient plusieurs couches cachées. Dans ce modèle, chaque couche transmet l'entrée à la couche suivante et
 AlphaFold 3 est lancé, prédisant de manière exhaustive les interactions et les structures des protéines et de toutes les molécules de la vie, avec une précision bien plus grande que jamais
Jul 16, 2024 am 12:08 AM
AlphaFold 3 est lancé, prédisant de manière exhaustive les interactions et les structures des protéines et de toutes les molécules de la vie, avec une précision bien plus grande que jamais
Jul 16, 2024 am 12:08 AM
Editeur | Radis Skin Depuis la sortie du puissant AlphaFold2 en 2021, les scientifiques utilisent des modèles de prédiction de la structure des protéines pour cartographier diverses structures protéiques dans les cellules, découvrir des médicaments et dresser une « carte cosmique » de chaque interaction protéique connue. Tout à l'heure, Google DeepMind a publié le modèle AlphaFold3, capable d'effectuer des prédictions de structure conjointe pour des complexes comprenant des protéines, des acides nucléiques, de petites molécules, des ions et des résidus modifiés. La précision d’AlphaFold3 a été considérablement améliorée par rapport à de nombreux outils dédiés dans le passé (interaction protéine-ligand, interaction protéine-acide nucléique, prédiction anticorps-antigène). Cela montre qu’au sein d’un cadre unique et unifié d’apprentissage profond, il est possible de réaliser
 Comment utiliser les modèles hybrides CNN et Transformer pour améliorer les performances
Jan 24, 2024 am 10:33 AM
Comment utiliser les modèles hybrides CNN et Transformer pour améliorer les performances
Jan 24, 2024 am 10:33 AM
Convolutional Neural Network (CNN) et Transformer sont deux modèles d'apprentissage en profondeur différents qui ont montré d'excellentes performances sur différentes tâches. CNN est principalement utilisé pour les tâches de vision par ordinateur telles que la classification d'images, la détection de cibles et la segmentation d'images. Il extrait les caractéristiques locales de l'image via des opérations de convolution et effectue une réduction de dimensionnalité des caractéristiques et une invariance spatiale via des opérations de pooling. En revanche, Transformer est principalement utilisé pour les tâches de traitement du langage naturel (NLP) telles que la traduction automatique, la classification de texte et la reconnaissance vocale. Il utilise un mécanisme d'auto-attention pour modéliser les dépendances dans des séquences, évitant ainsi le calcul séquentiel dans les réseaux neuronaux récurrents traditionnels. Bien que ces deux modèles soient utilisés pour des tâches différentes, ils présentent des similitudes dans la modélisation des séquences.
 Algorithme RMSprop amélioré
Jan 22, 2024 pm 05:18 PM
Algorithme RMSprop amélioré
Jan 22, 2024 pm 05:18 PM
RMSprop est un optimiseur largement utilisé pour mettre à jour les poids des réseaux de neurones. Il a été proposé par Geoffrey Hinton et al. en 2012 et est le prédécesseur de l'optimiseur Adam. L'émergence de l'optimiseur RMSprop vise principalement à résoudre certains problèmes rencontrés dans l'algorithme de descente de gradient SGD, tels que la disparition de gradient et l'explosion de gradient. En utilisant l'optimiseur RMSprop, le taux d'apprentissage peut être ajusté efficacement et les pondérations mises à jour de manière adaptative, améliorant ainsi l'effet de formation du modèle d'apprentissage en profondeur. L'idée principale de l'optimiseur RMSprop est d'effectuer une moyenne pondérée des gradients afin que les gradients à différents pas de temps aient des effets différents sur les mises à jour de poids. Plus précisément, RMSprop calcule le carré de chaque paramètre





