 Périphériques technologiques
Périphériques technologiques
 IA
IA
 Des possibilités infinies Le modèle d'IA générative NVIDIA accélère la synthèse des protéines
Des possibilités infinies Le modèle d'IA générative NVIDIA accélère la synthèse des protéines
Des possibilités infinies Le modèle d'IA générative NVIDIA accélère la synthèse des protéines
Au cours des deux dernières années, l’apprentissage automatique a révolutionné la prédiction de la structure des protéines. Aujourd’hui, l’intelligence artificielle a déclenché une nouvelle révolution dans le domaine de la conception des protéines.
Depuis l’avènement de l’IA, de nombreux scientifiques se sont lancés dans la voie de son utilisation pour mener des recherches sur les protéines. Les biologistes ont découvert que grâce à l’apprentissage automatique, des molécules de protéines peuvent être créées en quelques secondes. Dans le passé, cette période pouvait durer plusieurs mois.
Récemment, la start-up Evozyne a utilisé des modèles d'IA pré-entraînés fournis par NVIDIA pour créer deux protéines au potentiel important dans les domaines médical et des énergies propres. L’une des protéines est utilisée pour traiter une maladie congénitale et une autre pour consommer du dioxyde de carbone afin de réduire le réchauffement climatique.
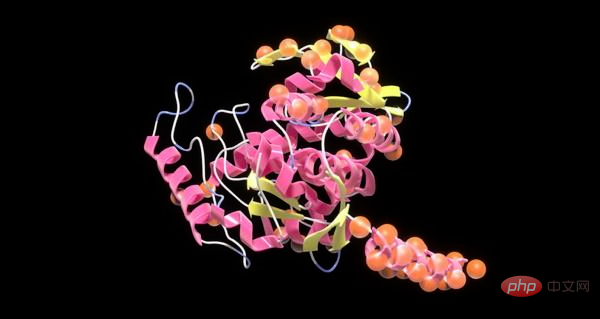
De cette manière, les scientifiques utilisent NVIDIA BioNeMo pour créer des modèles de langage à grande échelle capables de générer des protéines de haute qualité pour accélérer le développement de médicaments et contribuer à créer un environnement plus durable.
Une nouvelle façon d'accélérer la découverte de médicaments
Andrew Ferguson, co-fondateur d'Evozyne et co-auteur de l'article, a déclaré : "Ce qui est gratifiant, c'est que les protéines synthétiques produites par ce modèle d'IA au premier tour sont tout comme les protéines naturelles. Indique que le modèle a appris les règles de conception de la nature. »
Evozyne utilise le ProtT5 de NVIDIA. ProtT5 est un modèle Transformer qui fait partie de NVIDIA BioNeMo, un cadre logiciel et un service permettant de créer des modèles d'IA médicale.
L'ingénieur moléculaire Ferguson, dont les domaines de recherche couvrent les domaines de la chimie et de l'apprentissage automatique, a déclaré : "BioNeMo est très puissant et nous permet de former un modèle puis d'utiliser le modèle pour exécuter des tâches de travail à un coût très faible, en un rien de temps. de secondes. Générez des millions de séquences. Le modèle prédit comment assembler de nouvelles protéines qui répondent aux besoins d'Evozyne. "
Ce modèle est au cœur du pipeline Evovyne ProT-VAE. Le pipeline ProT-VAE d'Evozyne combine le puissant modèle Transformer de NVIDIA BioNeMo avec des auto-encodeurs variationnels (VAE).
Il a déclaré : "Il y a quelques années, personne n'avait remarqué que les protéines pouvaient être conçues à l'aide de grands modèles de langage combinés à des auto-encodeurs variationnels." acides aminés dans une protéine. Cela représente des centaines de mutations.
Joshua Moller, data scientist chez Evozyne, a déclaré : « Ils accélèrent la formation en étendant le travail à plusieurs GPU.
Cela réduit le temps de formation d'un grand modèle d'IA de plusieurs mois à une semaine. Nous pouvons ainsi former des modèles qui seraient autrement impossibles. à entraîner, comme certains avec des milliards de paramètres pouvant être entraînés. «
Modèle d'IA révolutionnaireLa méthode traditionnelle de conception d'ingénierie des protéines, l'évolution dirigée, utilise une approche lente et non planifiée qui ne modifie généralement que la séquence de quelques acides aminés à la fois. L'apprentissage automatique aide
BioNeMo est une IA. -Service cloud et cadre de développement de médicaments basés sur NVIDIA NeMo Megatron pour la formation et le déploiement d'applications à grande échelle à l'échelle du supercalcul. Le service Biomolecule Transformer AI comprend un LLM pré-entraîné, une prise en charge native des formats de fichiers courants pour les protéines, l'ADN, l'ARN. et chimie, et est disponible pour SMILES (pour les structures moléculaires) et FASTA (pour les séquences d'acides aminés et de nucléotides)) à l'aide du chargeur de données
Avec BioNeMo, les scientifiques peuvent commencer facilement à utiliser des modèles pré-entraînés, des téléchargeurs automatiques et des préprocesseurs pour UniRef50. et les bases de données ZINC, avec divers modèles, intégrations et sorties. La pré-formation non supervisée élimine également le besoin de données étiquetées pour générer rapidement des intégrations apprises qui prédisent la structure, la fonction, l'emplacement cellulaire, la solubilité dans l'eau et la liaison membranaire, économisant ainsi de la surface et des variables. zone, etc.
Parmi eux, MegaMolBART est un modèle de chimie générative formé à l'aide de 1,4 milliard de molécules (chaîne SMILES), qui peut être utilisée pour une variété d'applications informatiques chimiques, et BioNeMo fournit ProtT5 et ESM1 -85M et d'autres Transformer-. modèles de langage protéique basés sur
BioNeMo propose également OpenFold, un modèle d'apprentissage en profondeur pour prédire la structure 3D de nouvelles séquences de protéines.
Le modèle Transformer de NVIDIA lit des millions de séquences d'acides aminés. comprendre le texte et apprendre comment la nature construit les séquences d'acides aminés des protéines.
En ce qui concerne l'avenir, les perspectives d'utilisation de l'IA pour accélérer l'ingénierie des protéines sont très prometteuses par rapport à celles trouvées à l'origine dans la nature. plus stable et peut réaliser l'une de ses fonctions dans des conditions extrêmes telles que l'absence d'énergie ou une température élevée
.De plus, l'intelligence artificielle peut également être utilisée pour concevoir des séquences d'acides aminés adaptées au squelette, qui peuvent être utilisées pour améliorer la stabilité de protéines spécifiques telles que les enzymes et les anticorps. La technologie de l’intelligence artificielle joue un rôle très important dans la conception de protéines de différentes tailles et conformations. À l’avenir, elle pourra également contribuer à concevoir de plus en plus de protéines utiles, notamment de nouveaux matériaux biologiques pouvant être utilisés pour réduire la pollution et améliorer l’environnement. .
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!

Outils d'IA chauds

Undresser.AI Undress
Application basée sur l'IA pour créer des photos de nu réalistes

AI Clothes Remover
Outil d'IA en ligne pour supprimer les vêtements des photos.

Undress AI Tool
Images de déshabillage gratuites

Clothoff.io
Dissolvant de vêtements AI

AI Hentai Generator
Générez AI Hentai gratuitement.

Article chaud

Outils chauds

Bloc-notes++7.3.1
Éditeur de code facile à utiliser et gratuit

SublimeText3 version chinoise
Version chinoise, très simple à utiliser

Envoyer Studio 13.0.1
Puissant environnement de développement intégré PHP

Dreamweaver CS6
Outils de développement Web visuel

SublimeText3 version Mac
Logiciel d'édition de code au niveau de Dieu (SublimeText3)
 Quelle méthode est utilisée pour convertir les chaînes en objets dans vue.js?
Apr 07, 2025 pm 09:39 PM
Quelle méthode est utilisée pour convertir les chaînes en objets dans vue.js?
Apr 07, 2025 pm 09:39 PM
Lors de la conversion des chaînes en objets dans vue.js, JSON.Parse () est préféré pour les chaînes JSON standard. Pour les chaînes JSON non standard, la chaîne peut être traitée en utilisant des expressions régulières et réduisez les méthodes en fonction du format ou du codé décodé par URL. Sélectionnez la méthode appropriée en fonction du format de chaîne et faites attention aux problèmes de sécurité et d'encodage pour éviter les bogues.
 Vue et Element-UI Cascade déroulante Boîte en V Mode en V
Apr 07, 2025 pm 08:06 PM
Vue et Element-UI Cascade déroulante Boîte en V Mode en V
Apr 07, 2025 pm 08:06 PM
Vue et Element-UI Boîtes déroulantes en cascade Points de fosse de liaison V-model: V-model lie un tableau représentant les valeurs sélectionnées à chaque niveau de la boîte de sélection en cascade, pas une chaîne; La valeur initiale de SelectOptions doit être un tableau vide, non nul ou non défini; Le chargement dynamique des données nécessite l'utilisation de compétences de programmation asynchrones pour gérer les mises à jour des données en asynchrone; Pour les énormes ensembles de données, les techniques d'optimisation des performances telles que le défilement virtuel et le chargement paresseux doivent être prises en compte.
 Vue.js Comment convertir un tableau de type de chaîne en un tableau d'objets?
Apr 07, 2025 pm 09:36 PM
Vue.js Comment convertir un tableau de type de chaîne en un tableau d'objets?
Apr 07, 2025 pm 09:36 PM
Résumé: Il existe les méthodes suivantes pour convertir les tableaux de chaîne Vue.js en tableaux d'objets: Méthode de base: utilisez la fonction de carte pour convenir à des données formatées régulières. Gameplay avancé: l'utilisation d'expressions régulières peut gérer des formats complexes, mais ils doivent être soigneusement écrits et considérés. Optimisation des performances: Considérant la grande quantité de données, des opérations asynchrones ou des bibliothèques efficaces de traitement des données peuvent être utilisées. MEILLEUR PRATIQUE: Effacer le style de code, utilisez des noms de variables significatifs et des commentaires pour garder le code concis.
 Comment définir le délai de Vue Axios
Apr 07, 2025 pm 10:03 PM
Comment définir le délai de Vue Axios
Apr 07, 2025 pm 10:03 PM
Afin de définir le délai d'expiration de Vue Axios, nous pouvons créer une instance AxiOS et spécifier l'option Timeout: dans les paramètres globaux: vue.prototype. $ Axios = axios.create ({timeout: 5000}); Dans une seule demande: ce. $ axios.get ('/ api / utilisateurs', {timeout: 10000}).
 Comment utiliser MySQL après l'installation
Apr 08, 2025 am 11:48 AM
Comment utiliser MySQL après l'installation
Apr 08, 2025 am 11:48 AM
L'article présente le fonctionnement de la base de données MySQL. Tout d'abord, vous devez installer un client MySQL, tel que MySQLWorkBench ou le client de ligne de commande. 1. Utilisez la commande MySQL-UROot-P pour vous connecter au serveur et connecter avec le mot de passe du compte racine; 2. Utilisez Createdatabase pour créer une base de données et utilisez Sélectionner une base de données; 3. Utilisez CreateTable pour créer une table, définissez des champs et des types de données; 4. Utilisez InsertInto pour insérer des données, remettre en question les données, mettre à jour les données par mise à jour et supprimer les données par Supprimer. Ce n'est qu'en maîtrisant ces étapes, en apprenant à faire face à des problèmes courants et à l'optimisation des performances de la base de données que vous pouvez utiliser efficacement MySQL.
 Géospatial de Laravel: optimisation des cartes interactives et de grandes quantités de données
Apr 08, 2025 pm 12:24 PM
Géospatial de Laravel: optimisation des cartes interactives et de grandes quantités de données
Apr 08, 2025 pm 12:24 PM
Traiter efficacement 7 millions d'enregistrements et créer des cartes interactives avec la technologie géospatiale. Cet article explore comment traiter efficacement plus de 7 millions d'enregistrements en utilisant Laravel et MySQL et les convertir en visualisations de cartes interactives. Exigences initiales du projet de défi: extraire des informations précieuses en utilisant 7 millions d'enregistrements dans la base de données MySQL. Beaucoup de gens considèrent d'abord les langages de programmation, mais ignorent la base de données elle-même: peut-il répondre aux besoins? La migration des données ou l'ajustement structurel est-il requis? MySQL peut-il résister à une charge de données aussi importante? Analyse préliminaire: les filtres et les propriétés clés doivent être identifiés. Après analyse, il a été constaté que seuls quelques attributs étaient liés à la solution. Nous avons vérifié la faisabilité du filtre et établi certaines restrictions pour optimiser la recherche. Recherche de cartes basée sur la ville
 Les ingénieurs de backend senior à distance (plates-formes) ont besoin de cercles
Apr 08, 2025 pm 12:27 PM
Les ingénieurs de backend senior à distance (plates-formes) ont besoin de cercles
Apr 08, 2025 pm 12:27 PM
Ingénieur backend à distance Emploi Vacant Société: Emplacement du cercle: Bureau à distance Type d'emploi: Salaire à temps plein: 130 000 $ - 140 000 $ Description du poste Participez à la recherche et au développement des applications mobiles Circle et des fonctionnalités publiques liées à l'API couvrant l'intégralité du cycle de vie de développement logiciel. Les principales responsabilités complètent indépendamment les travaux de développement basés sur RubyOnRails et collaborent avec l'équipe frontale React / Redux / Relay. Créez les fonctionnalités de base et les améliorations des applications Web et travaillez en étroite collaboration avec les concepteurs et le leadership tout au long du processus de conception fonctionnelle. Promouvoir les processus de développement positifs et hiérarchiser la vitesse d'itération. Nécessite plus de 6 ans de backend d'applications Web complexe
 Comment résoudre MySQL ne peut pas être démarré
Apr 08, 2025 pm 02:21 PM
Comment résoudre MySQL ne peut pas être démarré
Apr 08, 2025 pm 02:21 PM
Il existe de nombreuses raisons pour lesquelles la startup MySQL échoue, et elle peut être diagnostiquée en vérifiant le journal des erreurs. Les causes courantes incluent les conflits de port (vérifier l'occupation du port et la configuration de modification), les problèmes d'autorisation (vérifier le service exécutant les autorisations des utilisateurs), les erreurs de fichier de configuration (vérifier les paramètres des paramètres), la corruption du répertoire de données (restaurer les données ou reconstruire l'espace de la table), les problèmes d'espace de la table InNODB (vérifier les fichiers IBDATA1), la défaillance du chargement du plug-in (vérification du journal des erreurs). Lors de la résolution de problèmes, vous devez les analyser en fonction du journal d'erreur, trouver la cause profonde du problème et développer l'habitude de sauvegarder régulièrement les données pour prévenir et résoudre des problèmes.





