 Périphériques technologiques
Périphériques technologiques
 IA
IA
 Meta permet à un modèle de langage de 15 milliards de paramètres d'apprendre à concevoir de « nouvelles » protéines à partir de zéro ! LeCun : des résultats étonnants
Meta permet à un modèle de langage de 15 milliards de paramètres d'apprendre à concevoir de « nouvelles » protéines à partir de zéro ! LeCun : des résultats étonnants
Meta permet à un modèle de langage de 15 milliards de paramètres d'apprendre à concevoir de « nouvelles » protéines à partir de zéro ! LeCun : des résultats étonnants
L'IA a encore une fois réalisé de nouveaux progrès dans le domaine de la biomédecine. Oui, cette fois, il s’agit de protéines.
La différence est que dans le passé, l'IA découvrait des structures protéiques, mais cette fois, elle a commencé à concevoir et à générer elle-même des structures protéiques. S'il était un « procureur » dans le passé, il n'est pas impossible de dire qu'il est désormais devenu un « créateur ».
L’équipe de recherche sur les protéines de FAIR, qui fait partie de l’institut de recherche en IA de Meta, participe à cette étude. En tant que scientifique en chef de l'IA ayant travaillé pendant de nombreuses années chez Facebook, Yann LeCun a également immédiatement transmis les résultats de sa propre équipe et en a fait l'éloge.

Ces deux articles sur BioRxiv sont les résultats « étonnants » de Meta dans la conception/génération de protéines. Le système utilise un algorithme d'hybridation simulé pour trouver une séquence d'acides aminés qui se plie d'une manière qui se conforme à une forme souhaitée ou satisfait des contraintes telles que la symétrie.
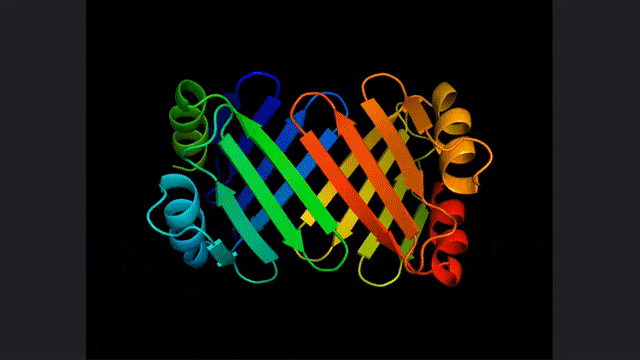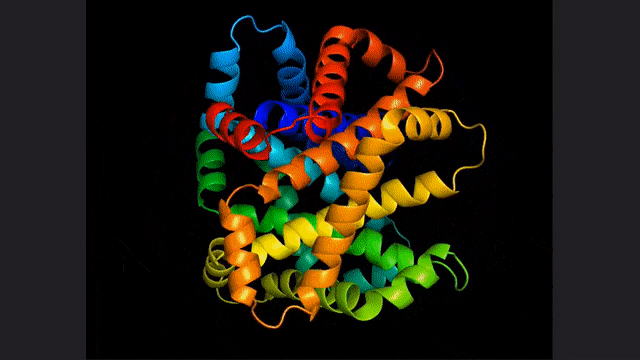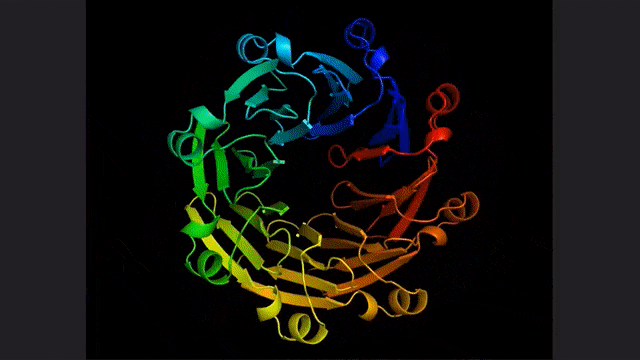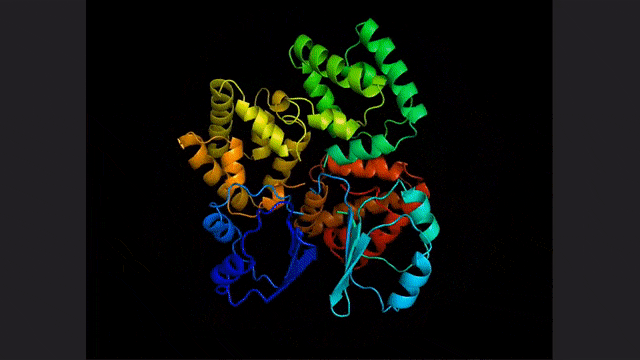
ESM2, un modèle pour la prédiction de la structure hiérarchique atomique
Vous l'avez bien deviné, la base de cette recherche et de ces deux articles est le grand modèle linguistique pour la prédiction et la découverte des protéines proposé par Meta il n'y a pas si longtemps :ESM2 .
Il s'agit d'un grand modèle avec 15 milliards de paramètres. À mesure que le modèle évolue de 8 à 15 millions de paramètres, les informations émergeant de la représentation interne permettent des prédictions de structure tridimensionnelle à une résolution atomique.
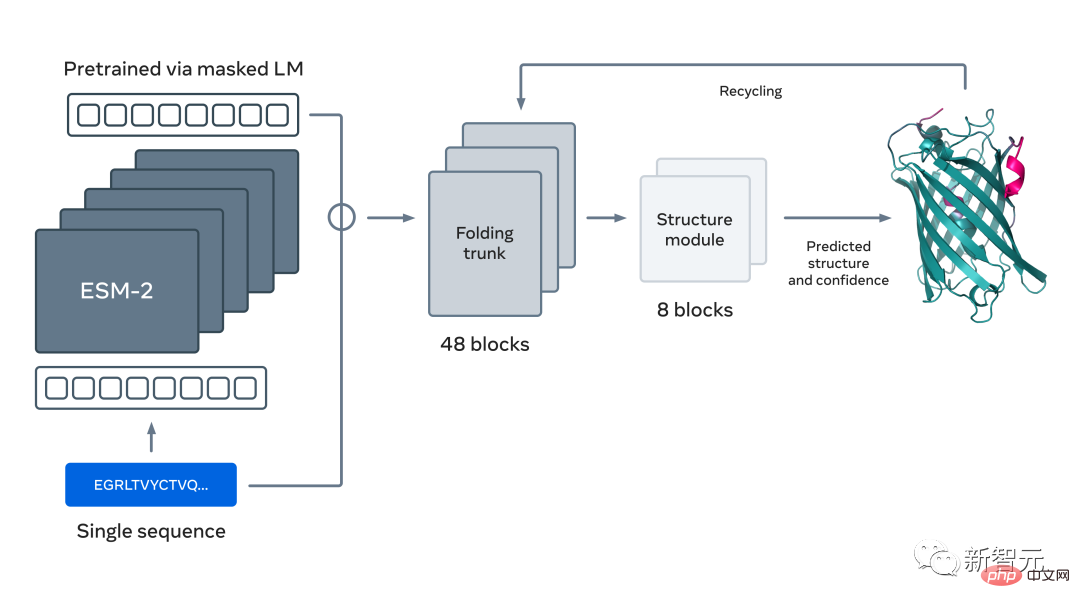
En utilisant de grands modèles de langage pour apprendre des modèles évolutifs, des prédictions de structure précises peuvent être générées de bout en bout directement à partir de séquences de protéines, prédisant plus rapidement que les méthodes de pointe actuelles tout en maintenant la précision 60 fois.
En fait, grâce à cette nouvelle capacité de prédiction de structure, Meta a prédit plus de 600 millions de protéines métagénomiques sur la carte en seulement deux semaines à l'aide d'un cluster d'environ 2 000 séquences GPU.
Alex Rives de Meta AI, l'auteur correspondant des deux articles, a déclaré que le modèle de langage ESM2 démontre une polyvalence non seulement au-delà de la portée des protéines naturelles, mais peut également générer de manière programmable une structure de protéines complexe et modulaire.
Protein Design "Langage de programmation spécialisé"
Si un travailleur veut bien faire son travail, il doit d'abord affûter ses outils.
Afin de rendre la conception et la génération de protéines plus efficaces, les chercheurs ont également développé un langage de programmation de haut niveau spécifiquement pour la conception de protéines basé sur des résultats antérieurs (principalement ESM2).
Adresse papier : https://www.biorxiv.org/content/10.1101/2022.12.21.521526v1
L'un des principaux leaders de cette recherche, le papier « Une programmation de haut niveau langage" Alex Rives, l'auteur correspondant de "For Generative Protein Design", a déclaré sur les réseaux sociaux que ce résultat permet de programmer la génération de grandes protéines et de complexes avec des structures complexes et modulaires.
Brian Hie, l'un des auteurs de l'article et chercheur à l'Université de Stanford, a également expliqué les principales idées de recherche et les résultats de cet article sur Twitter.
Dans l'ensemble, cet article décrit comment l'apprentissage automatique génératif permet la conception modulaire de protéines complexes contrôlées par des langages de programmation de haut niveau pour la conception de protéines.
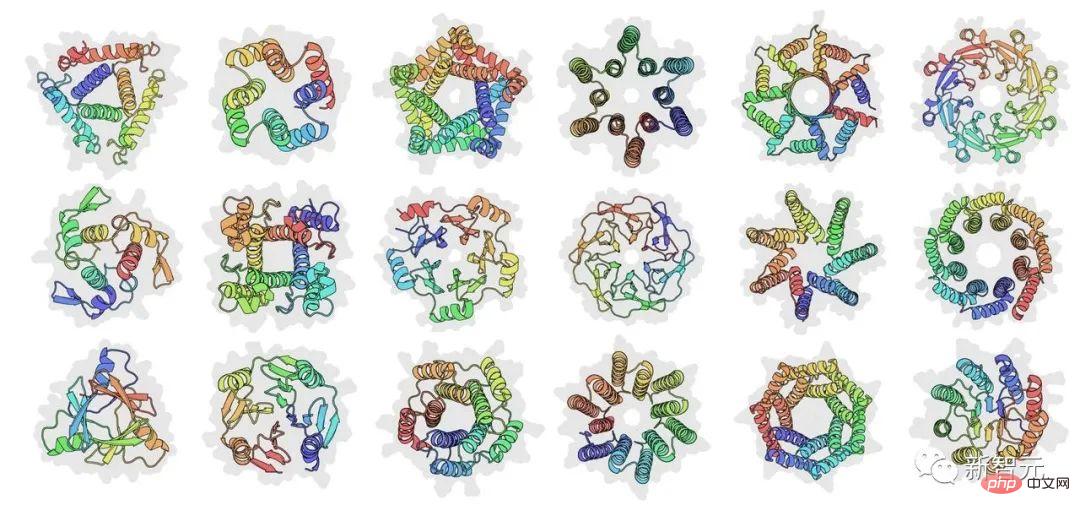
Il a déclaré que l'idée principale de l'article n'est pas d'utiliser des blocs de construction séquentiels ou structurels, mais de placer la modularité à un niveau d'abstraction plus élevé et de laisser l'optimisation de la boîte noire générer des conceptions spécifiques. La structure au niveau atomique est prédite à chaque étape de l’optimisation.
Par rapport aux méthodes de conception de protéines précédentes, cette nouvelle idée crée une méthode qui permet aux concepteurs de spécifier des contraintes arbitraires et non différenciables, allant de la spécification de coordonnées au niveau atomique à des plans de conception abstraits pour les protéines, tels que conception symétrique.
Il est important pour la programmabilité que les contraintes soient modulaires. Par exemple, la figure ci-dessous montre la situation où la même contrainte est appliquée hiérarchiquement à deux niveaux de programmation de symétrie.
Ces contraintes sont également faciles à recombiner. Par exemple, les contraintes sur les coordonnées atomiques peuvent être combinées avec des contraintes sur la symétrie. Différentes formes de symétrie à deux niveaux peuvent également être combinées pour programmer une structure composite asymétrique.
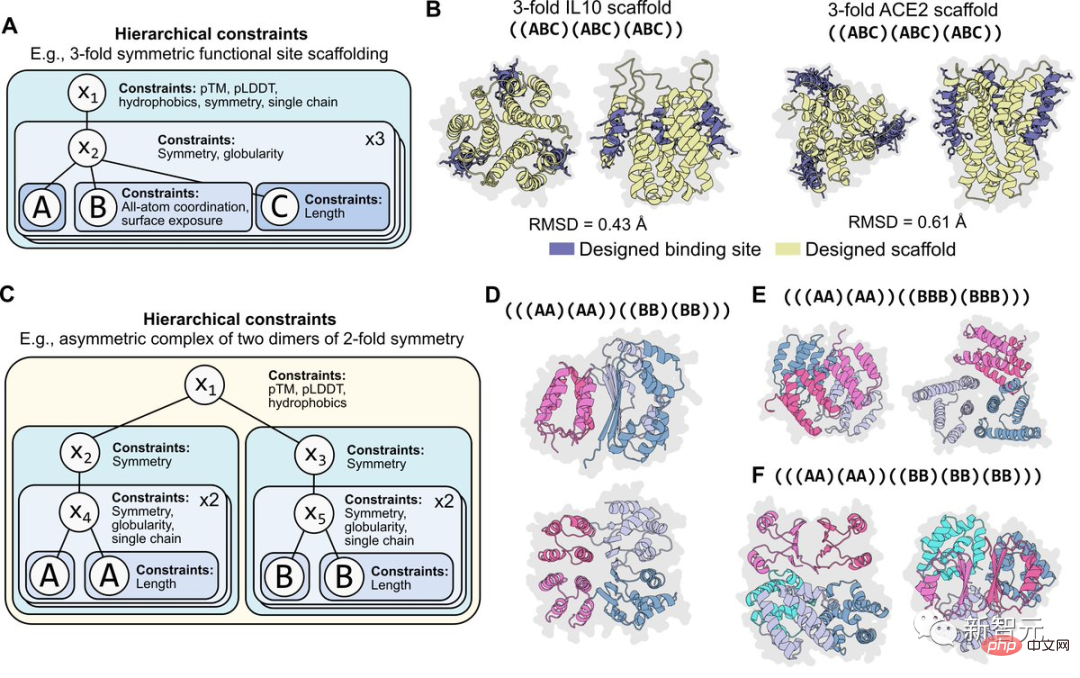
Brian Hie estime que cette réalisation est une étape vers une conception de protéines plus contrôlable, régulière et expressive. Il a également remercié Meta AI et d'autres collaborateurs pour leurs efforts conjoints.
Faites de la conception de protéines « comme la construction d'un bâtiment »
Dans l'article, les chercheurs pensent que la conception de protéines bénéficiera de la régularité, de la simplicité et de la programmabilité fournies par un ensemble de concepts abstraits de base comme ceux utilisés. dans la construction, les machines, les circuits et le génie logiciel.
Mais contrairement à ces créations artificielles, les protéines ne peuvent pas être décomposées en parties facilement réorganisées car la structure locale de la séquence est intriquée avec son environnement global. La conception classique des protéines ab initio tente d’identifier un ensemble d’éléments structurels de base, puis de les assembler en structures d’ordre supérieur.
De même, l'ingénierie protéique traditionnelle recombine souvent des fragments ou des domaines de séquences protéiques natives en chimères hybrides. Cependant, les approches existantes ne sont pas encore capables d’atteindre la complexité combinatoire élevée requise pour une véritable programmabilité.
Cet article démontre que les modèles génératifs modernes atteignent les objectifs classiques de modularité et de programmabilité à de nouveaux niveaux de complexité combinatoire. Plaçant la modularité et la programmabilité à un niveau d'abstraction plus élevé, les modèles génératifs comblent le fossé entre l'intuition humaine et la génération de séquences et de structures spécifiques.
Dans ce cas, le concepteur de protéines n'a qu'à réassembler les instructions de haut niveau, et la tâche d'obtenir une protéine qui satisfait à ces instructions est placée sur le modèle génératif.
Les chercheurs proposent un langage de programmation pour la conception générative de protéines qui permet aux concepteurs de spécifier des procédures intuitives, modulaires et hiérarchiques. Les programmes de haut niveau peuvent être transformés en séquences et structures de bas niveau grâce à des modèles génératifs. Cette approche exploite les progrès des modèles de langage protéique, qui peuvent apprendre des informations structurelles et des principes de conception pour les protéines.
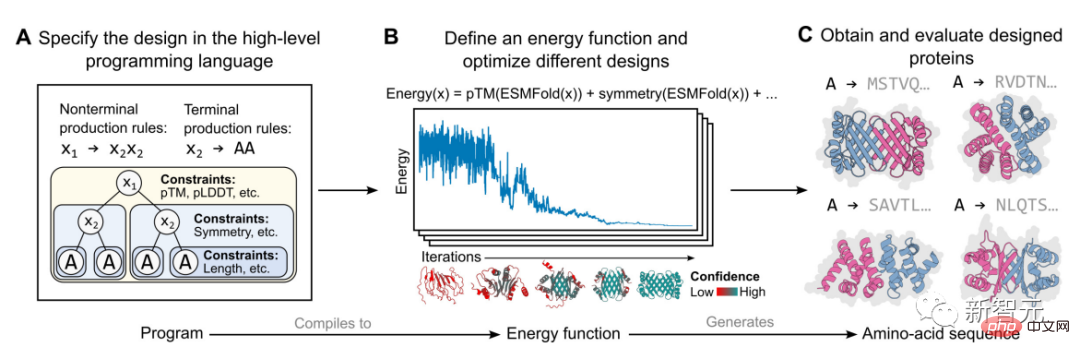
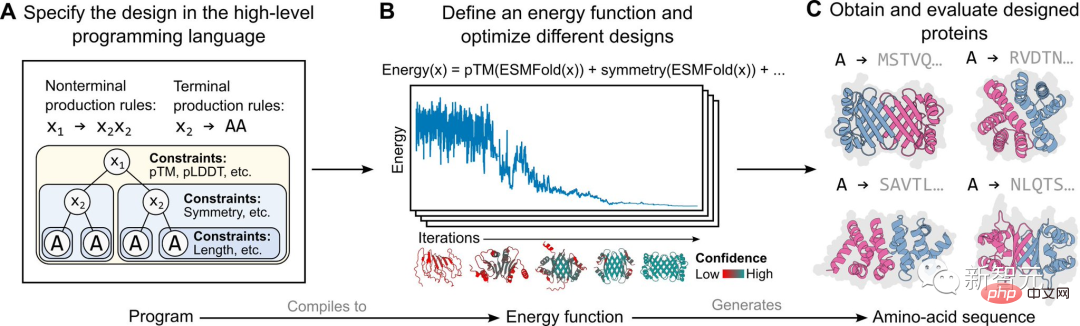
La mise en œuvre spécifique dans cette étude est basée sur un modèle de production basé sur l'énergie, comme le montre la figure ci-dessus.
Tout d'abord, un concepteur de protéines spécifie un programme de haut niveau constitué d'un ensemble de contraintes organisées hiérarchiquement (Figure A).
Ce programme se compile ensuite en une fonction énergétique qui évalue la compatibilité avec les contraintes, qui peuvent être arbitraires et indiscernables (Figure B).
Appliquez des contraintes structurelles en incorporant des prédictions de structure au niveau atomique (activées par des modèles de langage) dans des fonctions énergétiques. Cette approche est capable de générer une large gamme de conceptions complexes (Figure C).
Générer des séquences de protéines à partir de zéro
Dans l'article "Les modèles de langage se généralisent au-delà des protéines naturelles", Tom Sercu, l'auteur de l'équipe MetaAI, a déclaré que ce travail accomplissait principalement deux tâches.

Adresse papier : https://www.biorxiv.org/content/10.1101/2022.12.21.521521v1
Le premier élément consiste à concevoir une séquence pour une structure de chaîne principale donnée. En utilisant un modèle de langage, une conception réussie pour tous les objectifs peut être obtenue, avec un taux de réussite de 19/20, tandis qu'une conception séquentielle sans la participation d'un modèle de langage a un taux de réussite de seulement 1/20.
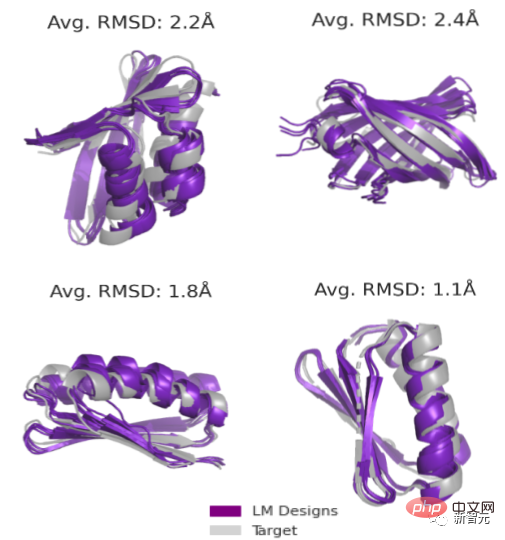
La deuxième tâche est la génération sans contrainte. L’équipe de recherche propose une nouvelle méthode d’échantillonnage de couples (séquence, structure) à partir d’un paysage énergétique défini par un modèle de langage.

L'échantillonnage à travers différentes topologies augmente encore une fois le taux de réussite de l'expérience (jusqu'à 71/129 ou 55%).
Pour prouver que la structure protéique prédite transcende les limites des protéines naturelles, l'équipe de recherche a recherché les séquences protéiques générées par le modèle de langage dans une base de données de séquences couvrant toutes les protéines naturelles connues.
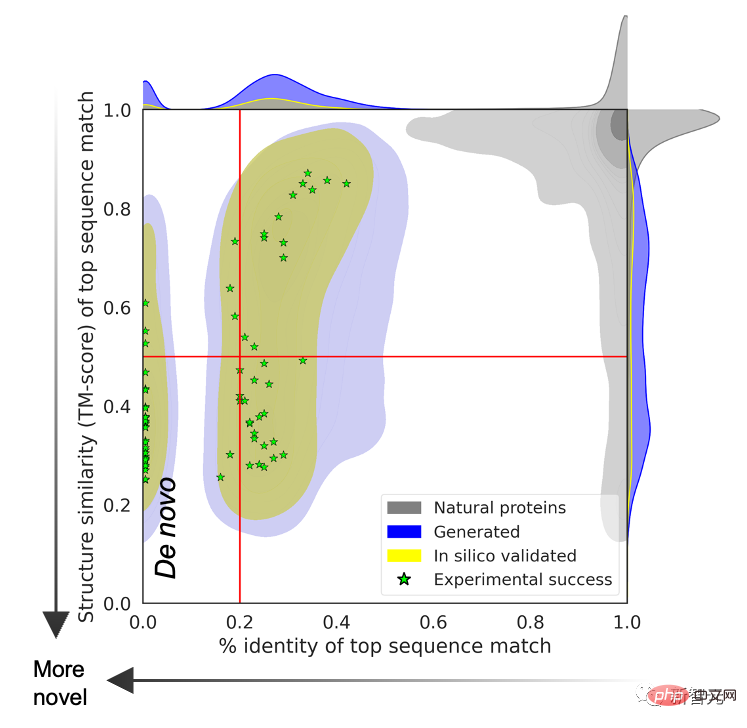
Les résultats montrent qu'il n'y a pas de relation de correspondance entre les deux et que les structures de prédiction générées par les séquences naturelles et les modèles de langage sont différentes.
Sercu a déclaré que la structure de la protéine peut être conçue en utilisant uniquement le modèle de langage protéique ESM2. L’équipe de recherche a testé expérimentalement 228 protéines, avec un taux de réussite de 67 % !
Sercu estime que les modèles de langage protéique formés uniquement sur des séquences peuvent apprendre des modèles profonds reliant la séquence et la structure et peuvent être utilisés pour concevoir des protéines à partir de zéro, au-delà de l'espace de conception naturellement exploré.
Explorer la grammaire profonde de la production de protéines
Dans l'article, les chercheurs de Meta ont déclaré que même si le modèle de langage n'est formé que sur des séquences, le modèle peut toujours concevoir la structure grammaticale profonde des protéines, dépassant ainsi les limites de protéines naturelles.
Si les carrés de la figure A représentent l'espace composé de toutes les séquences protéiques, alors la séquence protéique naturelle est la partie grise, qui en recouvre une petite partie. Afin de généraliser au-delà des séquences naturelles, les modèles de langage doivent accéder aux modèles de conception sous-jacents.
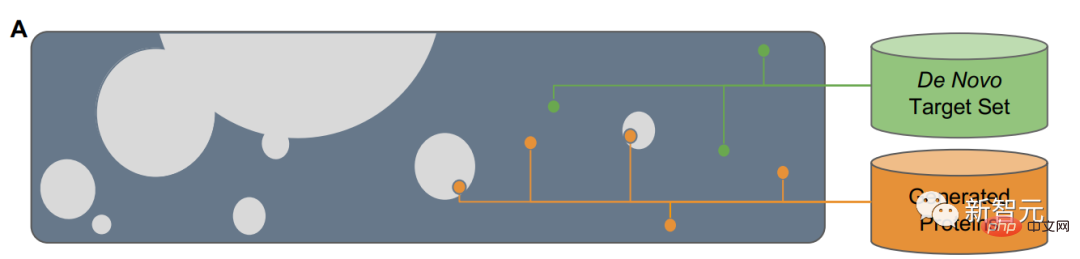
Ce que l'équipe de recherche doit faire, c'est deux choses : premièrement, concevoir le squelette protéique (de novo) à partir de zéro, et deuxièmement, générer la séquence protéique à partir de zéro en fonction du squelette.
L'équipe de recherche a utilisé un modèle de langage de masque pour entraîner ESM2, et le contenu de la formation comprenait des millions de protéines naturelles différentes au cours du processus d'évolution.
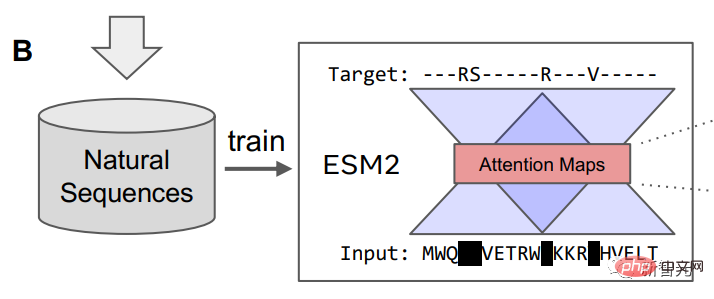
Une fois le modèle de langage formé, des informations sur la structure tertiaire de la protéine peuvent être identifiées dans l'état d'attention interne du modèle. Ensuite, les chercheurs ont converti l’attention de deux positions dans la séquence protéique en une distribution de distances entre les résidus par projection linéaire.
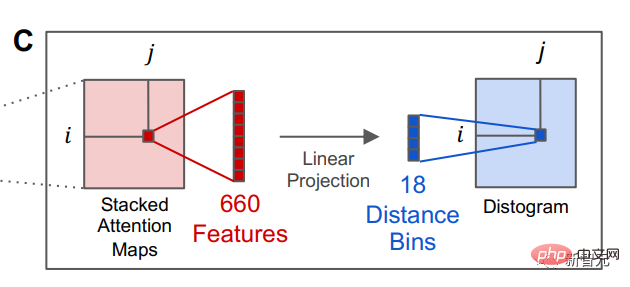
La capacité des modèles de langage à prédire les structures protéiques indique les séquences structurelles plus profondes qui sous-tendent les séquences protéiques naturelles, et la possibilité qu'il existe une grammaire profonde qui puisse être apprise par le modèle, ont déclaré les chercheurs. .
Les résultats montrent qu'au cours du processus d'évolution, un grand nombre de séquences protéiques contiennent des structures et des fonctions biologiques, révélant la structure de conception des protéines. Cette construction est entièrement reproductible par l’apprentissage automatique de modèles de séquences protéiques.
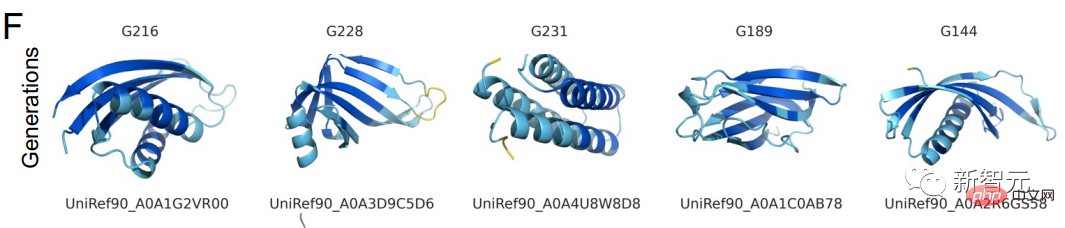
Les structures protéiques ont été prédites avec succès par des modèles de langage dans 6 expériences
L'existence d'une grammaire profonde entre les protéines explique deux ensembles de résultats apparemment contradictoires : la compréhension des protéines natives dépend des données d'entraînement à son tour, le modèle linguistique peut prédire et explorer au-delà des familles de protéines naturelles connues.
Si la loi d'échelle des modèles de langage protéiques continue d'être efficace, on peut s'attendre à ce que les capacités de génération des modèles de langage d'IA continuent de s'améliorer.
L'équipe de recherche a déclaré qu'en raison de l'existence de la grammaire de base de la structure des protéines, le modèle de machine apprendra des structures protéiques plus rares, élargissant ainsi la capacité de prédiction et l'espace d'exploration du modèle.
Il y a un an, AlphaFold2, l'open source de DeepMind, était lancé dans les domaines de la nature et des sciences, bouleversant les cercles universitaires de la biologie et de l'IA.
Un an plus tard, des modèles de prédiction par intelligence artificielle ont vu le jour, comblant fréquemment des lacunes dans le domaine de la structure des protéines.
Si les humains donnent vie à l'intelligence artificielle, l'intelligence artificielle est-elle la dernière pièce du puzzle permettant aux humains de compléter le mystère de la vie ?
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!

Outils d'IA chauds

Undresser.AI Undress
Application basée sur l'IA pour créer des photos de nu réalistes

AI Clothes Remover
Outil d'IA en ligne pour supprimer les vêtements des photos.

Undress AI Tool
Images de déshabillage gratuites

Clothoff.io
Dissolvant de vêtements AI

AI Hentai Generator
Générez AI Hentai gratuitement.

Article chaud

Outils chauds

Bloc-notes++7.3.1
Éditeur de code facile à utiliser et gratuit

SublimeText3 version chinoise
Version chinoise, très simple à utiliser

Envoyer Studio 13.0.1
Puissant environnement de développement intégré PHP

Dreamweaver CS6
Outils de développement Web visuel

SublimeText3 version Mac
Logiciel d'édition de code au niveau de Dieu (SublimeText3)
 Le modèle MoE open source le plus puissant au monde est ici, avec des capacités chinoises comparables à celles du GPT-4, et le prix ne représente que près d'un pour cent de celui du GPT-4-Turbo.
May 07, 2024 pm 04:13 PM
Le modèle MoE open source le plus puissant au monde est ici, avec des capacités chinoises comparables à celles du GPT-4, et le prix ne représente que près d'un pour cent de celui du GPT-4-Turbo.
May 07, 2024 pm 04:13 PM
Imaginez un modèle d'intelligence artificielle qui non seulement a la capacité de surpasser l'informatique traditionnelle, mais qui permet également d'obtenir des performances plus efficaces à moindre coût. Ce n'est pas de la science-fiction, DeepSeek-V2[1], le modèle MoE open source le plus puissant au monde est ici. DeepSeek-V2 est un puissant mélange de modèle de langage d'experts (MoE) présentant les caractéristiques d'une formation économique et d'une inférence efficace. Il est constitué de 236B paramètres, dont 21B servent à activer chaque marqueur. Par rapport à DeepSeek67B, DeepSeek-V2 offre des performances plus élevées, tout en économisant 42,5 % des coûts de formation, en réduisant le cache KV de 93,3 % et en augmentant le débit de génération maximal à 5,76 fois. DeepSeek est une entreprise explorant l'intelligence artificielle générale
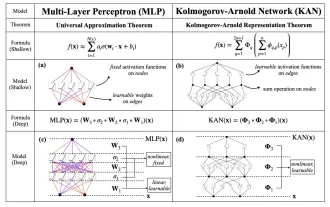 KAN, qui remplace MLP, a été étendu à la convolution par des projets open source
Jun 01, 2024 pm 10:03 PM
KAN, qui remplace MLP, a été étendu à la convolution par des projets open source
Jun 01, 2024 pm 10:03 PM
Plus tôt ce mois-ci, des chercheurs du MIT et d'autres institutions ont proposé une alternative très prometteuse au MLP – KAN. KAN surpasse MLP en termes de précision et d’interprétabilité. Et il peut surpasser le MLP fonctionnant avec un plus grand nombre de paramètres avec un très petit nombre de paramètres. Par exemple, les auteurs ont déclaré avoir utilisé KAN pour reproduire les résultats de DeepMind avec un réseau plus petit et un degré d'automatisation plus élevé. Plus précisément, le MLP de DeepMind compte environ 300 000 paramètres, tandis que le KAN n'en compte qu'environ 200. KAN a une base mathématique solide comme MLP est basé sur le théorème d'approximation universelle, tandis que KAN est basé sur le théorème de représentation de Kolmogorov-Arnold. Comme le montre la figure ci-dessous, KAN a
 Bonjour, Atlas électrique ! Le robot Boston Dynamics revient à la vie, des mouvements étranges à 180 degrés effraient Musk
Apr 18, 2024 pm 07:58 PM
Bonjour, Atlas électrique ! Le robot Boston Dynamics revient à la vie, des mouvements étranges à 180 degrés effraient Musk
Apr 18, 2024 pm 07:58 PM
Boston Dynamics Atlas entre officiellement dans l’ère des robots électriques ! Hier, l'Atlas hydraulique s'est retiré "en larmes" de la scène de l'histoire. Aujourd'hui, Boston Dynamics a annoncé que l'Atlas électrique était au travail. Il semble que dans le domaine des robots humanoïdes commerciaux, Boston Dynamics soit déterminé à concurrencer Tesla. Après la sortie de la nouvelle vidéo, elle a déjà été visionnée par plus d’un million de personnes en seulement dix heures. Les personnes âgées partent et de nouveaux rôles apparaissent. C'est une nécessité historique. Il ne fait aucun doute que cette année est l’année explosive des robots humanoïdes. Les internautes ont commenté : Les progrès des robots ont fait ressembler la cérémonie d'ouverture de cette année à des êtres humains, et le degré de liberté est bien plus grand que celui des humains. Mais n'est-ce vraiment pas un film d'horreur ? Au début de la vidéo, Atlas est allongé calmement sur le sol, apparemment sur le dos. Ce qui suit est à couper le souffle
 Les robots Tesla travaillent dans les usines, Musk : Le degré de liberté des mains atteindra 22 cette année !
May 06, 2024 pm 04:13 PM
Les robots Tesla travaillent dans les usines, Musk : Le degré de liberté des mains atteindra 22 cette année !
May 06, 2024 pm 04:13 PM
La dernière vidéo du robot Optimus de Tesla est sortie, et il peut déjà fonctionner en usine. À vitesse normale, il trie les batteries (les batteries 4680 de Tesla) comme ceci : Le responsable a également publié à quoi cela ressemble à une vitesse 20 fois supérieure - sur un petit "poste de travail", en sélectionnant et en sélectionnant et en sélectionnant : Cette fois, il est publié L'un des points forts de la vidéo est qu'Optimus réalise ce travail en usine, de manière totalement autonome, sans intervention humaine tout au long du processus. Et du point de vue d'Optimus, il peut également récupérer et placer la batterie tordue, en se concentrant sur la correction automatique des erreurs : concernant la main d'Optimus, le scientifique de NVIDIA Jim Fan a donné une évaluation élevée : la main d'Optimus est l'un des robots à cinq doigts du monde. le plus adroit. Ses mains ne sont pas seulement tactiles
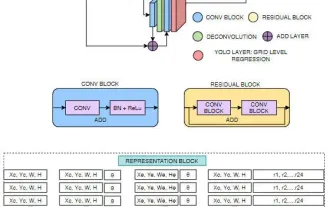 FisheyeDetNet : le premier algorithme de détection de cible basé sur une caméra fisheye
Apr 26, 2024 am 11:37 AM
FisheyeDetNet : le premier algorithme de détection de cible basé sur une caméra fisheye
Apr 26, 2024 am 11:37 AM
La détection de cibles est un problème relativement mature dans les systèmes de conduite autonome, parmi lesquels la détection des piétons est l'un des premiers algorithmes à être déployés. Des recherches très complètes ont été menées dans la plupart des articles. Cependant, la perception de la distance à l’aide de caméras fisheye pour une vue panoramique est relativement moins étudiée. En raison de la distorsion radiale importante, la représentation standard du cadre de délimitation est difficile à mettre en œuvre dans les caméras fisheye. Pour alléger la description ci-dessus, nous explorons les conceptions étendues de boîtes englobantes, d'ellipses et de polygones généraux dans des représentations polaires/angulaires et définissons une métrique de segmentation d'instance mIOU pour analyser ces représentations. Le modèle fisheyeDetNet proposé avec une forme polygonale surpasse les autres modèles et atteint simultanément 49,5 % de mAP sur l'ensemble de données de la caméra fisheye Valeo pour la conduite autonome.
 Un nouveau casque VR Meta Quest 3S abordable apparaît sur FCC, suggérant un lancement imminent
Sep 04, 2024 am 06:51 AM
Un nouveau casque VR Meta Quest 3S abordable apparaît sur FCC, suggérant un lancement imminent
Sep 04, 2024 am 06:51 AM
L'événement Meta Connect 2024 est prévu du 25 au 26 septembre et lors de cet événement, la société devrait dévoiler un nouveau casque de réalité virtuelle abordable. Selon la rumeur, il s'agirait du Meta Quest 3S, le casque VR serait apparemment apparu sur la liste FCC. Cela suggère
 Le premier modèle open source à dépasser le niveau GPT4o ! Llama 3.1 fuite : 405 milliards de paramètres, liens de téléchargement et cartes de modèles sont disponibles
Jul 23, 2024 pm 08:51 PM
Le premier modèle open source à dépasser le niveau GPT4o ! Llama 3.1 fuite : 405 milliards de paramètres, liens de téléchargement et cartes de modèles sont disponibles
Jul 23, 2024 pm 08:51 PM
Préparez votre GPU ! Llama3.1 est finalement apparu, mais la source n'est pas officielle de Meta. Aujourd'hui, la nouvelle divulguée du nouveau grand modèle Llama est devenue virale sur Reddit. En plus du modèle de base, elle comprend également des résultats de référence de 8B, 70B et le paramètre maximum de 405B. La figure ci-dessous montre les résultats de comparaison de chaque version de Llama3.1 avec OpenAIGPT-4o et Llama38B/70B. On peut voir que même la version 70B dépasse GPT-4o sur plusieurs benchmarks. Source de l'image : https://x.com/mattshumer_/status/1815444612414087294 Évidemment, version 3.1 de 8B et 70
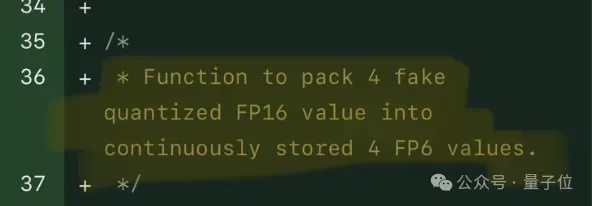 Une seule carte exécute Llama 70B plus rapidement que deux cartes, Microsoft vient de mettre le FP6 dans l'Open source A100 |
Apr 29, 2024 pm 04:55 PM
Une seule carte exécute Llama 70B plus rapidement que deux cartes, Microsoft vient de mettre le FP6 dans l'Open source A100 |
Apr 29, 2024 pm 04:55 PM
Le FP8 et la précision de quantification inférieure en virgule flottante ne sont plus le « brevet » du H100 ! Lao Huang voulait que tout le monde utilise INT8/INT4, et l'équipe Microsoft DeepSpeed a commencé à exécuter FP6 sur A100 sans le soutien officiel de NVIDIA. Les résultats des tests montrent que la quantification FP6 de la nouvelle méthode TC-FPx sur A100 est proche ou parfois plus rapide que celle de INT4, et a une précision supérieure à celle de cette dernière. En plus de cela, il existe également une prise en charge de bout en bout des grands modèles, qui ont été open source et intégrés dans des cadres d'inférence d'apprentissage profond tels que DeepSpeed. Ce résultat a également un effet immédiat sur l'accélération des grands modèles : dans ce cadre, en utilisant une seule carte pour exécuter Llama, le débit est 2,65 fois supérieur à celui des cartes doubles. un





