 Périphériques technologiques
Périphériques technologiques
 IA
IA
 L'équipe de l'Académie chinoise des sciences crée un cadre unifié pour améliorer la précision des prévisions des paramètres cinétiques des enzymes
L'équipe de l'Académie chinoise des sciences crée un cadre unifié pour améliorer la précision des prévisions des paramètres cinétiques des enzymes
L'équipe de l'Académie chinoise des sciences crée un cadre unifié pour améliorer la précision des prévisions des paramètres cinétiques des enzymes
Éditeur | Radis Skin
La prédiction des paramètres cinétiques des enzymes est cruciale pour la conception et l'optimisation des enzymes dans les applications biotechnologiques et industrielles, mais les performances limitées des outils de prédiction actuels sur diverses tâches limitent leur application pratique.
Des chercheurs de l'Académie chinoise des sciences ont récemment proposé UniKP, un cadre unifié basé sur des modèles linguistiques pré-entraînés qui peuvent être utilisés pour prédire les paramètres cinétiques des enzymes, notamment le nombre de renouvellement enzymatique (kcat), la constante de Michaelis-Menten (Km) et le catalyseur. efficacité ( kcat/Km), ces paramètres sont obtenus à partir de la séquence protéique et de la structure du substrat.
Un cadre à deux couches basé sur UniKP (EF-UniKP) est également proposé, qui peut prédire de manière stable les valeurs kcat en tenant compte de facteurs environnementaux tels que le pH et la température. Dans le même temps, l’équipe de recherche a également exploré systématiquement quatre méthodes de repondération représentatives, réduisant ainsi les erreurs de prédiction dans les tâches de prédiction de grande valeur.
L'étude s'intitule « UniKP : un cadre unifié pour la prédiction des paramètres cinétiques enzymatiques » et a été publiée dans la revue « Nature Communications » le 11 décembre 2023.

L'étude de l'efficacité catalytique des enzymes sur des substrats spécifiques est un enjeu important en biologie et a un impact profond sur l'évolution des enzymes, l'ingénierie métabolique et la biologie synthétique. Les données expérimentales in vitro mesurant kcat et Km, ainsi que le taux de renouvellement maximal et la constante de Michaelis-Menten, peuvent être utilisées comme indicateurs pour mesurer l'efficacité des enzymes dans la catalyse de réactions spécifiques et pour comparer les activités catalytiques relatives de différentes enzymes.
À l'heure actuelle, la mesure des paramètres cinétiques des enzymes repose principalement sur des mesures expérimentales, qui prennent du temps, sont coûteuses et demandent beaucoup de main d'œuvre, ce qui donne lieu à une petite base de données de valeurs de paramètres cinétiques mesurées expérimentalement. Par exemple, la base de données de séquences UniProt contient plus de 230 millions de séquences enzymatiques, tandis que les bases de données enzymatiques BRENDA et SABIO-RK contiennent des dizaines de milliers de valeurs kcat mesurées expérimentalement. L'intégration des identifiants Uniprot dans ces bases de données enzymatiques facilite la connexion entre les paramètres mesurés et les séquences protéiques. Cependant, l’échelle de ces connexions est encore beaucoup plus petite par rapport au nombre de séquences enzymatiques, ce qui limite les progrès dans les applications en aval telles que l’évolution dirigée et l’ingénierie métabolique.
Cadre de prédiction des paramètres cinétiques enzymatiques
Dans cette étude, des chercheurs de l'Académie chinoise des sciences ont proposé un nouveau cadre appelé UniKP, qui est basé sur des modèles de langage pré-entraînés et vise à améliorer la précision de la prédiction des paramètres cinétiques enzymatiques. . Ces paramètres incluent kcat, Km et kcat/Km, qui peuvent être prédits compte tenu de la séquence enzymatique et de la structure du substrat. Les chercheurs ont effectué une comparaison complète de 16 modèles d’apprentissage automatique différents et de 2 modèles d’apprentissage profond et ont constaté qu’UniKP fonctionnait bien en termes de précision des prédictions. Cette recherche devrait fournir de nouveaux outils et méthodes pour la recherche et les applications dans le domaine de la cinétique enzymatique.
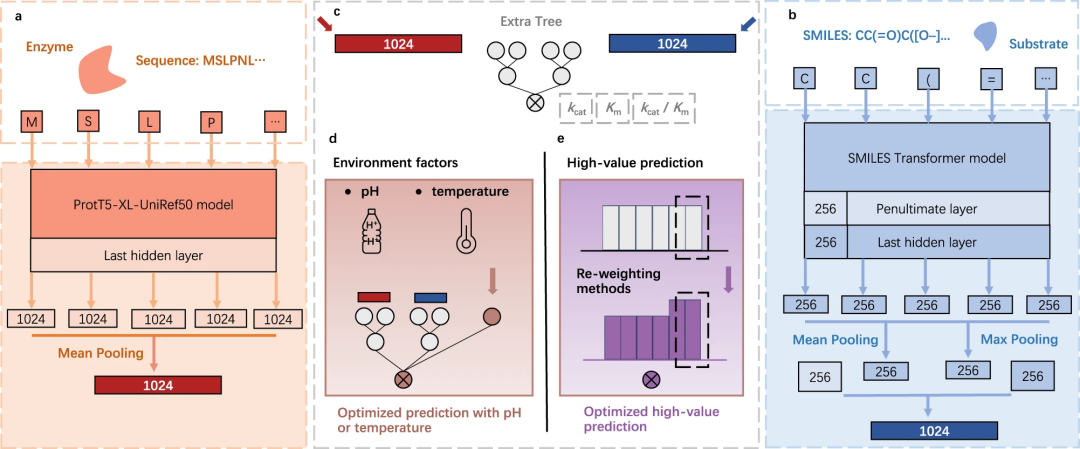
Illustration : aperçu de l'UniKP. (Source : article)
Par rapport au précédent modèle de pointe DLKcat, UniKP montre des performances supérieures dans la tâche de prédiction kcat, avec un coefficient de détermination moyen de 0,68, soit une amélioration de 20 %. Les chercheurs supposent que les modèles pré-entraînés ont contribué de manière significative aux performances d'UniKP en utilisant des informations non supervisées provenant de l'ensemble de la base de données pour créer des représentations faciles à apprendre des séquences enzymatiques et des structures de substrat.
L'analyse de l'apprentissage des modèles montre que l'information sur les protéines a un rôle dominant, peut-être en raison de la complexité de la structure de l'enzyme par rapport à la structure du substrat. De plus, UniKP peut capturer efficacement de petites différences dans les valeurs kcat entre les enzymes et leurs mutants, y compris les cas mesurés expérimentalement, ce qui est crucial pour la conception et la modification des enzymes. La différence entre le R^2 des prédictions UniKP et le R^2 de la méthode gmean pour les régions à identité élevée et faible démontre la capacité d'UniKP à extraire des informations interconnectées plus profondes et ainsi à bien performer dans ces tâches.
Cadre à deux couches EF-UniKP
La plupart des modèles actuels ne prennent pas en compte les facteurs environnementaux, ce qui constitue une limitation clé dans la simulation de conditions expérimentales réelles. Pour résoudre ce problème, les chercheurs ont proposé un cadre à deux couches EF-UniKP, qui prend en compte les facteurs environnementaux. Basé sur deux ensembles de données nouvellement construits contenant respectivement des informations sur le pH et la température, EF-UniKP présente des performances améliorées par rapport à l'UniKP initial. Il s’agit d’une prédiction kcat précise, à haut débit, indépendante de l’organisme et dépendant du contexte. De plus, cette approche a le potentiel d’être élargie pour inclure d’autres facteurs tels que le co-substrat et la concentration de NaCl.
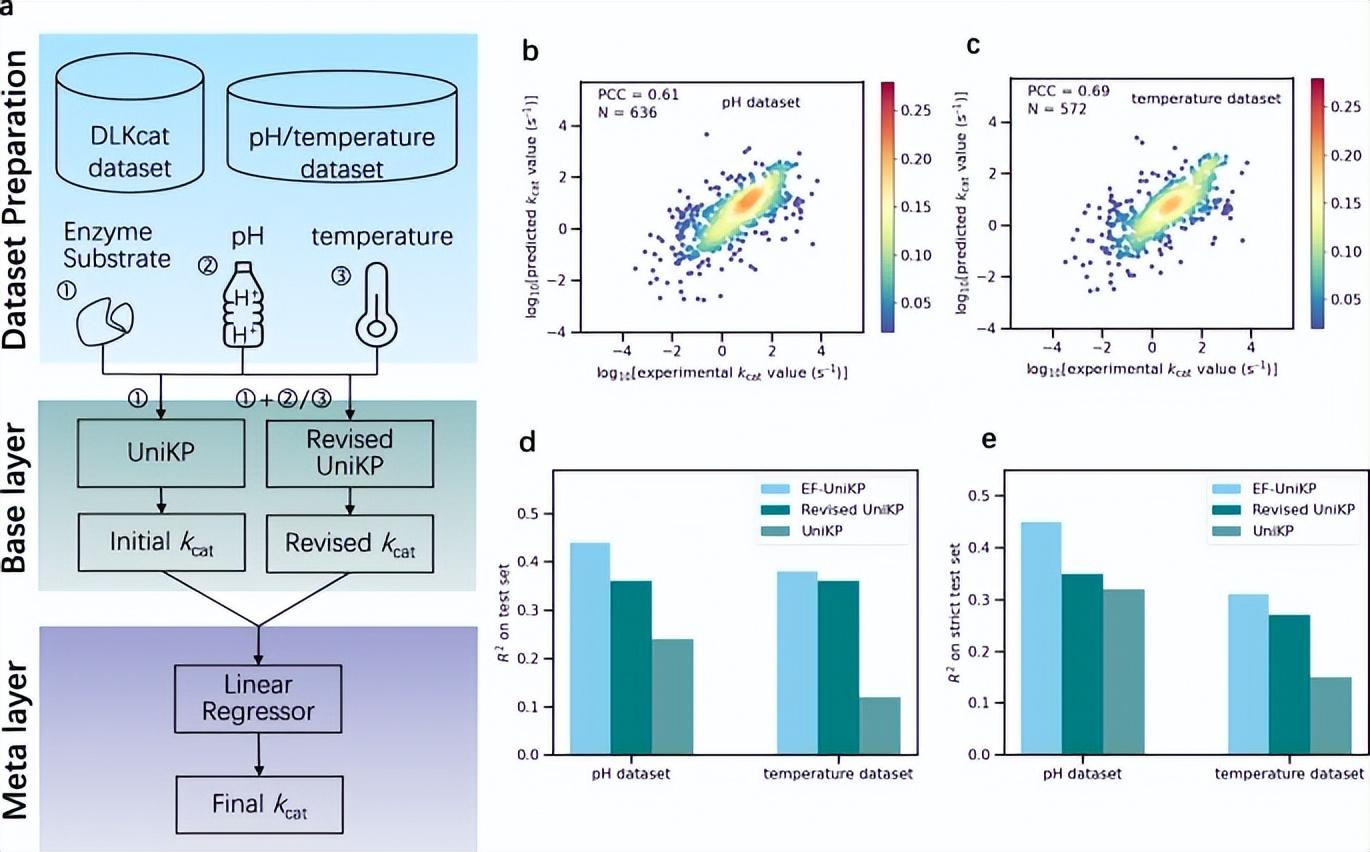
Illustration : Cadre à deux niveaux prenant en compte les facteurs environnementaux. (Source : Article)
Cependant, les modèles existants ne prennent pas en compte l'interaction entre ces facteurs en raison d'un manque de données complètes. À mesure que les techniques expérimentales progressent, notamment l’automatisation des laboratoires de biodiffusion et les méthodes d’évolution continue, les chercheurs anticipent une prolifération de données cinétiques enzymatiques. Cet afflux a non seulement enrichi le domaine mais a également amélioré la précision des modèles prédictifs.
En raison du déséquilibre élevé de l'ensemble de données kcat, qui entraîne des erreurs plus élevées dans les prédictions de valeurs kcat élevées, l'équipe a systématiquement exploré quatre méthodes de repondération représentatives pour atténuer ce problème. Les résultats montrent que les paramètres d’hyperparamètres de chaque méthode sont essentiels à l’amélioration des prédictions de valeurs kcat élevées.
L'équipe a confirmé la forte généralité du cadre actuel dans la prédiction de la constante de Michaelis (Km) et la prédiction kcat/Km. UniKP atteint des performances de pointe dans la prévision des valeurs Km et, de manière plus impressionnante, surpasse les résultats combinés des modèles de pointe actuels dans la prévision des valeurs kcat/Km. En outre, les chercheurs ont validé le cadre UniKP sur la base de valeurs kcat/Km mesurées expérimentalement et de valeurs kcat/Km calculées à l'aide de modèles de prédiction kcat et Km sur l'ensemble de données kcat/Km.
Il est à noter que la corrélation observée entre les valeurs dérivées d'UniKP kcat/UniKP Km et l'expérimental kcat/Km est relativement faible (PCC = −0,01). Cette différence peut être due aux différents ensembles de données utilisés dans la construction des modèles respectifs, nécessitant ainsi le développement d'un modèle différent pour prédire les valeurs kcat/Km. À l’avenir, avec l’émergence d’ensembles de données unifiés contenant les valeurs kcat et Km, il est prévu que les résultats informatiques des modèles kcat et Km soient étroitement cohérents avec les résultats générés par le modèle dédié kcat/Km.
Applications du béton dans l'extraction et l'évolution des enzymes
L'application d'UniKP dans l'extraction des enzymes tyrosine ammoniac lyase (TAL) et l'évolution dirigée démontre son potentiel pour révolutionner la recherche en biologie synthétique et en biochimie. Cette étude montre qu'UniKP reconnaît efficacement les TAL hautement actifs et améliore rapidement l'efficacité catalytique des TAL existants, le RgTAL-489T ayant une valeur kcat/Km 3,5 fois supérieure à celle de l'enzyme de type sauvage.
De plus, le cadre dérivé EF-UniKP a toujours été capable d'identifier les enzymes TAL hautement actives avec une précision extrêmement élevée, la valeur kcat/Km de TrTAL de Tephrocybe rancida étant 2,6 fois supérieure à celle de l'enzyme de type sauvage. Les résultats ont montré que les valeurs kcat et kcat/Km des cinq séquences dépassaient celles de l'enzyme de type sauvage.
En accélérant le processus de découverte et d'optimisation des enzymes, UniKP devrait devenir un outil puissant pour faire progresser la biocatalyse, la découverte de médicaments, l'ingénierie métabolique et d'autres domaines qui reposent sur des processus catalysés par des enzymes.
Limitations et perspectives
Cependant, la version actuelle d'UniKP présente encore certaines limitations. Par exemple, alors qu'UniKP est capable de faire la différence entre les valeurs kcat mesurées expérimentalement d'une enzyme et ses variantes, les valeurs kcat prédites ne sont pas suffisamment précises. Cela peut être dû à des ensembles de données insuffisants par rapport au nombre de séquences protéiques et de structures de substrat connues.
Bien que la méthode de repondération puisse atténuer dans une certaine mesure le biais de prédiction causé par l'ensemble de données kcat déséquilibré (amélioration d'environ 6,5 %), des améliorations plus significatives peuvent être obtenues grâce à des techniques de suréchantillonnage minoritaire synthétique et d'autres méthodes de synthèse d'échantillons.
Un objectif central de la biologie synthétique est le développement de cellules numériques qui révolutionneront la façon dont les scientifiques étudient la biologie. Une condition préalable essentielle à cette étude est la détermination minutieuse des paramètres enzymatiques pour toutes les enzymes de la voie. Les outils assistés par l’intelligence artificielle mettent en lumière ce défi, en fournissant une méthode à haut débit pour prédire la cinétique des enzymes.
Bien que l'erreur des prédicteurs UniKP soit réduite par rapport aux modèles précédents, l'imprécision reste un obstacle important à la construction de modèles métaboliques précis. L'intégration d'un nombre croissant de valeurs kcat et Km déterminées expérimentalement peut améliorer la précision du modèle.
Ensuite, les chercheurs ont l'intention de combiner des algorithmes de pointe tels que l'apprentissage par transfert, l'apprentissage par renforcement et d'autres algorithmes d'apprentissage à petite échelle pour gérer efficacement des ensembles de données déséquilibrés. Et l’équipe vise à explorer des applications supplémentaires, notamment l’évolution des enzymes et l’analyse globale des organismes.
Lien papier : https://www.nature.com/articles/s41467-023-44113-1
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!

Outils d'IA chauds

Undresser.AI Undress
Application basée sur l'IA pour créer des photos de nu réalistes

AI Clothes Remover
Outil d'IA en ligne pour supprimer les vêtements des photos.

Undress AI Tool
Images de déshabillage gratuites

Clothoff.io
Dissolvant de vêtements AI

AI Hentai Generator
Générez AI Hentai gratuitement.

Article chaud

Outils chauds

Bloc-notes++7.3.1
Éditeur de code facile à utiliser et gratuit

SublimeText3 version chinoise
Version chinoise, très simple à utiliser

Envoyer Studio 13.0.1
Puissant environnement de développement intégré PHP

Dreamweaver CS6
Outils de développement Web visuel

SublimeText3 version Mac
Logiciel d'édition de code au niveau de Dieu (SublimeText3)
 Repoussant les limites de la détection de défauts traditionnelle, « Defect Spectrum » permet pour la première fois une détection de défauts industriels d'une ultra haute précision et d'une sémantique riche.
Jul 26, 2024 pm 05:38 PM
Repoussant les limites de la détection de défauts traditionnelle, « Defect Spectrum » permet pour la première fois une détection de défauts industriels d'une ultra haute précision et d'une sémantique riche.
Jul 26, 2024 pm 05:38 PM
Dans la fabrication moderne, une détection précise des défauts est non seulement la clé pour garantir la qualité des produits, mais également la clé de l’amélioration de l’efficacité de la production. Cependant, les ensembles de données de détection de défauts existants manquent souvent de précision et de richesse sémantique requises pour les applications pratiques, ce qui rend les modèles incapables d'identifier des catégories ou des emplacements de défauts spécifiques. Afin de résoudre ce problème, une équipe de recherche de premier plan composée de l'Université des sciences et technologies de Hong Kong, Guangzhou et de Simou Technology a développé de manière innovante l'ensemble de données « DefectSpectrum », qui fournit une annotation à grande échelle détaillée et sémantiquement riche des défauts industriels. Comme le montre le tableau 1, par rapport à d'autres ensembles de données industrielles, l'ensemble de données « DefectSpectrum » fournit le plus grand nombre d'annotations de défauts (5 438 échantillons de défauts) et la classification de défauts la plus détaillée (125 catégories de défauts).
 Le modèle de dialogue NVIDIA ChatQA a évolué vers la version 2.0, avec la longueur du contexte mentionnée à 128 Ko
Jul 26, 2024 am 08:40 AM
Le modèle de dialogue NVIDIA ChatQA a évolué vers la version 2.0, avec la longueur du contexte mentionnée à 128 Ko
Jul 26, 2024 am 08:40 AM
La communauté ouverte LLM est une époque où une centaine de fleurs fleurissent et s'affrontent. Vous pouvez voir Llama-3-70B-Instruct, QWen2-72B-Instruct, Nemotron-4-340B-Instruct, Mixtral-8x22BInstruct-v0.1 et bien d'autres. excellents interprètes. Cependant, par rapport aux grands modèles propriétaires représentés par le GPT-4-Turbo, les modèles ouverts présentent encore des lacunes importantes dans de nombreux domaines. En plus des modèles généraux, certains modèles ouverts spécialisés dans des domaines clés ont été développés, tels que DeepSeek-Coder-V2 pour la programmation et les mathématiques, et InternVL pour les tâches de langage visuel.
 Google AI a remporté la médaille d'argent de l'Olympiade mathématique de l'OMI, le modèle de raisonnement mathématique AlphaProof a été lancé et l'apprentissage par renforcement est de retour.
Jul 26, 2024 pm 02:40 PM
Google AI a remporté la médaille d'argent de l'Olympiade mathématique de l'OMI, le modèle de raisonnement mathématique AlphaProof a été lancé et l'apprentissage par renforcement est de retour.
Jul 26, 2024 pm 02:40 PM
Pour l’IA, l’Olympiade mathématique n’est plus un problème. Jeudi, l'intelligence artificielle de Google DeepMind a réalisé un exploit : utiliser l'IA pour résoudre la vraie question de l'Olympiade mathématique internationale de cette année, l'OMI, et elle n'était qu'à un pas de remporter la médaille d'or. Le concours de l'OMI qui vient de se terminer la semaine dernière comportait six questions portant sur l'algèbre, la combinatoire, la géométrie et la théorie des nombres. Le système d'IA hybride proposé par Google a répondu correctement à quatre questions et a marqué 28 points, atteignant le niveau de la médaille d'argent. Plus tôt ce mois-ci, le professeur titulaire de l'UCLA, Terence Tao, venait de promouvoir l'Olympiade mathématique de l'IA (AIMO Progress Award) avec un prix d'un million de dollars. De manière inattendue, le niveau de résolution de problèmes d'IA s'était amélioré à ce niveau avant juillet. Posez les questions simultanément sur l'OMI. La chose la plus difficile à faire correctement est l'OMI, qui a la plus longue histoire, la plus grande échelle et la plus négative.
 Le point de vue de la nature : les tests de l'intelligence artificielle en médecine sont dans le chaos. Que faut-il faire ?
Aug 22, 2024 pm 04:37 PM
Le point de vue de la nature : les tests de l'intelligence artificielle en médecine sont dans le chaos. Que faut-il faire ?
Aug 22, 2024 pm 04:37 PM
Editeur | ScienceAI Sur la base de données cliniques limitées, des centaines d'algorithmes médicaux ont été approuvés. Les scientifiques se demandent qui devrait tester les outils et comment le faire au mieux. Devin Singh a vu un patient pédiatrique aux urgences subir un arrêt cardiaque alors qu'il attendait un traitement pendant une longue période, ce qui l'a incité à explorer l'application de l'IA pour réduire les temps d'attente. À l’aide des données de triage des salles d’urgence de SickKids, Singh et ses collègues ont construit une série de modèles d’IA pour fournir des diagnostics potentiels et recommander des tests. Une étude a montré que ces modèles peuvent accélérer les visites chez le médecin de 22,3 %, accélérant ainsi le traitement des résultats de près de 3 heures par patient nécessitant un examen médical. Cependant, le succès des algorithmes d’intelligence artificielle dans la recherche ne fait que le vérifier.
 Formation avec des millions de données cristallines pour résoudre le problème de la phase cristallographique, la méthode d'apprentissage profond PhAI est publiée dans Science
Aug 08, 2024 pm 09:22 PM
Formation avec des millions de données cristallines pour résoudre le problème de la phase cristallographique, la méthode d'apprentissage profond PhAI est publiée dans Science
Aug 08, 2024 pm 09:22 PM
Editeur | KX À ce jour, les détails structurels et la précision déterminés par cristallographie, des métaux simples aux grandes protéines membranaires, sont inégalés par aucune autre méthode. Cependant, le plus grand défi, appelé problème de phase, reste la récupération des informations de phase à partir d'amplitudes déterminées expérimentalement. Des chercheurs de l'Université de Copenhague au Danemark ont développé une méthode d'apprentissage en profondeur appelée PhAI pour résoudre les problèmes de phase cristalline. Un réseau neuronal d'apprentissage en profondeur formé à l'aide de millions de structures cristallines artificielles et de leurs données de diffraction synthétique correspondantes peut générer des cartes précises de densité électronique. L'étude montre que cette méthode de solution structurelle ab initio basée sur l'apprentissage profond peut résoudre le problème de phase avec une résolution de seulement 2 Angströms, ce qui équivaut à seulement 10 à 20 % des données disponibles à la résolution atomique, alors que le calcul ab initio traditionnel
 Afin de fournir un nouveau système de référence et d'évaluation de questions-réponses scientifiques et complexes pour les grands modèles, l'UNSW, Argonne, l'Université de Chicago et d'autres institutions ont lancé conjointement le cadre SciQAG.
Jul 25, 2024 am 06:42 AM
Afin de fournir un nouveau système de référence et d'évaluation de questions-réponses scientifiques et complexes pour les grands modèles, l'UNSW, Argonne, l'Université de Chicago et d'autres institutions ont lancé conjointement le cadre SciQAG.
Jul 25, 2024 am 06:42 AM
L'ensemble de données ScienceAI Question Answering (QA) joue un rôle essentiel dans la promotion de la recherche sur le traitement du langage naturel (NLP). Des ensembles de données d'assurance qualité de haute qualité peuvent non seulement être utilisés pour affiner les modèles, mais également évaluer efficacement les capacités des grands modèles linguistiques (LLM), en particulier la capacité à comprendre et à raisonner sur les connaissances scientifiques. Bien qu’il existe actuellement de nombreux ensembles de données scientifiques d’assurance qualité couvrant la médecine, la chimie, la biologie et d’autres domaines, ces ensembles de données présentent encore certaines lacunes. Premièrement, le formulaire de données est relativement simple, et la plupart sont des questions à choix multiples. Elles sont faciles à évaluer, mais limitent la plage de sélection des réponses du modèle et ne peuvent pas tester pleinement la capacité du modèle à répondre aux questions scientifiques. En revanche, les questions et réponses ouvertes
 Identifiez automatiquement les meilleures molécules et réduisez les coûts de synthèse. Le MIT développe un cadre d'algorithme de prise de décision en matière de conception moléculaire.
Jun 22, 2024 am 06:43 AM
Identifiez automatiquement les meilleures molécules et réduisez les coûts de synthèse. Le MIT développe un cadre d'algorithme de prise de décision en matière de conception moléculaire.
Jun 22, 2024 am 06:43 AM
Éditeur | L’utilisation de Ziluo AI pour rationaliser la découverte de médicaments explose. Ciblez des milliards de molécules candidates pour détecter celles qui pourraient posséder les propriétés nécessaires au développement de nouveaux médicaments. Il y a tellement de variables à prendre en compte, depuis le prix des matériaux jusqu’au risque d’erreur, qu’évaluer les coûts de synthèse des meilleures molécules candidates n’est pas une tâche facile, même si les scientifiques utilisent l’IA. Ici, les chercheurs du MIT ont développé SPARROW, un cadre d'algorithme de prise de décision quantitative, pour identifier automatiquement les meilleurs candidats moléculaires, minimisant ainsi les coûts de synthèse tout en maximisant la probabilité que les candidats possèdent les propriétés souhaitées. L’algorithme a également identifié les matériaux et les étapes expérimentales nécessaires à la synthèse de ces molécules. SPARROW prend en compte le coût de synthèse d'un lot de molécules à la fois, puisque plusieurs molécules candidates sont souvent disponibles
 Les performances de SOTA, la méthode d'IA de prédiction d'affinité protéine-ligand multimodale de Xiamen, combinent pour la première fois des informations sur la surface moléculaire
Jul 17, 2024 pm 06:37 PM
Les performances de SOTA, la méthode d'IA de prédiction d'affinité protéine-ligand multimodale de Xiamen, combinent pour la première fois des informations sur la surface moléculaire
Jul 17, 2024 pm 06:37 PM
Editeur | KX Dans le domaine de la recherche et du développement de médicaments, il est crucial de prédire avec précision et efficacité l'affinité de liaison des protéines et des ligands pour le criblage et l'optimisation des médicaments. Cependant, les études actuelles ne prennent pas en compte le rôle important des informations sur la surface moléculaire dans les interactions protéine-ligand. Sur cette base, des chercheurs de l'Université de Xiamen ont proposé un nouveau cadre d'extraction de caractéristiques multimodales (MFE), qui combine pour la première fois des informations sur la surface des protéines, la structure et la séquence 3D, et utilise un mécanisme d'attention croisée pour comparer différentes modalités. alignement. Les résultats expérimentaux démontrent que cette méthode atteint des performances de pointe dans la prédiction des affinités de liaison protéine-ligand. De plus, les études d’ablation démontrent l’efficacité et la nécessité des informations sur la surface des protéines et de l’alignement des caractéristiques multimodales dans ce cadre. Les recherches connexes commencent par "S





