

清華大学AIR、北京大学、南京大学の研究チームがESM-AAモデルを提案しました。このモデルはタンパク質言語モデリングの分野で重要な進歩を遂げ、マルチスケール情報を統合する統合モデリング ソリューションを提供します。
アミノ酸情報と原子情報の両方を扱える初めてのタンパク質事前学習済み言語モデルです。モデルの優れたパフォーマンスは、既存の制限を克服し、新しい機能を解放するマルチスケール統合モデリングの大きな可能性を示しています。
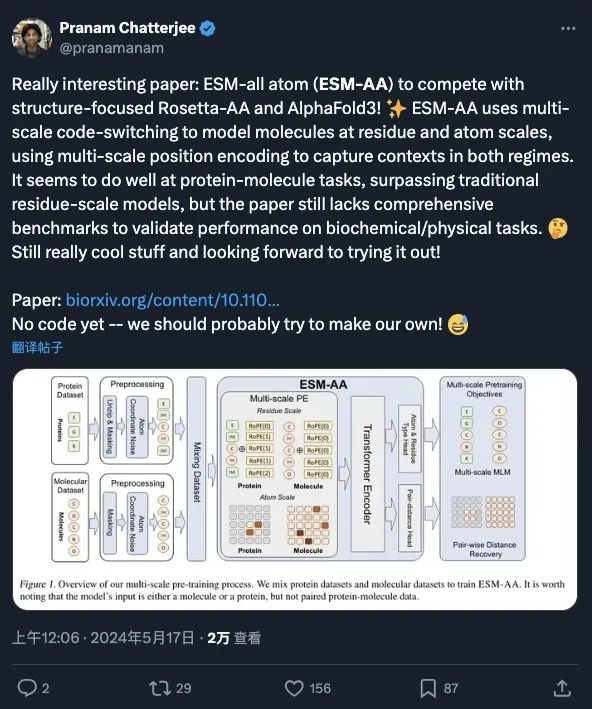
基本モデルとして、ESM-AA は多くの学者から注目と広範な議論を受けており (以下のスクリーンショットを参照)、AlphaFold3 や RoseTTAFold All-Atom と競合できる ESM-AA に基づくモデルを開発する可能性があると考えられています。それは、異なる生物学的構造間の相互作用を研究するための新しい道を開きます。現在の論文は ICML 2024 に受理されました。
タンパク質は、さまざまな生命活動の重要な実行者です。タンパク質およびタンパク質と他の生物学的構造との相互作用を深く理解することは生物科学の中核課題であり、これは標的薬物スクリーニング、酵素工学およびその他の分野にとって実用的に重要な意味を持っています。
そのため、タンパク質をよりよく理解し、モデル化する方法が、AI4Science の分野で研究のホットスポットとなっています。
ここ数日、Deepmind やワシントン大学の Baker Group などの主要な最先端研究機関も、タンパク質の全原子モデリングの問題について詳細な研究を実施し、AlphaFold 3、RoseTTAFold などの手法を提案しています。タンパク質やその他の生命活動のAll-Atomなど、関連分子の全原子スケールモデリングモデルにより、タンパク質の構造、分子構造、受容体-リガンド構造などの全原子スケールを高精度に予測することができます。
これらのモデルは全原子スケールでの構造モデリングにおいて大きな進歩を遂げましたが、現在の主流のタンパク質言語モデルは依然として全原子スケールでのタンパク質の理解と表現学習を達成することができません。
ESM-2に代表されるタンパク質表現学習モデルは、モデルを構築するための唯一のスケールとしてアミノ酸を使用しており、以下を重視する状況に適しています。タンパク質を処理することは合理的なアプローチです。
しかし、タンパク質の性質を完全に理解する鍵は、小分子、DNA、RNA などの他の生物学的構造との相互作用を解明することにあります。
この需要に直面して、異なる構造間の複雑な相互作用を記述する必要があり、単一スケールのモデリング戦略では効果的で包括的なカバレッジを提供することは困難です。
この欠点を克服するために、タンパク質モデルはマルチスケール モデルに向けて大幅な革新を受けています。たとえば、5 月初旬に Science 誌に掲載された RoseTTAFold All-Atom モデルは、RoseTTAFold の後継製品として、マルチスケールの概念を導入しました。
このモデルはタンパク質の構造予測に限定されるものではなく、タンパク質と分子・核酸のドッキング、タンパク質の翻訳後修飾など、より広い研究分野にも拡張できます。
同時に、DeepMind が新しくリリースした AlphaFold3 も、複数のタンパク質複合体の構造の予測をサポートするマルチスケール モデリング戦略を採用しており、そのパフォーマンスは素晴らしく、間違いなく人工知能と生物学の分野に大きな影響を与えるでしょう。 。
RoseTTAFold All-Atom と AlphaFold3 のマルチスケールへの適用の成功重要な考え方は、タンパク質ベースモデルとしてのタンパク質言語モデルがマルチスケールテクノロジーをどのように採用すべきかということです。これに基づいて、チームはマルチスケールタンパク質言語モデル ESM All-Atom (ESM-AA) を提案しました。
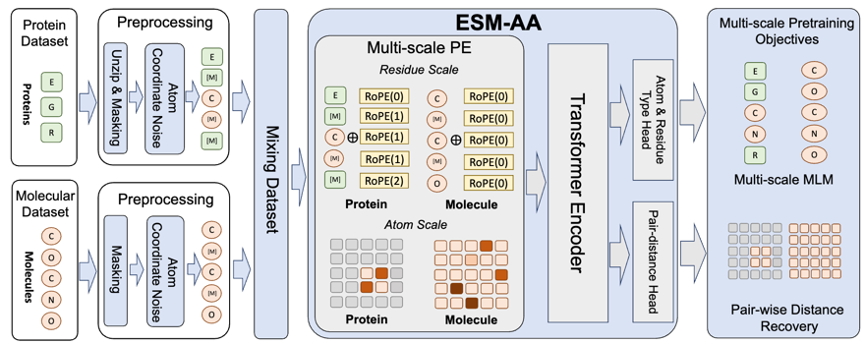
簡単に説明すると、ESM-AA は、一部のアミノ酸を対応する原子組成に「解凍」することにより、マルチスケールの概念を導入します。続いて、タンパク質データと分子データを混合して事前学習を行うことで、モデルは異なるスケールの生物学的構造を同時に扱えるようになりました。
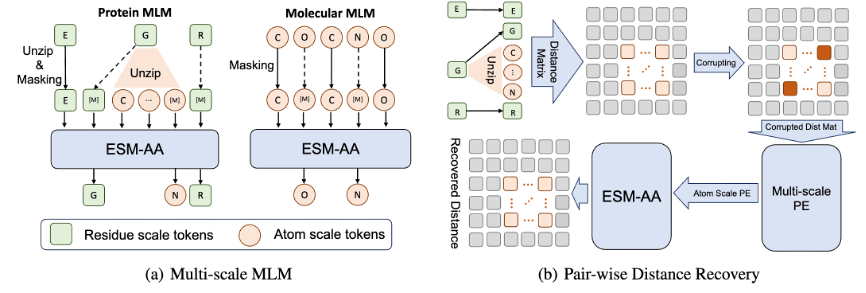
さらに、モデルが高品質の原子スケールの情報をよりよく学習できるようにするために、ESM-AA はトレーニングに原子スケールの分子構造データも使用します。さらに、図 2 に示すマルチスケール位置エンコード機構を導入することにより、ESM-AA モデルはさまざまなスケールで情報を適切に区別できるようになり、モデルが残基レベルおよび原子レベルで位置および構造情報を正確に理解できるようになります。
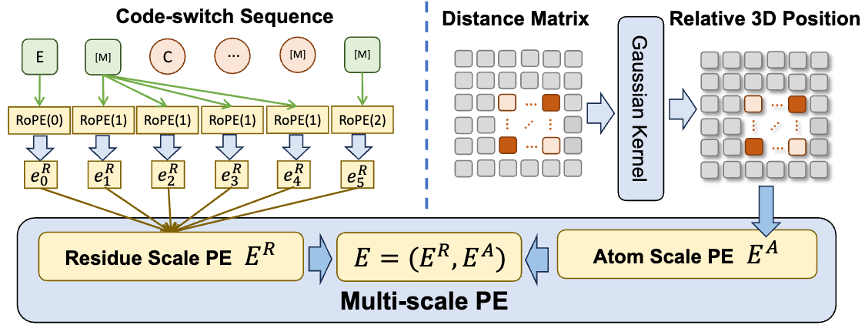
モデルがマルチスケールの情報を学習できるようにするために、チームは ESM-AA モデルのさまざまな事前トレーニング目標を設計しました。 ESM-AA のマルチスケール事前トレーニング目標には、マスク言語モデリング (MLM) とペアワイズ距離回復 (PDR) が含まれます。図 3(a) に示すように、MLM では、アミノ酸と原子をマスクすることで周囲のコンテキストに基づいて予測を行うモデルが必要です。このトレーニング タスクは、アミノ酸スケールと原子スケールの両方で実行できます。 PDR では、モデルが原子レベルの構造情報を理解できるようにトレーニングするために、異なる原子間のユークリッド距離を正確に予測する必要があります (図 3(b) を参照)。
ESM-AA モデルは、酵素を含む複数のタンパク質-低分子ベンチマーク タスクで微調整され、評価されます-基質物質親和性回帰タスク (結果を図 4 に示す)、酵素-基質ペア分類タスク (結果を図 4 に示す)、および薬物-標的親和性回帰タスク (結果を図 5 に示す)。
結果は、ESM-AA がこれらのタスクにおいて以前のモデルよりも優れていることを示しており、アミノ酸および原子スケールでタンパク質の事前トレーニング済み言語モデルの可能性を完全に実現していることを示しています。
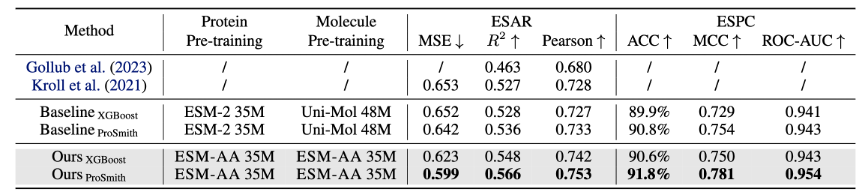
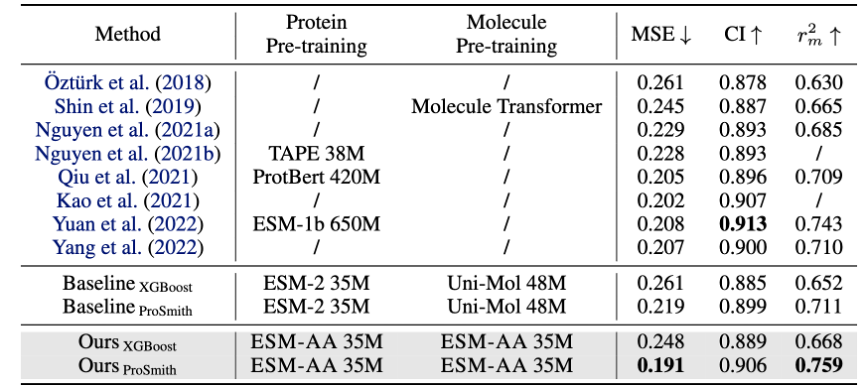
図 5: 薬物-ターゲット親和性回帰タスクのパフォーマンス比較
さらに、ESM-AAモデルもタンパク質接触予測、タンパク質機能分類、分子特性予測などのタスクでパフォーマンスをテストしました。
その結果は、タンパク質のみを含むタスクを扱う場合、ESM-AA は ESM-2 と同等のパフォーマンスを発揮し、分子タスクでは ESM-AA モデルがほとんどのベンチマーク モデルを上回り、Uni-Mol と同様であることを示しています。
これは、ESM-AA が強力な分子知識を獲得する過程でタンパク質を理解する能力を犠牲にしていないことを示しています。また、ESM-AA モデルが最初から始める必要なく、ESM-2 モデルの知識をうまく再利用していることも示しています。スクラッチ開発により、モデルのトレーニング コストが大幅に削減されます。
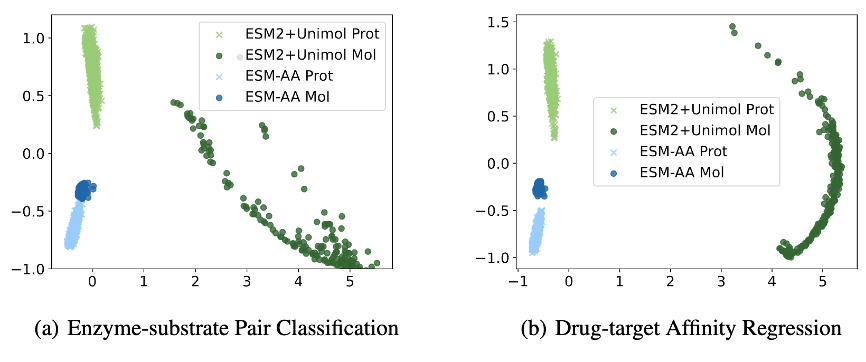
ESM-AA がタンパク質-低分子ベンチマーク タスクで優れたパフォーマンスを発揮する理由をさらに分析するために、この論文では ESM-AA モデルと ESM-2+Uni-Mol の抽出を示します。このタスクでのモデルの組み合わせ サンプル表現分布の視覚化。
図 6 に示すように、ESM-AA モデルによって学習されたタンパク質と小分子の表現はよりコンパクトです。これは、この 2 つが同じ表現空間内にあることを示しています。これが、ESM-AA モデルが ESM よりも優れている理由です。 -2+Uni-Mol モデル。マルチスケールの統一分子モデリングの利点をさらに示しています。
結論
清華 AIR チームによって開発された ESM-AA は、アミノ酸と原子の情報処理を統合した初のタンパク質事前トレーニング済み言語モデルです。このモデルは、マルチスケールの情報を統合することによって堅牢で優れたパフォーマンスを実証し、生物学的構造間の相互作用の問題を解決する新しい方法を提供します。
ESM-AA は、タンパク質のより深い理解を促進するだけでなく、複数の生体分子のタスクでも優れたパフォーマンスを発揮し、タンパク質の理解能力を維持しながら分子レベルの知識を効果的に統合できることを証明し、モデルのトレーニングのコストを削減します。 AI支援による生物学研究の新たな方向性。
論文タイトル: ESM All-Atom: 統一分子モデリングのためのマルチスケールタンパク質言語モデル
以上が清華航空などは、アミノ酸から原子スケールまでの初のタンパク質言語モデルである ESM-AA を提案しましたの詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。