Nature サブジャーナル、精度 96% の AI が配列からタンパク質とリガンドの相互作用を予測
医薬品開発では、タンパク質に対する小分子リガンドの結合親和性と機能的影響を決定することが重要です。現在の計算手法では、これらのタンパク質とリガンドの相互作用特性を予測できますが、高解像度のタンパク質構造がなければ、多くの場合、精度が失われ、機能的効果を予測できません。
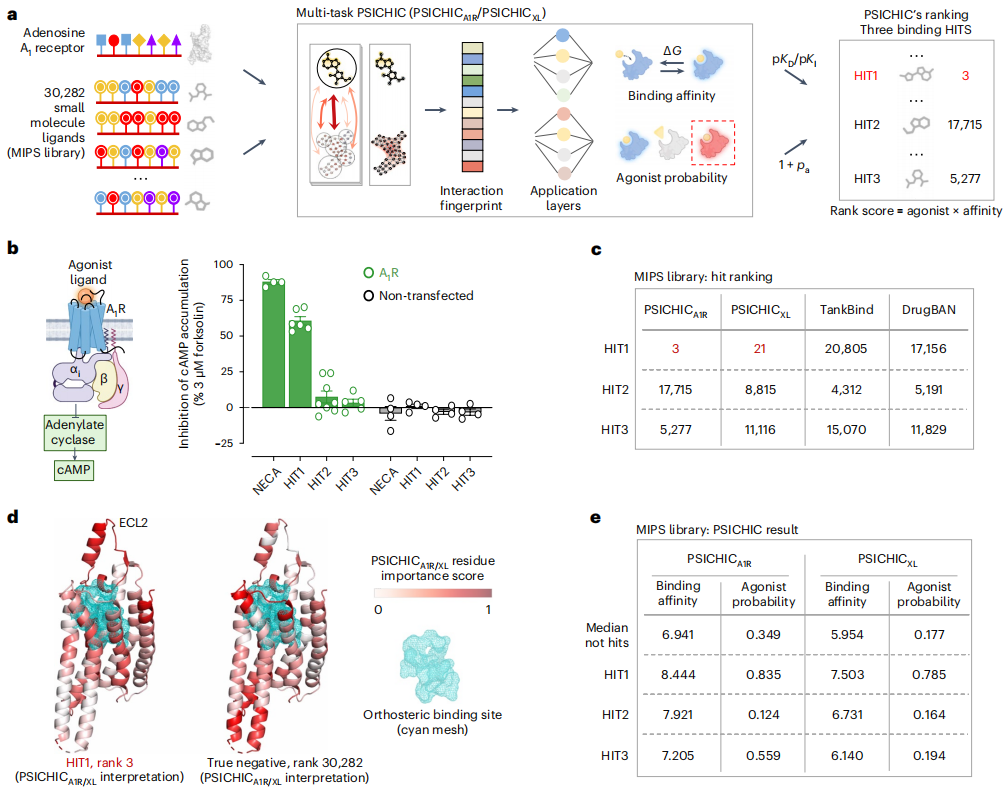
モナッシュ大学とグリフィス大学の研究者は、相互作用フィンガープリントをデコードする配列データから物理化学的制約を直接結合するフレームワークであるPSICHIC (PhySIcoCHhemICal グラフ ニューラル ネットワーク) を開発しました。これにより、PSICHIC はタンパク質とリガンドの相互作用の背後にあるメカニズムを解読し、最先端の精度と解釈可能性を達成することができます。
構造データを使用せずに同じタンパク質-リガンドのペアでトレーニングしたPSICHICは、結合親和性予測において主要な構造ベースの方法と同等、またはそれを上回りました。
PSICHIC の解釈可能なフィンガープリントは、相互作用に関与するタンパク質残基とリガンド原子を特定し、タンパク質-リガンド相互作用の選択性決定要因を明らかにするのに役立ちます。
この研究は「シーケンスデータからタンパク質-リガンド相互作用フィンガープリントを学習するための物理化学グラフニューラルネットワーク」と題され、2024年6月17日に「Nature Machine Intelligence」に掲載されました。

創薬プロセスでは、特定のタンパク質とリガンドの選択的相互作用が決定するため、タンパク質に対する小分子リガンドの結合親和性と機能的影響を理解することが重要です。期待される薬の効果。
しかし、現在の計算手法ではタンパク質とリガンドの相互作用特性を予測することは可能ですが、高解像度のタンパク質構造がないと予測精度が低下することが多く、機能効果の予測も困難です。
シーケンスベースの方法は、コストとリソースの点でより多くの利点がありますが(たとえば、高価な実験による構造決定プロセスを必要としない)、これらの方法は、パターンマッチングにおける過剰な自由度の問題に直面することが多く、簡単に過剰適合につながる可能性があります。一般化機能が限られているため、構造ベースまたは複合ベースの方法と比較してパフォーマンスのギャップが生じます。
物理化学グラフ ニューラル ネットワーク
モナッシュ大学とグリフィス大学の研究チームは、物理的および化学的な身体相互作用に従って配列データからタンパク質-リガンドを直接解読する方法である PSICHIC (物理化学グラフ ニューラル ネットワーク) を開発しました。指紋認証方式。以前の配列ベースのモデルとは異なり、PSICHIC は物理化学的制約を独自に組み込んで、最先端の精度と解釈可能性を実現します。
2D シーケンスベースのメソッドとして、PSICHIC はクラスタリング アルゴリズムを適用することでこれらの制約を生成し、2D プロットに課します。これにより、PSICHIC はトレーニング中にタンパク質とリガンドの相互作用を決定する合理的な基礎パターンに主に適応できます。

(出典: 論文)
パフォーマンスの検証と比較
構造データなしで同じタンパク質-リガンドペアでトレーニングした後、PSICHIC は結合親和性予測で優れたパフォーマンスを示します- アート構造ベースの手法と複合ベースの手法は、それらに匹敵するか、さらにはそれを上回ります。
PDBBind v2016 および PDBBind v2020 データセットの実験結果は、PSICHIC が複数の指標において TransCPI、MolTrans、DrugBAN などの他の配列ベースの手法よりも優れていることを示しています。
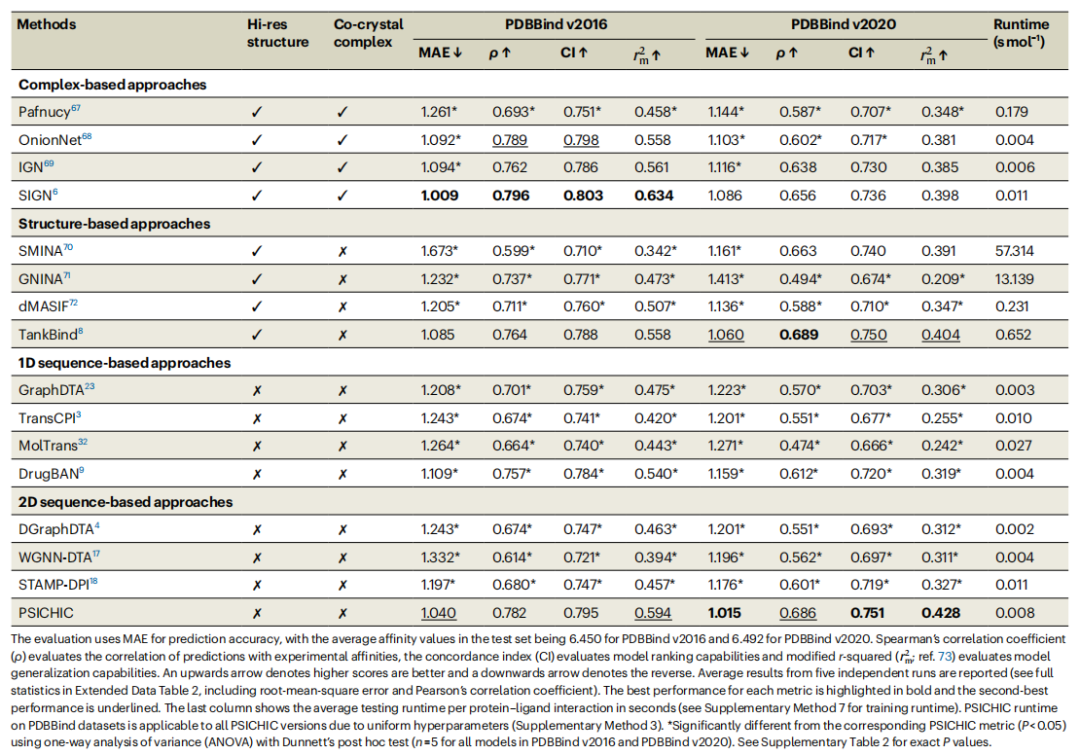
PDBBind v2016 および PDBBind v2020 ベンチマークでのタンパク質-リガンド結合親和性予測のパフォーマンス統計概要。 (出典: 論文)
具体的には:
- PSICHIC は、特に予測精度と一般化能力の点で、予測誤差が低く、相関指数が高いことが示されています。
- PSICHIC は、機能効果予測において最大 96% の精度を達成します。
また:
- PSICHIC は、結合部位と主要なリガンド官能基の同定に優れています。
- 複数のタンパク質-リガンド複合体構造(PDB 6K1S や 6OXV など)の解析において、PSICHIC は重要な結合残基とリガンド官能基を正確に特定することができ、配列データ内のタンパク質-リガンドを直接解読する能力を検証しました。身体の相互作用パターン。
- この能力は、配列データからタンパク質とリガンドの結合部位や重要な残基を予測する能力に特に反映されています。
イラスト: インタラクティブな指紋を使用した仮想スクリーニング。 (出典: 論文)
興味深いことに、PSICHIC の解釈可能なフィンガープリントは、PSICHIC が配列データのみからタンパク質-リガンド相互作用の根底にあるメカニズムを解読し、結合部位タンパク質残基と関与するリガンドの原子能力を特定する能力を獲得していることを示しています。結合親和性ラベルを持ち、相互作用情報を持たない配列データ。
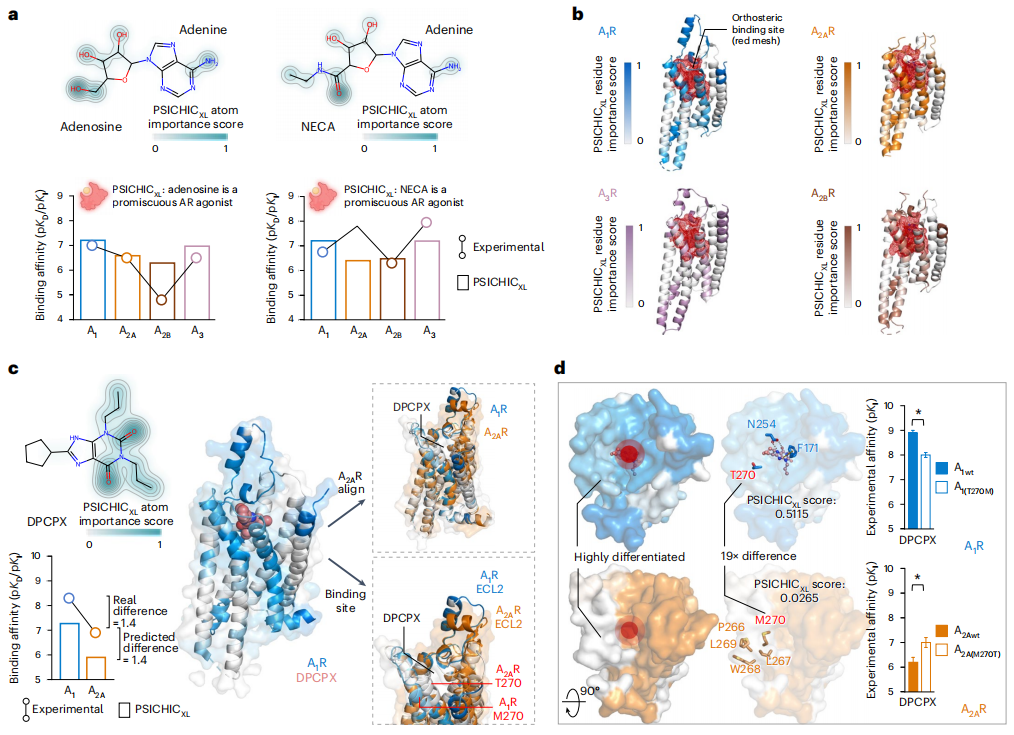
- タンパク質-リガンド相互作用フィンガープリントは、リガンドとタンパク質残基間の特定の相互作用特性を記述します。
- PSICHIC は、シーケンス データのみを使用して解釈可能なインタラクション フィンガープリントを取得するための独自のアプローチを提供します。
- PSICHIC は制約を組み込んで、タンパク質-リガンド相互作用メカニズムを明らかにし、相互作用特性を効率的に予測するための新たな機能を実証します。
- PSICHIC は 3D データの必要性を排除し、大規模な配列データベースでの堅牢な学習への道を開きます。
将来の展望
- PSICHIC 分析をヘテロ三量体 G タンパク質と複合体を形成した GPCR などのタンパク質複合体に拡張します。
- アロステリック制御などの複雑な相互作用を調べることは、アロステリック リガンドがタンパク質標的内のオルソステリック リガンドをどのように制御するかを理解するのに役立ちます。
- PSICHIC は、さまざまなアプリケーション分野でその堅牢性と有効性が証明されており、将来の開発の幅広い可能性を秘めています。
以上がNature サブジャーナル、精度 96% の AI が配列からタンパク質とリガンドの相互作用を予測の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。

ホットAIツール

Undresser.AI Undress
リアルなヌード写真を作成する AI 搭載アプリ

AI Clothes Remover
写真から衣服を削除するオンライン AI ツール。

Undress AI Tool
脱衣画像を無料で

Clothoff.io
AI衣類リムーバー

Video Face Swap
完全無料の AI 顔交換ツールを使用して、あらゆるビデオの顔を簡単に交換できます。

人気の記事

ホットツール

メモ帳++7.3.1
使いやすく無料のコードエディター

SublimeText3 中国語版
中国語版、とても使いやすい

ゼンドスタジオ 13.0.1
強力な PHP 統合開発環境

ドリームウィーバー CS6
ビジュアル Web 開発ツール

SublimeText3 Mac版
神レベルのコード編集ソフト(SublimeText3)

ホットトピック
 1667
1667
 14
14
 1426
1426
 52
52
 1328
1328
 25
25
 1273
1273
 29
29
 1255
1255
 24
24
 DeepMind ロボットが卓球をすると、フォアハンドとバックハンドが空中に滑り出し、人間の初心者を完全に打ち負かしました
Aug 09, 2024 pm 04:01 PM
DeepMind ロボットが卓球をすると、フォアハンドとバックハンドが空中に滑り出し、人間の初心者を完全に打ち負かしました
Aug 09, 2024 pm 04:01 PM
でももしかしたら公園の老人には勝てないかもしれない?パリオリンピックの真っ最中で、卓球が注目を集めています。同時に、ロボットは卓球のプレーにも新たな進歩をもたらしました。先ほど、DeepMind は、卓球競技において人間のアマチュア選手のレベルに到達できる初の学習ロボット エージェントを提案しました。論文のアドレス: https://arxiv.org/pdf/2408.03906 DeepMind ロボットは卓球でどれくらい優れていますか?おそらく人間のアマチュアプレーヤーと同等です: フォアハンドとバックハンドの両方: 相手はさまざまなプレースタイルを使用しますが、ロボットもそれに耐えることができます: さまざまなスピンでサーブを受ける: ただし、ゲームの激しさはそれほど激しくないようです公園の老人。ロボット、卓球用
 初のメカニカルクロー!元羅宝は2024年の世界ロボット会議に登場し、家庭に入ることができる初のチェスロボットを発表した
Aug 21, 2024 pm 07:33 PM
初のメカニカルクロー!元羅宝は2024年の世界ロボット会議に登場し、家庭に入ることができる初のチェスロボットを発表した
Aug 21, 2024 pm 07:33 PM
8月21日、2024年世界ロボット会議が北京で盛大に開催された。 SenseTimeのホームロボットブランド「Yuanluobot SenseRobot」は、全製品ファミリーを発表し、最近、世界初の家庭用チェスロボットとなるYuanluobot AIチェスプレイロボット - Chess Professional Edition(以下、「Yuanluobot SenseRobot」という)をリリースした。家。 Yuanluobo の 3 番目のチェス対局ロボット製品である新しい Guxiang ロボットは、AI およびエンジニアリング機械において多くの特別な技術アップグレードと革新を経て、初めて 3 次元のチェスの駒を拾う機能を実現しました。家庭用ロボットの機械的な爪を通して、チェスの対局、全員でのチェスの対局、記譜のレビューなどの人間と機械の機能を実行します。
 クロードも怠け者になってしまった!ネチズン: 自分に休日を与える方法を学びましょう
Sep 02, 2024 pm 01:56 PM
クロードも怠け者になってしまった!ネチズン: 自分に休日を与える方法を学びましょう
Sep 02, 2024 pm 01:56 PM
もうすぐ学校が始まり、新学期を迎える生徒だけでなく、大型AIモデルも気を付けなければなりません。少し前、レディットはクロードが怠け者になったと不満を漏らすネチズンでいっぱいだった。 「レベルが大幅に低下し、頻繁に停止し、出力も非常に短くなりました。リリースの最初の週は、4 ページの文書全体を一度に翻訳できましたが、今では 0.5 ページの出力さえできません」 !」 https://www.reddit.com/r/ClaudeAI/comments/1by8rw8/something_just_feels_wrong_with_claude_in_the/ というタイトルの投稿で、「クロードには完全に失望しました」という内容でいっぱいだった。
 世界ロボット会議で「未来の高齢者介護の希望」を担う家庭用ロボットを囲みました
Aug 22, 2024 pm 10:35 PM
世界ロボット会議で「未来の高齢者介護の希望」を担う家庭用ロボットを囲みました
Aug 22, 2024 pm 10:35 PM
北京で開催中の世界ロボット会議では、人型ロボットの展示が絶対的な注目となっているスターダストインテリジェントのブースでは、AIロボットアシスタントS1がダルシマー、武道、書道の3大パフォーマンスを披露した。文武両道を備えた 1 つの展示エリアには、多くの専門的な聴衆とメディアが集まりました。弾性ストリングのエレガントな演奏により、S1 は、スピード、強さ、正確さを備えた繊細な操作と絶対的なコントロールを発揮します。 CCTVニュースは、「書道」の背後にある模倣学習とインテリジェント制御に関する特別レポートを実施し、同社の創設者ライ・ジエ氏は、滑らかな動きの背後にあるハードウェア側が最高の力制御と最も人間らしい身体指標(速度、負荷)を追求していると説明した。など)、AI側では人の実際の動きのデータが収集され、強い状況に遭遇したときにロボットがより強くなり、急速に進化することを学習することができます。そしてアジャイル
 ACL 2024 賞の発表: HuaTech による Oracle 解読に関する最優秀論文の 1 つ、GloVe Time Test Award
Aug 15, 2024 pm 04:37 PM
ACL 2024 賞の発表: HuaTech による Oracle 解読に関する最優秀論文の 1 つ、GloVe Time Test Award
Aug 15, 2024 pm 04:37 PM
貢献者はこの ACL カンファレンスから多くのことを学びました。 6日間のACL2024がタイのバンコクで開催されています。 ACL は、計算言語学と自然言語処理の分野におけるトップの国際会議で、国際計算言語学協会が主催し、毎年開催されます。 ACL は NLP 分野における学術的影響力において常に第一位にランクされており、CCF-A 推奨会議でもあります。今年の ACL カンファレンスは 62 回目であり、NLP 分野における 400 以上の最先端の作品が寄せられました。昨日の午後、カンファレンスは最優秀論文およびその他の賞を発表しました。今回の優秀論文賞は7件(未発表2件)、最優秀テーマ論文賞1件、優秀論文賞35件です。このカンファレンスでは、3 つの Resource Paper Award (ResourceAward) と Social Impact Award (
 Li Feifei 氏のチームは、ロボットに空間知能を与え、GPT-4o を統合する ReKep を提案しました
Sep 03, 2024 pm 05:18 PM
Li Feifei 氏のチームは、ロボットに空間知能を与え、GPT-4o を統合する ReKep を提案しました
Sep 03, 2024 pm 05:18 PM
ビジョンとロボット学習の緊密な統合。最近話題の1X人型ロボットNEOと合わせて、2つのロボットハンドがスムーズに連携して服をたたむ、お茶を入れる、靴を詰めるといった動作をしていると、いよいよロボットの時代が到来するのではないかと感じられるかもしれません。実際、これらの滑らかな動きは、高度なロボット技術 + 精緻なフレーム設計 + マルチモーダル大型モデルの成果です。有用なロボットは多くの場合、環境との複雑かつ絶妙な相互作用を必要とし、環境は空間領域および時間領域の制約として表現できることがわかっています。たとえば、ロボットにお茶を注いでもらいたい場合、ロボットはまずティーポットのハンドルを掴んで、お茶をこぼさないように垂直に保ち、次にポットの口がカップの口と揃うまでスムーズに動かす必要があります。 、そしてティーポットを一定の角度に傾けます。これ
 分散型人工知能カンファレンス DAI 2024 論文募集: エージェント デイ、強化学習の父であるリチャード サットン氏が出席します。 Yan Shuicheng、Sergey Levine、DeepMind の科学者が基調講演を行います
Aug 22, 2024 pm 08:02 PM
分散型人工知能カンファレンス DAI 2024 論文募集: エージェント デイ、強化学習の父であるリチャード サットン氏が出席します。 Yan Shuicheng、Sergey Levine、DeepMind の科学者が基調講演を行います
Aug 22, 2024 pm 08:02 PM
会議の紹介 科学技術の急速な発展に伴い、人工知能は社会の進歩を促進する重要な力となっています。この時代に、分散型人工知能 (DAI) の革新と応用を目撃し、参加できることは幸運です。分散型人工知能は人工知能分野の重要な分野であり、近年ますます注目を集めています。大規模言語モデル (LLM) に基づくエージェントは、大規模モデルの強力な言語理解機能と生成機能を組み合わせることで、自然言語対話、知識推論、タスク計画などにおいて大きな可能性を示しました。 AIAgent は大きな言語モデルを引き継ぎ、現在の AI 界隈で話題になっています。アウ
 宏蒙スマートトラベルS9とフルシナリオ新製品発売カンファレンス、多数の大ヒット新製品が一緒にリリースされました
Aug 08, 2024 am 07:02 AM
宏蒙スマートトラベルS9とフルシナリオ新製品発売カンファレンス、多数の大ヒット新製品が一緒にリリースされました
Aug 08, 2024 am 07:02 AM
今日の午後、Hongmeng Zhixingは新しいブランドと新車を正式に歓迎しました。 8月6日、ファーウェイはHongmeng Smart Xingxing S9およびファーウェイのフルシナリオ新製品発表カンファレンスを開催し、パノラマスマートフラッグシップセダンXiangjie S9、新しいM7ProおよびHuawei novaFlip、MatePad Pro 12.2インチ、新しいMatePad Air、Huawei Bisheng Withを発表しました。レーザー プリンタ X1 シリーズ、FreeBuds6i、WATCHFIT3、スマート スクリーン S5Pro など、スマート トラベル、スマート オフィスからスマート ウェアに至るまで、多くの新しいオールシナリオ スマート製品を開発し、ファーウェイは消費者にスマートな体験を提供するフル シナリオのスマート エコシステムを構築し続けています。すべてのインターネット。宏孟志興氏:スマートカー業界のアップグレードを促進するための徹底的な権限付与 ファーウェイは中国の自動車業界パートナーと提携して、