

編集者|サイエンスAI
最近、研究所中国科学院コンピューティング技術部門の張海滄氏率いる研究チームは、CarbonNovo に対し、エンドツーエンド方式でタンパク質の主鎖構造と配列を共同設計することを提案した。
この研究は、機械学習カンファレンスICML 2024で「CarbonNovo: 統合エネルギーベースモデルを使用したタンパク質の構造と配列の共同設計」というタイトルで発表されました。

背景紹介
タンパク質は生物学的機能にとって重要な高分子です。 De novo タンパク質設計は、まったく新しいタンパク質を作成することを目的としており、医薬品開発や酵素工学に幅広く応用できます。
近年、AIベースのタンパク質デノボデザインは急速に発展しており、従来の設計手法と比較して、抗体設計や低分子タンパク質医薬品の設計などの分野で成功裏に使用されています。成功率と効率。
AI タンパク質の設計は、近年の 2 つの大きな技術的進歩の恩恵を受けています:
1 つ目は、タンパク質構造予測の分野における AlphaFold2 モデルであり、これは、タンパク質設計を含むタンパク質コンピューティングの分野。基本的なニューラル ネットワーク モデル アーキテクチャ、タンパク質配列表現および構造表現方法、高度なトレーニング戦略 (蒸留トレーニング、エンドツーエンド トレーニング) およびその他のテクノロジーを提供します。 #
2 つ目は、テキスト、画像の AIGC です。ビデオ生成分野の急速な発展により、DDPM、SDE、フロー マッチング、ベイジアン フロー ネットワークなど、タンパク質設計のための成熟した生成モデルが提供されています。 RFDiffusion や Chroma などの代表的なタンパク質設計モデルの主なアイデアは、これら 2 つの技術を統合し、タンパク質の配列と構造表現ネットワークを AI ベースの生成モデル フレームワークに組み込むことです。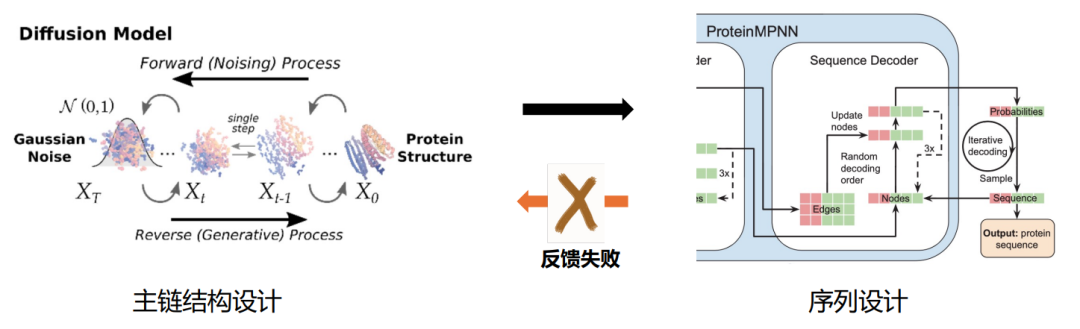
CarbonNovo はエンドツーエンドの構造とシーケンスの結合設計を実行します
「2 段階」フレームワークの制限に対応して、タンパク質設計、中国科学院計算技術研究所のZhang Haicang氏 同氏率いる研究チームは、エンドツーエンド方式でタンパク質の主鎖構造と配列を共同設計することをCarbonNovoに提案した。この論文は最近、機械学習カンファレンス ICML 2024 で発表されました。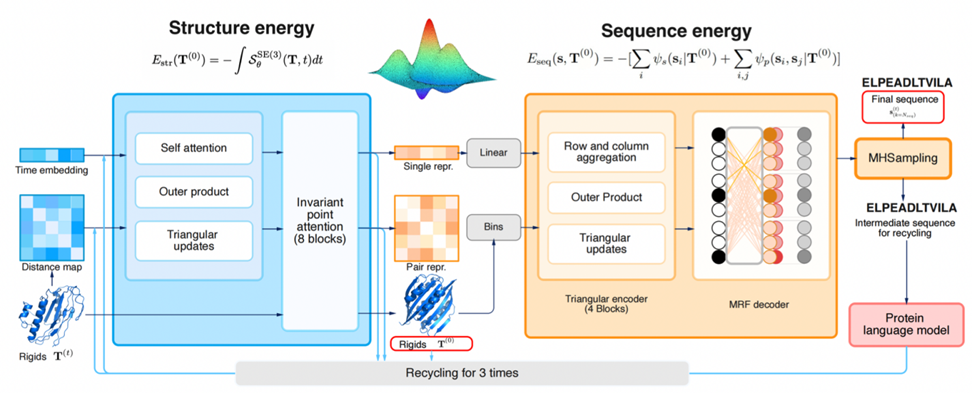
タンパク質構造-配列の結合エネルギーモデル
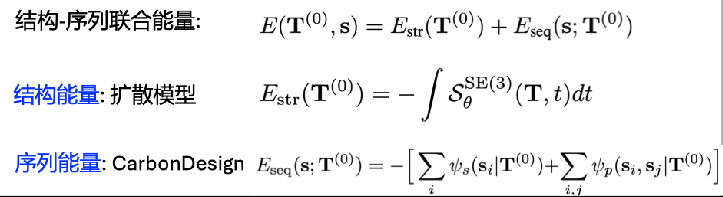
Dans le modèle physique classique, la conformation naturelle des protéines a une énergie libre relativement faible, ce qui constitue également une hypothèse générale pour la prédiction et la conception de la structure des protéines. Sur cette base, CarbonNovo a établi un modèle énergétique conjoint de la structure et de la séquence des protéines :
La figure 2 montre le processus de génération spécifique de CarbonNovo :
Évaluation des performances de CarbonNovo pour générer une structure-séquence de protéines

L'article utilise une variété d'indicateurs pour évaluer pleinement les performances de CarbonNovo dans la conception de protéines de novo (Figure 3). Par exemple, la pliabilité, la diversité et la nouveauté sont des indicateurs d'évaluation couramment utilisés dans le domaine. En outre, l'article utilise également l'énergie de Rosetta et la probabilité de vraisemblance (plausibilité des séquences) sous le modèle linguistique comme indicateurs d'évaluation.
CarbonNovo est comparé aux modèles de conception « à deux étages » actuels, tels que RFdiffusion, Chroma, Genie, FrameDiff et FrameFlow. CarbonNovo dépasse considérablement toutes les méthodes de base dans l'indicateur de pliabilité le plus critique, et dépasse également considérablement ou est équivalent à la méthode de base dans d'autres indicateurs.
Pour démontrer les avantages de CarbonNovo dans la conception conjointe de séquences et de structures, les auteurs ont également comparé les résultats de la génération de séquences à l'aide de ProteinMPNN (Figure 3 a-c). On peut observer que le modèle de conception conjointe peut concevoir des structures et des séquences de squelette protéique plus adaptées.

Les auteurs ont évalué en outre les performances de CarbonNovo sur des conceptions de protéines de différentes longueurs (Figure 4). Lors de la conception de protéines plus courtes (par exemple, longueur 100), les modèles fonctionnent tout aussi bien. À mesure que la longueur de la protéine augmente, les performances de conception de CarbonNovo sont nettement meilleures que celles du modèle de conception « en deux étapes ».
Expérience d'ablation

Les auteurs ont formé plusieurs modèles d'ablation pour évaluer la contribution relative des composants clés aux performances de CarbonNovo (Figure 5). Les modèles de langage, les modules de conception de séquences et les pertes de formation auxiliaires contribuent tous aux performances de CarbonNovo. Parmi eux, l’introduction de modèles linguistiques présente la contribution la plus significative. De plus, l’utilisation du module de conception de séquences basé sur l’énergie peut améliorer considérablement les performances de conception de séquences par rapport au modèle autorégressif.
Étude de cas : "interpolation" de la structure protéique
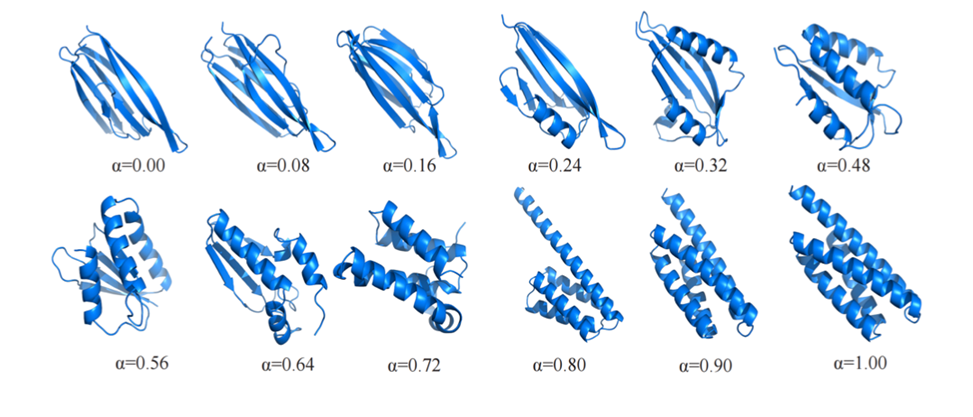
Dans le domaine de la génération d'images, l'interpolation/dégradé d'images de visage est une application classique des modèles génératifs. Les auteurs ont également essayé d’utiliser CarbonNovo pour l’interpolation de la structure des protéines.
La figure 5 montre un exemple représentatif. À mesure que le poids du vecteur de structure entièrement en hélices alpha augmente progressivement dans l'espace latent, la structure entièrement en feuilles bêta générée passera progressivement à la structure entièrement en hélices alpha.
Il s'agit de la première expérience d'interpolation sur la structure des protéines sur le terrain, et elle reflète également que l'espace caché des protéines appris par CarbonNovo est relativement compact.
Conclusion
Enfin, l'auteur a souligné que bien que CarbonNovo se concentre principalement sur la conception de monomères protéiques, il peut également être facilement étendu aux complexes protéiques Conception des matériaux et conception des conditions, telles que la conception des peptides, la conception des anticorps, etc.
L'équipe d'auteurs travaille actuellement avec l'équipe d'expérimentation biologique pour vérifier la protéine conçue par CarbonNovo à travers des expériences humides.
L'équipe CarbonMatrix où travaille l'auteur est engagée depuis longtemps dans la conception de protéines d'IA et la conception de médicaments d'IA, et établit un modèle de génération unifié pour la conception et la prédiction des structures de macromolécules biologiques.
Ses résultats de recherche ont été publiés dans les principales conférences sur l'apprentissage automatique telles que ICML et NeurIPS et dans des revues universitaires de premier plan telles que Nature Machine Intelligence et Nature Communications. Il coopère également actuellement avec des laboratoires de biologie pour promouvoir activement l'application. des modèles d'IA dans la mise en œuvre de l'industrialisation dans le domaine de la conception de médicaments.
以上が中国科学院計算技術研究所のチームは、タンパク質の構造と配列を AI ベースでエンドツーエンドのデノボ設計する CarbonNovo を提案しました。の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。