スタンフォードとマイクロソフトは、オープンソースとなっている拡散モデルを使用してタンパク質構造を生成するために提携しました。
タンパク質は生命にとって不可欠であり、ほぼすべての生物学的プロセスで役割を果たします。一方で、それらはニューロン間で信号を伝達し、微視的な侵入者を識別し、免疫反応を活性化することができます。一方、タンパク質は、病気の治療の一環として治療メディエーターとして広く研究されています。したがって、物理的に折りたためる新しいタンパク質構造を生成することにより、細胞経路を利用して疾患を治療する新しい方法への扉が開かれます。
この記事では、スタンフォード大学、マイクロソフト リサーチ、およびその他の機関の研究者が、生体内でのタンパク質のフォールディング プロセスに触発されて、フォールディング拡散 (フォールディング拡散、FoldingDiff) モデルを導入しました。これは、タンパク質の自然な折り畳みプロセスを反映することによってタンパク質の骨格構造を設計します。

- 論文アドレス: https://arxiv 。 org/pdf/2209.15611.pdf
- コードアドレス: https://github.com/microsoft/foldingdiff
具体的には、タンパク質の骨格構造を、構成アミノ酸残基の相対的な向きを捉える一連の連続した角度として記述します。そして、この表現の回転不変性により、複雑な等変ネットワークの必要性が大幅に軽減されます。
この研究では、トランスのバックボーンに基づいてノイズ除去された拡散確率モデルをトレーニングし、このモデルが天然タンパク質と同様の複雑さと構造パターンを持つ非常に現実的なタンパク質構造を無条件に生成できることを実証しました。
一部のネチズンは次のように述べています。このモデルは AlphaFold に競争をもたらすのだろうか。
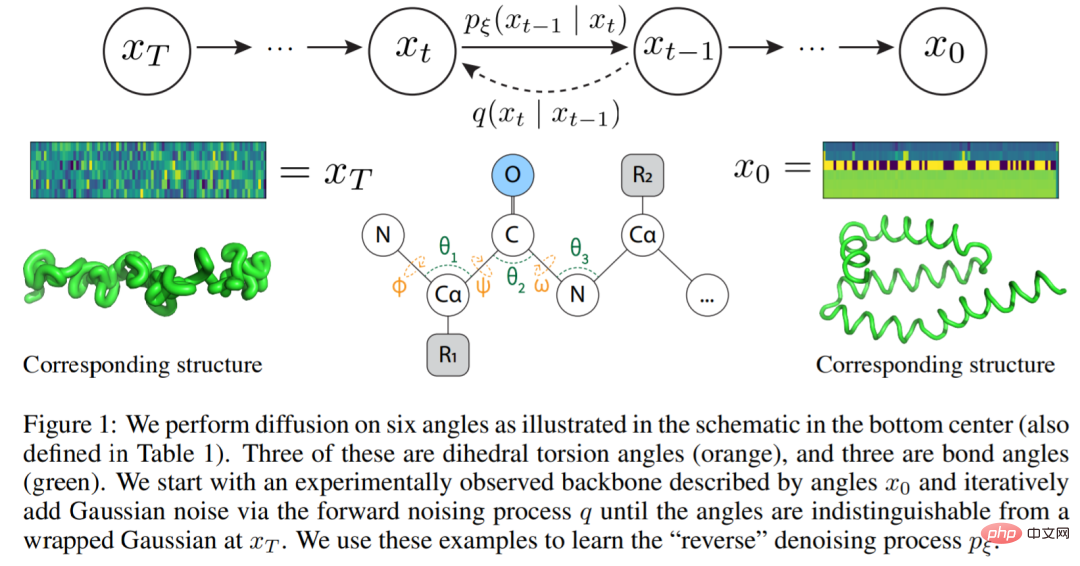
タンパク質は可変長のアミノ酸残基の鎖として理解できます。典型的なアミノ酸は 20 個です。これらの種は、同じ 3 原子 N-C_α-C 骨格を共有していますが、C_α 原子に接続されている異なる側鎖を持っています (通常、R と表示されます、図 1 を参照)。
これらの残基は集合して 3D 構造に折り畳まれるポリマー鎖を形成し、その形状がタンパク質の機能を大きく決定します。これらの折り畳まれた構造は、次の 4 つのレベルを使用して説明できます:
- 一次構造、単純にアミノ酸の直鎖状配列を捕捉します;
- 二次構造はアミノ酸の局所的配置を説明します。
- #三次構造はすべての残基の完全な空間配置を説明します。
- #四次構造はアミノ酸の配置を説明します。複数の異なるアミノ酸鎖が集まって、より大きな複合体を形成します。
この研究では、複雑な等変ネットワークの必要性を排除しながら、タンパク質の折り畳みの生物学的プロセスを追跡する、簡略化されたタンパク質骨格フレームワークを提案しています。彼らは、長さが N 個のアミノ酸であるタンパク質の骨格を 3 次元座標として見るのではなく、それを 6 つの連続した内部角度のシーケンスとして見ました。つまり、現在の残基の位置が与えられると、6 つの内角のベクトルは次の残基のすべての骨格原子の相対位置を表します。これらの内角は、タンパク質骨格に原子を繰り返し追加し、3D デカルト座標に変換し直すという三角関数を使用して簡単に計算できます。
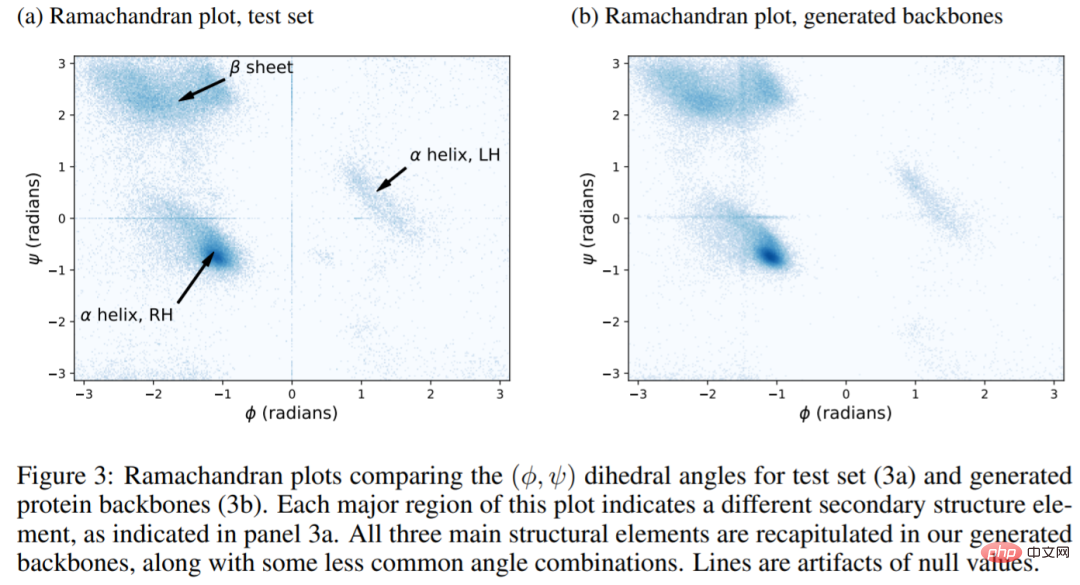
下の図は実験の結果を示しています。天然構造のラマチャンドラン図 (図 a) には、LH α ヘリックス、RH α ヘリックス、β シートに対応する 3 つの領域が含まれています。ここで生成された構造では、3 つの領域すべてが完全に再現されています (図 3b)。言い換えれば、FoldingDiff はタンパク質骨格内に二次構造要素を生成することができます。さらに、実験では、FoldingDiff モデルが RH α ヘリックスが LH α ヘリックスよりも一般的であることを正しく学習していることが示されています。等変ネットワークを使用した以前の研究では、これら 2 種類のスパイラルを区別できませんでした。
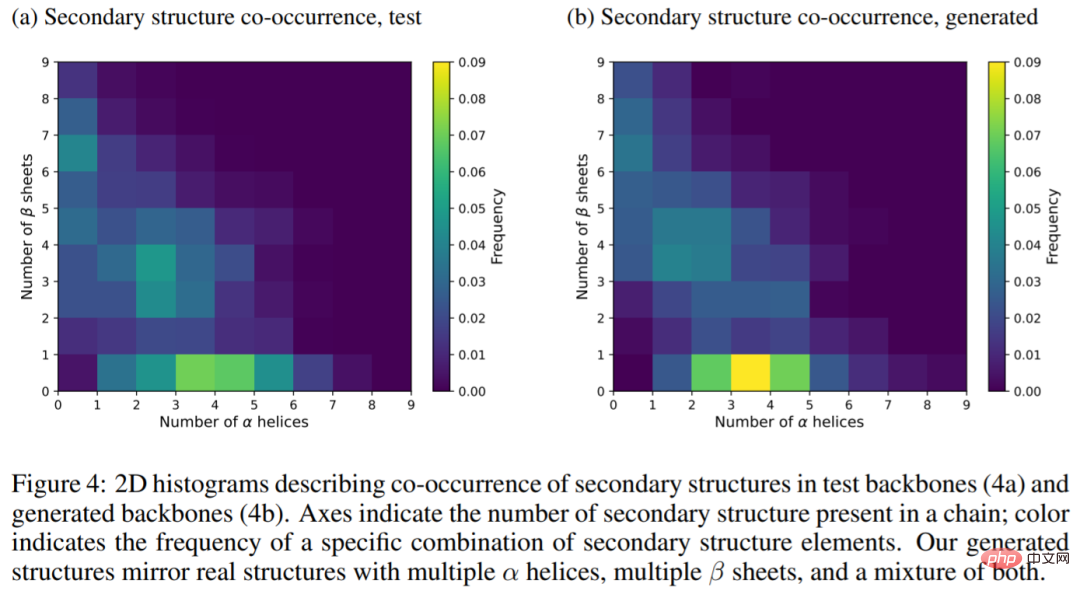
#次の図は、テスト主鎖の二次構造 (4a) と生成された主鎖 (4b) を示しています。二次元ヒストグラムの結果は、生成された構造が、複数のαヘリックス、複数のβシート、およびその 2 つの混合物を含むタンパク質の真の構造を反映していることを示しています。
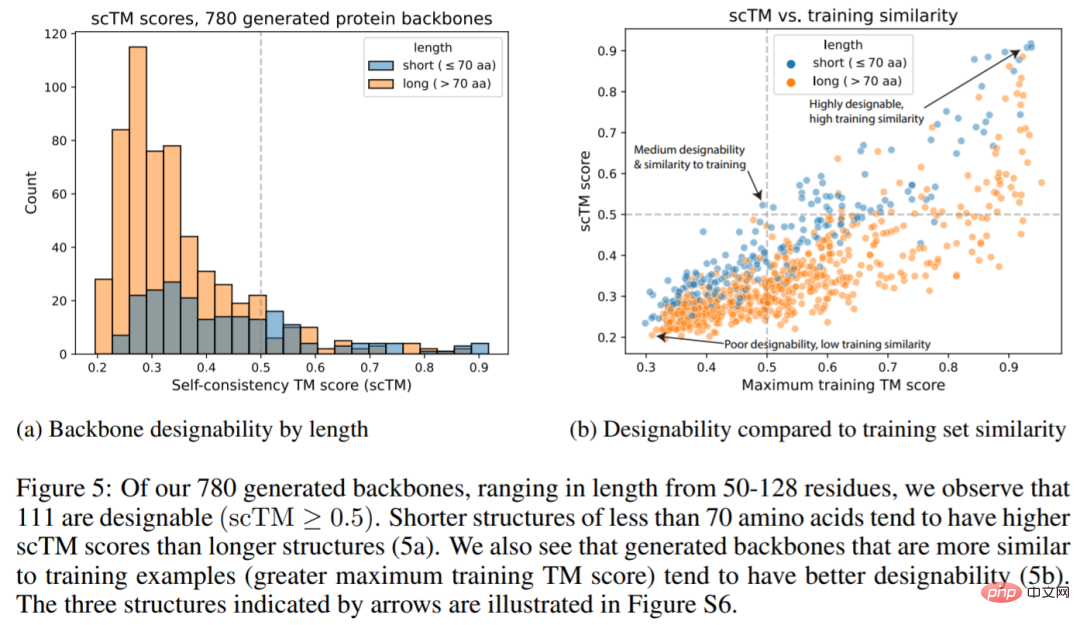
下の図は、生成された 780 個の構造のうち 111 個 (14.2%) が設計可能であり、それらの scTM スコアが ≥0.5 であることを示しています (図 5a)。これは、Trippe et al. が報告した値 11.8% よりも高いです。また、生成されたメインチェーンはトレーニング例により似ており、設計性が向上する傾向があることもわかります (5b)。
詳細については、原論文をお読みください。
以上がスタンフォードとマイクロソフトは、オープンソースとなっている拡散モデルを使用してタンパク質構造を生成するために提携しました。の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。

ホットAIツール

Undresser.AI Undress
リアルなヌード写真を作成する AI 搭載アプリ

AI Clothes Remover
写真から衣服を削除するオンライン AI ツール。

Undress AI Tool
脱衣画像を無料で

Clothoff.io
AI衣類リムーバー

AI Hentai Generator
AIヘンタイを無料で生成します。

人気の記事

ホットツール

メモ帳++7.3.1
使いやすく無料のコードエディター

SublimeText3 中国語版
中国語版、とても使いやすい

ゼンドスタジオ 13.0.1
強力な PHP 統合開発環境

ドリームウィーバー CS6
ビジュアル Web 開発ツール

SublimeText3 Mac版
神レベルのコード編集ソフト(SublimeText3)

ホットトピック
 7465
7465
 15
15
 1376
1376
 52
52
 77
77
 11
11
 18
18
 19
19
 世界で最も強力なオープンソース MoE モデルが登場。GPT-4 に匹敵する中国語機能を備え、価格は GPT-4-Turbo のわずか 1% 近くです
May 07, 2024 pm 04:13 PM
世界で最も強力なオープンソース MoE モデルが登場。GPT-4 に匹敵する中国語機能を備え、価格は GPT-4-Turbo のわずか 1% 近くです
May 07, 2024 pm 04:13 PM
従来のコンピューティングを超える能力を備えているだけでなく、より低コストでより効率的なパフォーマンスを実現する人工知能モデルを想像してみてください。これは SF ではありません。世界で最も強力なオープンソース MoE モデルである DeepSeek-V2[1] が登場しました。 DeepSeek-V2 は、経済的なトレーニングと効率的な推論の特徴を備えた強力な専門家混合 (MoE) 言語モデルです。これは 236B のパラメータで構成されており、そのうち 21B は各マーカーをアクティブにするために使用されます。 DeepSeek67B と比較して、DeepSeek-V2 はパフォーマンスが優れていると同時に、トレーニング コストを 42.5% 節約し、KV キャッシュを 93.3% 削減し、最大生成スループットを 5.76 倍に高めます。 DeepSeek は一般的な人工知能を研究する会社です
 こんにちは、電気アトラスです!ボストン・ダイナミクスのロボットが復活、180度の奇妙な動きにマスク氏も恐怖
Apr 18, 2024 pm 07:58 PM
こんにちは、電気アトラスです!ボストン・ダイナミクスのロボットが復活、180度の奇妙な動きにマスク氏も恐怖
Apr 18, 2024 pm 07:58 PM
Boston Dynamics Atlas は正式に電動ロボットの時代に突入します!昨日、油圧式アトラスが歴史の舞台から「涙ながらに」撤退したばかりですが、今日、ボストン・ダイナミクスは電動式アトラスが稼働することを発表しました。ボストン・ダイナミクス社は商用人型ロボットの分野でテスラ社と競争する決意を持っているようだ。新しいビデオが公開されてから、わずか 10 時間ですでに 100 万人以上が視聴しました。古い人が去り、新しい役割が現れるのは歴史的な必然です。今年が人型ロボットの爆発的な年であることは間違いありません。ネットユーザーは「ロボットの進歩により、今年の開会式は人間のように見え、人間よりもはるかに自由度が高い。しかし、これは本当にホラー映画ではないのか?」とコメントした。ビデオの冒頭では、アトラスは仰向けに見えるように地面に静かに横たわっています。次に続くのは驚くべきことです
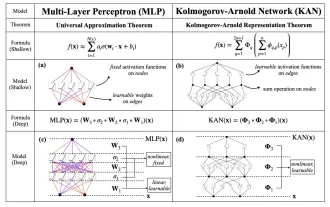 MLP に代わる KAN は、オープンソース プロジェクトによって畳み込みまで拡張されました
Jun 01, 2024 pm 10:03 PM
MLP に代わる KAN は、オープンソース プロジェクトによって畳み込みまで拡張されました
Jun 01, 2024 pm 10:03 PM
今月初め、MIT やその他の機関の研究者らは、MLP に代わる非常に有望な代替案である KAN を提案しました。 KAN は、精度と解釈可能性の点で MLP よりも優れています。また、非常に少数のパラメーターを使用して、多数のパラメーターを使用して実行する MLP よりも優れたパフォーマンスを発揮できます。たとえば、著者らは、KAN を使用して、より小規模なネットワークと高度な自動化で DeepMind の結果を再現したと述べています。具体的には、DeepMind の MLP には約 300,000 個のパラメーターがありますが、KAN には約 200 個のパラメーターしかありません。 KAN は、MLP が普遍近似定理に基づいているのに対し、KAN はコルモゴロフ-アーノルド表現定理に基づいているのと同様に、強力な数学的基礎を持っています。以下の図に示すように、KAN は
 AI が数学研究を破壊する!フィールズ賞受賞者で中国系アメリカ人の数学者が上位 11 件の論文を主導 | テレンス・タオが「いいね!」しました
Apr 09, 2024 am 11:52 AM
AI が数学研究を破壊する!フィールズ賞受賞者で中国系アメリカ人の数学者が上位 11 件の論文を主導 | テレンス・タオが「いいね!」しました
Apr 09, 2024 am 11:52 AM
AI は確かに数学を変えつつあります。最近、この問題に細心の注意を払っている陶哲軒氏が『米国数学協会会報』(米国数学協会会報)の最新号を送ってくれた。 「機械は数学を変えるのか?」というテーマを中心に、多くの数学者が意見を述べ、そのプロセス全体は火花に満ち、ハードコアで刺激的でした。著者には、フィールズ賞受賞者のアクシャイ・ベンカテシュ氏、中国の数学者鄭楽軍氏、ニューヨーク大学のコンピューター科学者アーネスト・デイビス氏、その他業界で著名な学者を含む強力な顔ぶれが揃っている。 AI の世界は劇的に変化しています。これらの記事の多くは 1 年前に投稿されたものです。
 Google は大喜び: JAX のパフォーマンスが Pytorch や TensorFlow を上回りました! GPU 推論トレーニングの最速の選択肢となる可能性があります
Apr 01, 2024 pm 07:46 PM
Google は大喜び: JAX のパフォーマンスが Pytorch や TensorFlow を上回りました! GPU 推論トレーニングの最速の選択肢となる可能性があります
Apr 01, 2024 pm 07:46 PM
Google が推進する JAX のパフォーマンスは、最近のベンチマーク テストで Pytorch や TensorFlow のパフォーマンスを上回り、7 つの指標で 1 位にランクされました。また、テストは最高の JAX パフォーマンスを備えた TPU では行われませんでした。ただし、開発者の間では、依然として Tensorflow よりも Pytorch の方が人気があります。しかし、将来的には、おそらくより大規模なモデルが JAX プラットフォームに基づいてトレーニングされ、実行されるようになるでしょう。モデル 最近、Keras チームは、ネイティブ PyTorch 実装を使用して 3 つのバックエンド (TensorFlow、JAX、PyTorch) をベンチマークし、TensorFlow を使用して Keras2 をベンチマークしました。まず、主流のセットを選択します
 推奨: 優れた JS オープンソースの顔検出および認識プロジェクト
Apr 03, 2024 am 11:55 AM
推奨: 優れた JS オープンソースの顔検出および認識プロジェクト
Apr 03, 2024 am 11:55 AM
顔の検出および認識テクノロジーは、すでに比較的成熟しており、広く使用されているテクノロジーです。現在、最も広く使用されているインターネット アプリケーション言語は JS ですが、Web フロントエンドでの顔検出と認識の実装には、バックエンドの顔認識と比較して利点と欠点があります。利点としては、ネットワーク インタラクションの削減とリアルタイム認識により、ユーザーの待ち時間が大幅に短縮され、ユーザー エクスペリエンスが向上することが挙げられます。欠点としては、モデル サイズによって制限されるため、精度も制限されることが挙げられます。 js を使用して Web 上に顔検出を実装するにはどうすればよいですか? Web 上で顔認識を実装するには、JavaScript、HTML、CSS、WebRTC など、関連するプログラミング言語とテクノロジに精通している必要があります。同時に、関連するコンピューター ビジョンと人工知能テクノロジーを習得する必要もあります。 Web 側の設計により、次の点に注意してください。
 Alibaba 7B マルチモーダル文書理解の大規模モデルが新しい SOTA を獲得
Apr 02, 2024 am 11:31 AM
Alibaba 7B マルチモーダル文書理解の大規模モデルが新しい SOTA を獲得
Apr 02, 2024 am 11:31 AM
マルチモーダル文書理解機能のための新しい SOTA!アリババの mPLUG チームは、最新のオープンソース作品 mPLUG-DocOwl1.5 をリリースしました。これは、高解像度の画像テキスト認識、一般的な文書構造の理解、指示の遵守、外部知識の導入という 4 つの主要な課題に対処するための一連のソリューションを提案しています。さっそく、その効果を見てみましょう。複雑な構造のグラフをワンクリックで認識しMarkdown形式に変換:さまざまなスタイルのグラフが利用可能:より詳細な文字認識や位置決めも簡単に対応:文書理解の詳しい説明も可能:ご存知「文書理解」 「」は現在、大規模な言語モデルの実装にとって重要なシナリオです。市場には文書の読み取りを支援する多くの製品が存在します。その中には、主にテキスト認識に OCR システムを使用し、テキスト処理に LLM と連携する製品もあります。
 テスラのロボットは工場で働く、マスク氏:手の自由度は今年22に達する!
May 06, 2024 pm 04:13 PM
テスラのロボットは工場で働く、マスク氏:手の自由度は今年22に達する!
May 06, 2024 pm 04:13 PM
テスラのロボット「オプティマス」の最新映像が公開され、すでに工場内で稼働可能となっている。通常の速度では、バッテリー(テスラの4680バッテリー)を次のように分類します:公式は、20倍の速度でどのように見えるかも公開しました - 小さな「ワークステーション」上で、ピッキング、ピッキング、ピッキング:今回は、それがリリースされたハイライトの1つビデオの内容は、オプティマスが工場内でこの作業を完全に自律的に行い、プロセス全体を通じて人間の介入なしに完了するというものです。そして、オプティマスの観点から見ると、自動エラー修正に重点を置いて、曲がったバッテリーを拾い上げたり配置したりすることもできます。オプティマスのハンドについては、NVIDIA の科学者ジム ファン氏が高く評価しました。オプティマスのハンドは、世界の 5 本指ロボットの 1 つです。最も器用。その手は触覚だけではありません




