Python遺伝アルゴリズムgeatpyのインストールと使い方
1. geatpy のインストール
最初のステップは geatpy をインストールすることで、pip3 コマンドを使用してインストールします:
pip3 install geatpy
次のプロンプトが表示されます: インストールの成功:
2. geatpy の基本データ構造
geatpy のデータのほとんどは、numpy 配列を使用して保存および計算されます。 , 以下では、遺伝的アルゴリズムの概念が numpy データによってどのように表現されるか、および行と列の意味を紹介します。
2.1 集団染色体
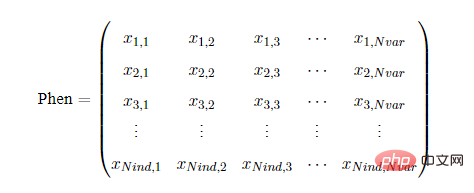
遺伝的アルゴリズムで最も重要なことは、個人の染色体表現です。geatpy では、集団染色体は Chrom で表現されます。これは 2 次元配列です。染色体のエンコーディングである Chrom の構造は次のとおりです: lind はエンコーディングの長さを表し、Nind は集団のサイズ (個体数) を表します。

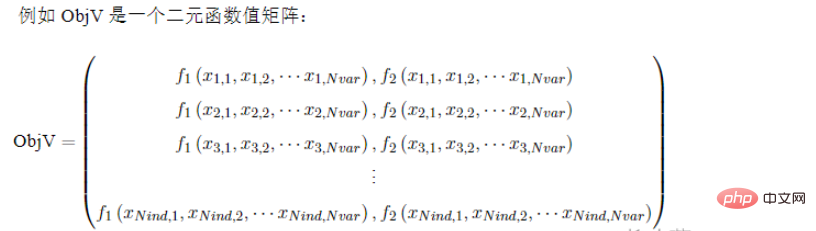
##2.4 個別の適応度
Geatpy は列ベクトルを使用して母集団の個別の適応度を保存します。 (フィットネス関数が計算されます)。一般に FitnV と呼ばれるこれも numpy の配列型であり、各行が母集団行列の各個人に対応します。そのため、Chrom と同じ行数があり、FitnV の形式は次のとおりです:

注: Geatpy のフィットネスは「最小フィットネスは 0」に従います。合意。
2.5 制約違反度行列Geatpy は、numpy 配列型行列 CV (制約違反値) を使用して、個々の母集団がさまざまな制約に違反する程度を保存します。 CV という名前が付けられ、その各行は母集団の各個人に対応するため、Chrom と同じ数の行があり、各列は制約に対応するため、制約がある場合、CV 行列には 1 つの列のみが含まれます。制約が複数ある場合、CV マトリックスには複数の列が含まれます。 num 制約がある場合、CV マトリックスの構造は次のようになります。
- BG: (バイナリ/グレー コード)
- RI : ((実数符号化、つまり実数と整数の混合符号化)
- P: (順列符号化、つまり各染色体の要素が互いに異なる)
注:’RI’和’P’编码的染色体都不需要解码,染色体上的每一位本身就代表着决策变量的真实值,因此“实整数编码”和“排列编码”可统称为“实值编码”
以BG编码为例,我们展示一下编译矩阵FieldD。FieldD的结构如下:
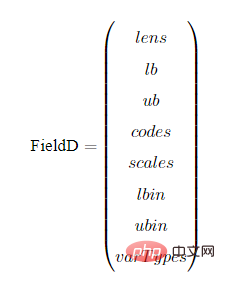
其中,lens,lb,ub,codes,scales,lbin,ubin,varTypes都是行向量,其长度等于决策变量的个数。
lens:代表以条染色体中,每个子染色体的长度。
lb:代表每个变量的上界
ub:代表每个变量的下界
codes:代表染色体字串用的编码方式,[1,0,1]代表第一个变量用的格雷编码,第二个变量用的二进制编码,第3个变量用的格雷编码。
scales:指明每个子串用的是算术刻度还是对数刻度。scales[i] = 0为算术刻度,scales[i] = 1为对数刻度(对数刻度很少用,可以忽略。)
lbin:代表变量上界是否包含其范围边界。0代表不包含,1代表包含。‘[ ’和 ‘(’ 的区别
ubin:代表变量下界是否包含其范围边界。0代表不包含,1代表包含。
varTypes:代表决策变量的类型,元素为0表示对应位置的决策变量是连续型变量;1表示对应的是离散型变量。
例如:有以下一个译码矩阵

它表示待解码的种群染色体矩阵Chrom解码后可以表示成3个决策变量,每个决策变量的取值范围分别是[1,10], [2,9], [3,15]。其中第一第二个变量采用的是二进制编码,第三个变量采用的是格雷编码,且第一、第三个决策变量为连续型变量;第二个为离散型变量。
#通过种群染色体chrom和译码矩阵FieldD,可解码成种群表现型矩阵。 import geatpy as ea Phen = ea.bs2ri(Chrom, FieldD)
2.7 进化追踪器
在使用Geatpy进行进化算法编程时,常常建立一个进化追踪器(如pop_trace)来记录种群在进化的过程中各代的最优个体,尤其是采用无精英保留机制时,进化追踪器帮助我们记录种群在进化过程中的最优个体。待进化完成后,再从进化追踪器中挑选出“历史最优”的个体。这种进化记录器有多种,其中一种是numpy的array类型的,结构如下:其中MAXGEN是种群进化的代数(迭代次数)。
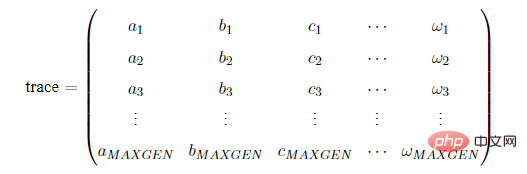
trace的每一列代表不同的指标,比如第一列记录各代种群的最佳目标函数值,第二列记录各代种群的平均目标函数值…trace的每一行对应每一代,如第一行代表第一代,第二行代表第二代…另外一种进化记录器是一个列表,列表中的每一个元素都是一个拥有相同数据类型的数据。比如在Geatpy的面向对象进化算法框架中的pop_trace,它是一个列表,列表中的每一个元素都是历代的种群对象。
3. geatpy的种群结构
3.1 Population类
在Geatpy提供的面向对象进化算法框架中,种群类(Population)是一个存储着与种群个体相关信息的类。它有以下基本属性:
sizes : int -种群规模,即种群的个体数目。
ChromNum : int -染色体的数目,即每个个体有多少条染色体。
Encoding : str -染色体编码方式。
Field : array -译码矩阵,可以是FieldD或FieldDR。
Chrom : array -种群染色体矩阵,每一行对应一个个体的一条染色体。
Lind : int -种群染色体长度。
ObjV : array -种群目标函数值矩阵。
FitnV : array -种群个体适应度列向量。
CV : array -种群个体违反约束条件程度的矩阵。
Phen : array -种群表现型矩阵。
可以直接对种群对象进行提取个体、个体合并等操作,比如pop1和pop2是两个种群对象,则通过语句“pop3 = pop1 + pop2”,即可把两个种群的个体合并,得到一个新的种群。在合并的过程中,实际上是把种群的各个属性进行合并,然后用合并的数据来生成一个新的种群(详见Population.py)。又比如执行语句“pop3 = pop1[[0]]”,可以把种群的第0号个体抽取出来,得到一个新的只有一个个体的种群对象pop3。值得注意的是,种群的这种个体抽取操作要求下标必须为列表或是Numpy array类型的行向量,不能是标量(详见Population.py)
3.2 PsyPopulation类
PsyPopulation类是Population的子类,它提供Population类所不支持的多染色体混合编码。它有以下基本属性:
sizes : int -种群规模,即种群的个体数目。
ChromNum : int -染色体的数目,即每个个体有多少条染色体。
Encodings : list -存储各染色体编码方式的列表。
Fields : list -存储各染色体对应的译码矩阵的列表。
Chroms : list -存储种群各染色体矩阵的列表。
Linds : list -存储种群各染色体长度的列表。
ObjV : array -种群目标函数值矩阵。
FitnV : array -种群个体适应度列向量。
CV : array -种群个体违反约束条件程度的矩阵。
Phen : array -种群表现型矩阵。
可见PsyPopulation类基本与Population类一样,不同之处是采用Linds、Encodings、Fields和Chroms分别存储多个Lind、Encoding、Field和Chrom。
PsyPopulation类的对象往往与带“psy”字样的进化算法模板配合使用,以实现多染色体混合编码的进化优化。
4. 求解标准测试函数——McCormick函数
遗传算法求解以下函数的最小值:
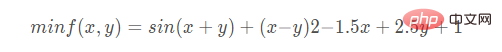
代码实现:
#-*-coding:utf-8-*-
import numpy as np
import geatpy as ea#导入geatpy库
import time
"""============================目标函数============================"""
def aim(Phen):#传入种群染色体矩阵解码后的基因表现型矩阵
x1 = Phen[:, [0]]#取出第一列,得到所有个体的第一个自变量
x2 = Phen[:, [1]]#取出第二列,得到所有个体的第二个自变量
return np.sin(x1 + x2) + (x1 - x2) ** 2 - 1.5 * x1 + 2.5 * x2+1
"""============================变量设置============================"""
x1 = [-1.5, 4]#第一个决策变量范围
x2 = [-3, 4]#第二个决策变量范围
b1 = [1, 1]#第一个决策变量边界,1表示包含范围的边界,0表示不包含
b2 = [1, 1]#第二个决策变量边界,1表示包含范围的边界,0表示不包含
#生成自变量的范围矩阵,使得第一行为所有决策变量的下界,第二行为上界
ranges=np.vstack([x1, x2]).T
#生成自变量的边界矩阵
borders=np.vstack([b1, b2]).T
varTypes = np.array([0, 0])#决策变量的类型,0表示连续,1表示离散
"""==========================染色体编码设置========================="""
Encoding ='BG'#'BG'表示采用二进制/格雷编码
codes = [1, 1]#决策变量的编码方式,两个1表示变量均使用格雷编码
precisions =[6, 6]#决策变量的编码精度,表示解码后能表示的决策变量的精度可达到小数点后6位
scales = [0, 0]#0表示采用算术刻度,1表示采用对数刻度#调用函数创建译码矩阵
FieldD =ea.crtfld(Encoding,varTypes,ranges,borders,precisions,codes,scales)
"""=========================遗传算法参数设置========================"""
NIND = 20#种群个体数目
MAXGEN = 100#最大遗传代数
maxormins = np.array([1])#表示目标函数是最小化,元素为-1则表示对应的目标函数是最大化
selectStyle ='sus'#采用随机抽样选择
recStyle ='xovdp'#采用两点交叉
mutStyle ='mutbin'#采用二进制染色体的变异算子
Lind =int(np.sum(FieldD[0, :]))#计算染色体长度
pc= 0.9#交叉概率
pm= 1/Lind#变异概率
obj_trace = np.zeros((MAXGEN, 2))#定义目标函数值记录器
var_trace = np.zeros((MAXGEN, Lind))#染色体记录器,记录历代最优个体的染色体
"""=========================开始遗传算法进化========================"""
start_time = time.time()#开始计时
Chrom = ea.crtpc(Encoding,NIND, FieldD)#生成种群染色体矩阵
variable = ea.bs2ri(Chrom, FieldD)#对初始种群进行解码
ObjV = aim(variable)#计算初始种群个体的目标函数值
best_ind = np.argmin(ObjV)#计算当代最优个体的序号
#开始进化
for gen in range(MAXGEN):
FitnV = ea.ranking(maxormins * ObjV)#根据目标函数大小分配适应度值
SelCh = Chrom[ea.selecting(selectStyle,FitnV,NIND-1),:]#选择
SelCh = ea.recombin(recStyle, SelCh, pc)#重组
SelCh = ea.mutate(mutStyle, Encoding, SelCh, pm)#变异
# #把父代精英个体与子代的染色体进行合并,得到新一代种群
Chrom = np.vstack([Chrom[best_ind, :], SelCh])
Phen = ea.bs2ri(Chrom, FieldD)#对种群进行解码(二进制转十进制)
ObjV = aim(Phen)#求种群个体的目标函数值
#记录
best_ind = np.argmin(ObjV)#计算当代最优个体的序号
obj_trace[gen,0]=np.sum(ObjV)/ObjV.shape[0]#记录当代种群的目标函数均值
obj_trace[gen,1]=ObjV[best_ind]#记录当代种群最优个体目标函数值
var_trace[gen,:]=Chrom[best_ind,:]#记录当代种群最优个体的染色体
# 进化完成
end_time = time.time()#结束计时
ea.trcplot(obj_trace, [['种群个体平均目标函数值','种群最优个体目标函数值']])#绘制图像
"""============================输出结果============================"""
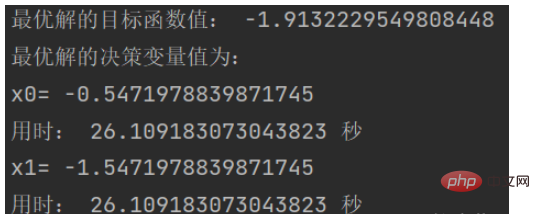
best_gen = np.argmin(obj_trace[:, [1]])
print('最优解的目标函数值:', obj_trace[best_gen, 1])
variable = ea.bs2ri(var_trace[[best_gen], :], FieldD)#解码得到表现型(即对应的决策变量值)
print('最优解的决策变量值为:')
for i in range(variable.shape[1]):
print('x'+str(i)+'=',variable[0, i])
print('用时:', end_time - start_time,'秒')效果图:
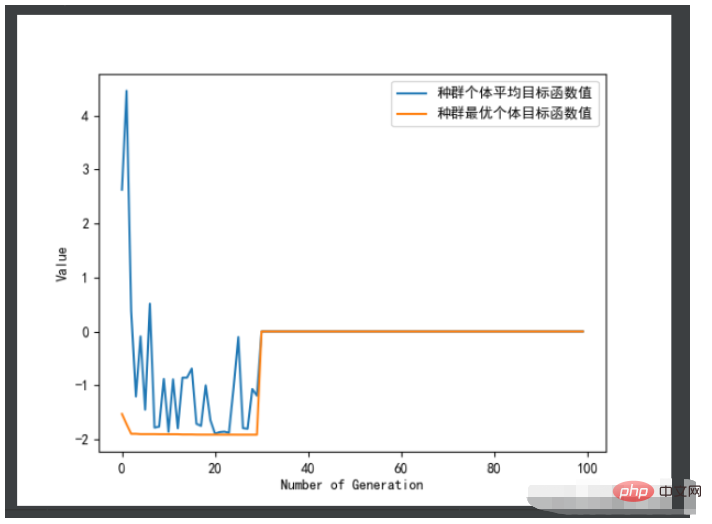
结果如下:
以上がPython遺伝アルゴリズムgeatpyのインストールと使い方の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。

ホットAIツール

Undresser.AI Undress
リアルなヌード写真を作成する AI 搭載アプリ

AI Clothes Remover
写真から衣服を削除するオンライン AI ツール。

Undress AI Tool
脱衣画像を無料で

Clothoff.io
AI衣類リムーバー

Video Face Swap
完全無料の AI 顔交換ツールを使用して、あらゆるビデオの顔を簡単に交換できます。

人気の記事

ホットツール

メモ帳++7.3.1
使いやすく無料のコードエディター

SublimeText3 中国語版
中国語版、とても使いやすい

ゼンドスタジオ 13.0.1
強力な PHP 統合開発環境

ドリームウィーバー CS6
ビジュアル Web 開発ツール

SublimeText3 Mac版
神レベルのコード編集ソフト(SublimeText3)

ホットトピック
 7691
7691
 15
15
 1639
1639
 14
14
 1393
1393
 52
52
 1287
1287
 25
25
 1229
1229
 29
29
 PHPおよびPython:さまざまなパラダイムが説明されています
Apr 18, 2025 am 12:26 AM
PHPおよびPython:さまざまなパラダイムが説明されています
Apr 18, 2025 am 12:26 AM
PHPは主に手順プログラミングですが、オブジェクト指向プログラミング(OOP)もサポートしています。 Pythonは、OOP、機能、手続き上のプログラミングなど、さまざまなパラダイムをサポートしています。 PHPはWeb開発に適しており、Pythonはデータ分析や機械学習などのさまざまなアプリケーションに適しています。
 PHPとPythonの選択:ガイド
Apr 18, 2025 am 12:24 AM
PHPとPythonの選択:ガイド
Apr 18, 2025 am 12:24 AM
PHPはWeb開発と迅速なプロトタイピングに適しており、Pythonはデータサイエンスと機械学習に適しています。 1.PHPは、単純な構文と迅速な開発に適した動的なWeb開発に使用されます。 2。Pythonには簡潔な構文があり、複数のフィールドに適しており、強力なライブラリエコシステムがあります。
 Python vs. JavaScript:学習曲線と使いやすさ
Apr 16, 2025 am 12:12 AM
Python vs. JavaScript:学習曲線と使いやすさ
Apr 16, 2025 am 12:12 AM
Pythonは、スムーズな学習曲線と簡潔な構文を備えた初心者により適しています。 JavaScriptは、急な学習曲線と柔軟な構文を備えたフロントエンド開発に適しています。 1。Python構文は直感的で、データサイエンスやバックエンド開発に適しています。 2。JavaScriptは柔軟で、フロントエンドおよびサーバー側のプログラミングで広く使用されています。
 Windows 8でコードを実行できます
Apr 15, 2025 pm 07:24 PM
Windows 8でコードを実行できます
Apr 15, 2025 pm 07:24 PM
VSコードはWindows 8で実行できますが、エクスペリエンスは大きくない場合があります。まず、システムが最新のパッチに更新されていることを確認してから、システムアーキテクチャに一致するVSコードインストールパッケージをダウンロードして、プロンプトとしてインストールします。インストール後、一部の拡張機能はWindows 8と互換性があり、代替拡張機能を探すか、仮想マシンで新しいWindowsシステムを使用する必要があることに注意してください。必要な拡張機能をインストールして、適切に動作するかどうかを確認します。 Windows 8ではVSコードは実行可能ですが、開発エクスペリエンスとセキュリティを向上させるために、新しいWindowsシステムにアップグレードすることをお勧めします。
 PHPとPython:彼らの歴史を深く掘り下げます
Apr 18, 2025 am 12:25 AM
PHPとPython:彼らの歴史を深く掘り下げます
Apr 18, 2025 am 12:25 AM
PHPは1994年に発信され、Rasmuslerdorfによって開発されました。もともとはウェブサイトの訪問者を追跡するために使用され、サーバー側のスクリプト言語に徐々に進化し、Web開発で広く使用されていました。 Pythonは、1980年代後半にGuidovan Rossumによって開発され、1991年に最初にリリースされました。コードの読みやすさとシンプルさを強調し、科学的コンピューティング、データ分析、その他の分野に適しています。
 ターミナルVSCODEでプログラムを実行する方法
Apr 15, 2025 pm 06:42 PM
ターミナルVSCODEでプログラムを実行する方法
Apr 15, 2025 pm 06:42 PM
VSコードでは、次の手順を通じて端末でプログラムを実行できます。コードを準備し、統合端子を開き、コードディレクトリが端末作業ディレクトリと一致していることを確認します。プログラミング言語(pythonのpython your_file_name.pyなど)に従って実行コマンドを選択して、それが正常に実行されるかどうかを確認し、エラーを解決します。デバッガーを使用して、デバッグ効率を向上させます。
 VSCODE拡張機能は悪意がありますか?
Apr 15, 2025 pm 07:57 PM
VSCODE拡張機能は悪意がありますか?
Apr 15, 2025 pm 07:57 PM
VSコード拡張機能は、悪意のあるコードの隠れ、脆弱性の活用、合法的な拡張機能としての自慰行為など、悪意のあるリスクを引き起こします。悪意のある拡張機能を識別する方法には、パブリッシャーのチェック、コメントの読み取り、コードのチェック、およびインストールに注意してください。セキュリティ対策には、セキュリティ認識、良好な習慣、定期的な更新、ウイルス対策ソフトウェアも含まれます。
 Visual StudioコードはPythonで使用できますか
Apr 15, 2025 pm 08:18 PM
Visual StudioコードはPythonで使用できますか
Apr 15, 2025 pm 08:18 PM
VSコードはPythonの書き込みに使用でき、Pythonアプリケーションを開発するための理想的なツールになる多くの機能を提供できます。ユーザーは以下を可能にします。Python拡張機能をインストールして、コードの完了、構文の強調表示、デバッグなどの関数を取得できます。デバッガーを使用して、コードを段階的に追跡し、エラーを見つけて修正します。バージョンコントロールのためにGitを統合します。コードフォーマットツールを使用して、コードの一貫性を維持します。糸くずツールを使用して、事前に潜在的な問題を発見します。




