世界初:タンパク質側鎖予測と配列設計の問題を克服する、Molecular Heart のオープンソースの新しい AI アルゴリズム
タンパク質の構造と機能の形成は側鎖原子間の相互作用に大きく依存するため、正確なタンパク質側鎖予測 (PSCP) はタンパク質の構造予測とタンパク質設計の問題を解決する鍵となります。しかし、これまでのタンパク質の構造予測は主鎖の構造に焦点が当てられており、側鎖の構造予測は完全には解決されていない難しい問題でした。
最近、Molecular Heart の Xu Jinbo チームは、速度、メモリ効率、全体的な精度の大幅な向上を達成した新しい PSCP ディープ アーキテクチャ AttnPacker を立ち上げました。これは現在最もよく知られている側面です。鎖構造予測アルゴリズムは、タンパク質の側鎖と配列設計を同時に予測できる世界初のAIアルゴリズムでもある。
この論文は米国科学アカデミー紀要 (PNAS) に掲載され、その事前トレーニング済みモデル、ソース コード、推論スクリプトは Github でオープンソース化されています。

- 紙のリンク: https://www.pnas.org /doi/10.1073/pnas.2216438120#supplementary-materials
- オープンソースリンク: https://github.com/MattMcPartlon/AttnPacker
タンパク質はいくつかのアミノ酸から折りたたまれており、その構造は主鎖と側鎖に分かれています。側鎖の違いは、タンパク質の構造と機能、特に生物活性に大きな影響を与えます。側鎖構造の明確な理解に基づいて、科学者はタンパク質の三次元構造をより正確に決定し、タンパク質間相互作用を分析し、合理的なタンパク質設計を行うことができます。薬物設計の分野に適用すると、科学者は薬物と受容体に適した結合部位を迅速かつより正確に見つけ出し、必要に応じて結合部位を最適化または設計することもできます;酵素最適化の分野では、科学者は配列を最適化できます変換により複数の側鎖が可能になります触媒反応に参加して、より効率的かつ特異的な触媒効果を達成します。
現在のタンパク質構造予測アルゴリズムのほとんどは主鎖の構造解析に焦点を当てていますが、タンパク質の側鎖構造予測は依然として完全には克服されていない問題です。 AlphaFold2 などの一般的なタンパク質構造予測アルゴリズムであっても、DLPacker や RosettaPacker などの側鎖構造予測に重点を置いたアルゴリズムであっても、精度や速度が満足できるものではありません。これはタンパク質の設計にも制限を課します。
RosettaPacker などの従来の方法では、主にエネルギー最適化方法が使用され、最初に側鎖原子の分布をグループ化し、次に特定のアミノ酸の側鎖のグループ化を検索して見つけます。最小エネルギーの組み合わせ。これらの方法は、研究者が選択した回転異性体ライブラリー、エネルギー関数、エネルギー最小化手順とは主に異なり、検索ヒューリスティックや離散サンプリング手順の使用によって精度が制限されます。業界には、PSCP を画像から画像への変換問題として定式化し、U-net モデル構造を採用する DLPacker など、深層学習に基づくサイドチェーン予測手法もあります。ただし、予測の精度と速度はまだ理想的ではありません。
メソッドAttnPacker は、タンパク質側鎖の座標を予測するためのエンドツーエンドの深層学習メソッドです。側鎖相互作用を共同でシミュレートし、原子衝突が少なく、より理想的な結合長と角度を備えた、より物理的に実現可能な側鎖構造を直接予測します。
具体的には、AttnPacker は、PSCP の幾何学的な側面とリレーショナルな側面を活用する深度マップ コンバーター アーキテクチャを導入しています。 AlphaFold2 からインスピレーションを得た Molecular Heart は、三角形のアテンションと乗算更新を計算するグラフベースのフレームワークを使用してペアごとの特徴を最適化する、位置を認識した三角形の更新を提案します。このアプローチにより、AttnPacker のメモリは大幅に削減され、より大容量のモデルになります。さらに、Molecular Heart は、いくつかの SE (3) 等変注意メカニズムを調査し、3D ポイントから学習するための等変トランスフォーマー アーキテクチャを提案しています。
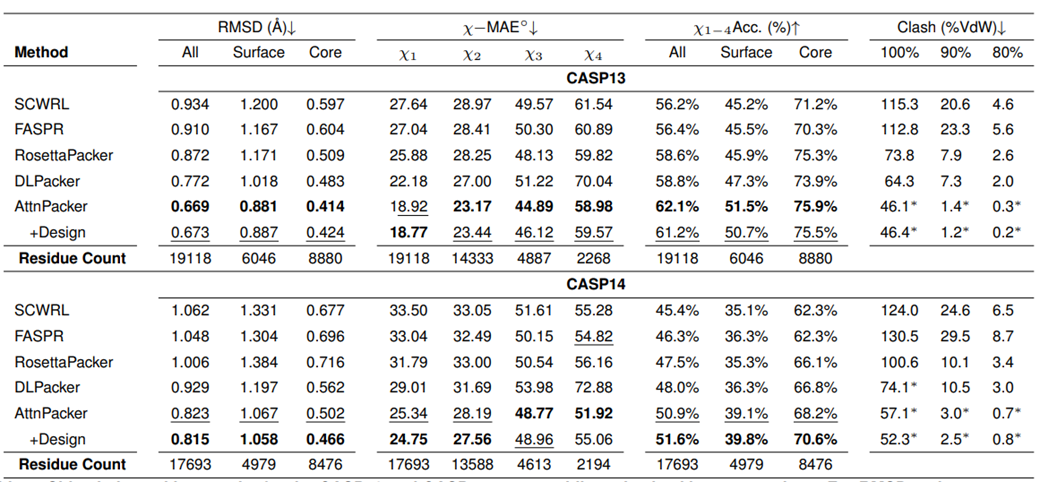
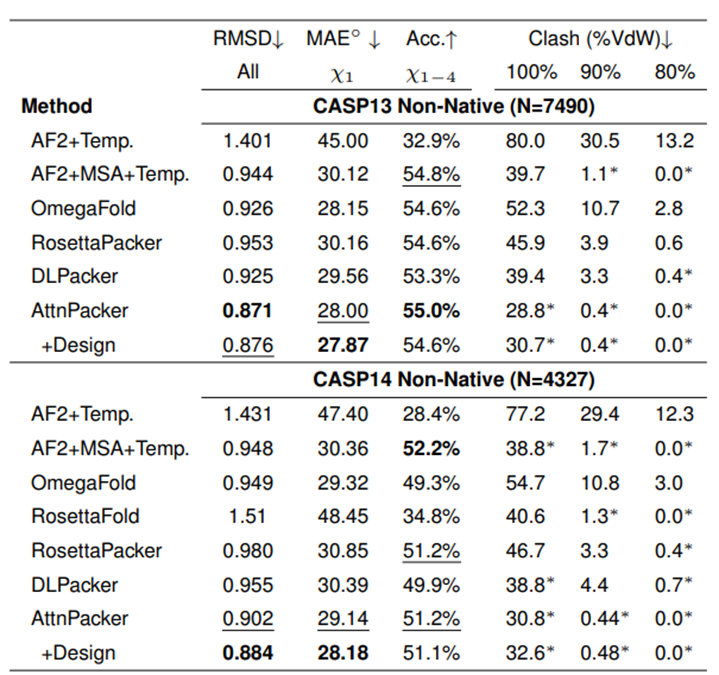
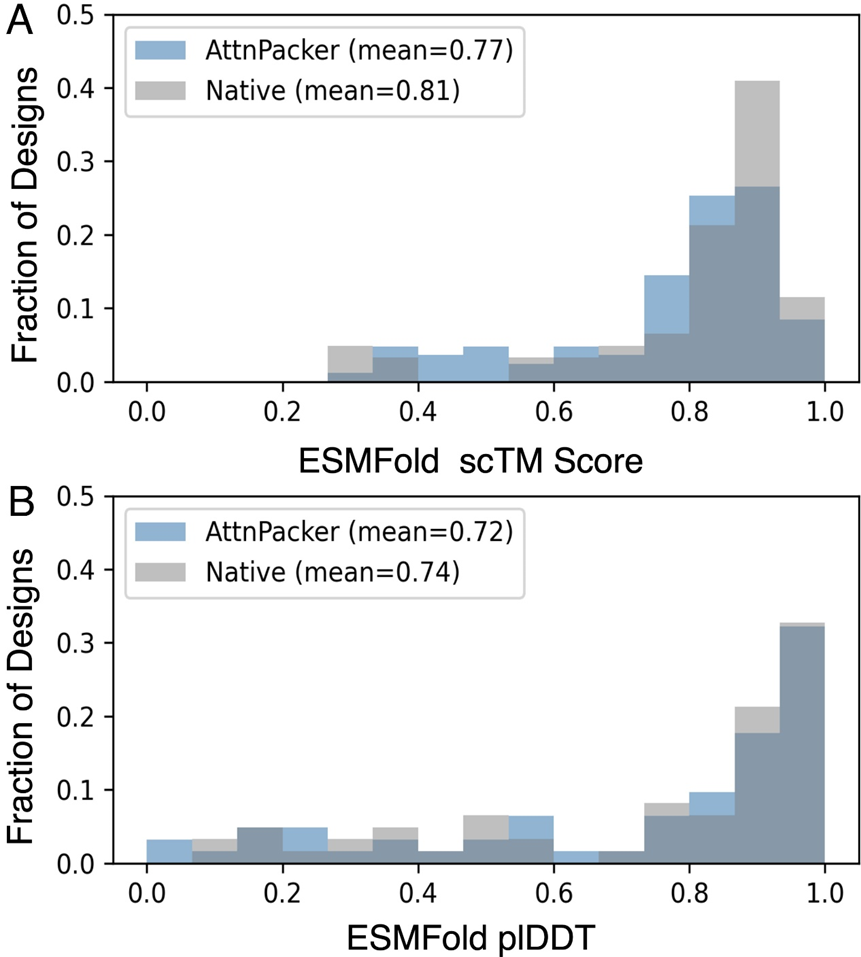
AttnPacker がプロセスを実行します。タンパク質骨格の座標と配列が入力として使用され、その座標情報に基づいて空間特徴マップと等変基底が導出されます。特徴マップは、不変グラフ トランスフォーマー モジュールによって処理され、予測された側鎖座標、各残基の信頼スコア、およびオプションの設計配列を出力する等変 TFN トランスフォーマーに渡されます。予測された座標は後処理されて、すべての空間的矛盾が除去され、理想的なジオメトリが保証されます。 予測パフォーマンスの観点から、AttnPacker は天然および非天然の両方のバックボーン構造の精度と効率が向上していることを示しています。同時に、物理的な実現可能性が確保され、理想的な結合の長さと角度からの逸脱は無視でき、原子の立体障害が最小限に抑えられます。 Molecular Heart は、AttnPacker と現在の最先端の方法 (SCWRL4、FASPR、RosettaPacker、DLPacker) の比較テストを CASP13 および CASP14 の天然および非天然タンパク質骨格に対して実施します。データセット。結果は、AttnPacker が CASP13 および CASP14 ネイティブ バックボーンに対して従来のタンパク質側鎖予測法を大幅に上回っており、平均再構成 RMSD が各テスト セットで次善の方法より 18% 以上低いことを示しています。また、AttnPacker は深層学習手法 DLPacker よりも優れたパフォーマンスを示し、平均 RMSD を 11% 以上削減すると同時に、サイドチェーンの二面角の精度も大幅に向上させます。精度に加えて、AttnPacker は他の方法よりもアトミック衝突が大幅に少ないです。 自然な主鎖構造が与えられると、各アルゴリズムは CASP13 で実行され、 CASP14 標的タンパク質の側鎖構造予測結果。アスタリスクは、平均競合値がネイティブ構造よりも低いことを示します (CASP13 では 56.0、5.9、および 0.4、CASP14 では 80.4、7.9、および 2.5)。 CASP13 および CASP14 の非ネイティブ バックボーンでは、AttnPacker は他の方法よりも大幅に優れており、アトミック衝突も他の方法よりも大幅に少なくなっています。 #非自然な主鎖構造が与えられた場合、各アルゴリズムは次のようになります。 CASP13 および CASP14 標的タンパク質の側鎖構造予測結果。アスタリスクは、平均競合値が対応するネイティブ構造よりも低いことを示します (CASP13 では 34.6、2.2、0.5、CASP14 では 40.0、2.7、0.7)。
さまざまな PSCP 方式の時間の比較。 83 個すべての CASP13 標的タンパク質の側鎖原子の相対時間を再構築します。
AttnPaker には、その驚くべき効果と効率に加えて、非常に使いやすいという非常に実用的な価値もあります。 AttnPaker を実行するにはタンパク質構造ファイルのみが必要です。対照的に、OPUS-Rota4 (28) は、DLPacker からの原子環境のボクセル表現、trRosetta100 からのロジック、二次構造、および OPUS-CM 出力からの制約ファイルを必要とします。さらに、AttnPacker は側鎖の座標を直接予測するため、出力は完全に微分可能であり、最適化やタンパク質間相互作用などの下流の予測タスクが容易になります。 「優れた予測効果、高効率、使いやすさという利点は、研究分野や産業分野でのAttnPackerの広範な使用に貢献します。」とXu Jinbo教授は述べました。 1. AttnPacker は、配列および側鎖座標を直接予測するために使用される SE (3) 等変モデルであり、タンパク質側鎖構造の予測に使用できます。 . タンパク質の配列設計にも使用でき、先駆的な研究です。 2. AttnPacker は他の方法に比べて精度が高く、効率が大幅に向上しており、非常に使いやすいです。 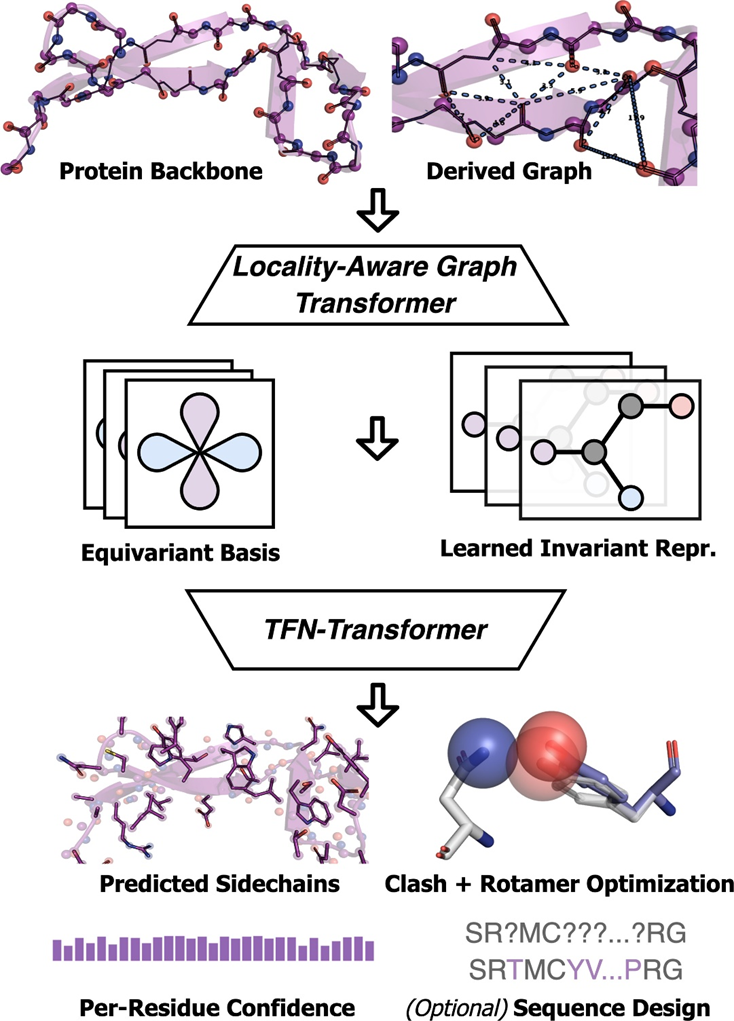
効果
概要
以上が世界初:タンパク質側鎖予測と配列設計の問題を克服する、Molecular Heart のオープンソースの新しい AI アルゴリズムの詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。

ホットAIツール

Undresser.AI Undress
リアルなヌード写真を作成する AI 搭載アプリ

AI Clothes Remover
写真から衣服を削除するオンライン AI ツール。

Undress AI Tool
脱衣画像を無料で

Clothoff.io
AI衣類リムーバー

Video Face Swap
完全無料の AI 顔交換ツールを使用して、あらゆるビデオの顔を簡単に交換できます。

人気の記事

ホットツール

メモ帳++7.3.1
使いやすく無料のコードエディター

SublimeText3 中国語版
中国語版、とても使いやすい

ゼンドスタジオ 13.0.1
強力な PHP 統合開発環境

ドリームウィーバー CS6
ビジュアル Web 開発ツール

SublimeText3 Mac版
神レベルのコード編集ソフト(SublimeText3)

ホットトピック
 1657
1657
 14
14
 1415
1415
 52
52
 1309
1309
 25
25
 1257
1257
 29
29
 1231
1231
 24
24
 ビットコインの価値はいくらですか
Apr 28, 2025 pm 07:42 PM
ビットコインの価値はいくらですか
Apr 28, 2025 pm 07:42 PM
ビットコインの価格は20,000ドルから30,000ドルの範囲です。 1。ビットコインの価格は2009年以来劇的に変動し、2017年には20,000ドル近くに達し、2021年にはほぼ60,000ドルに達しました。2。価格は、市場需要、供給、マクロ経済環境などの要因の影響を受けます。 3.取引所、モバイルアプリ、ウェブサイトを通じてリアルタイム価格を取得します。 4。ビットコインの価格は非常に不安定であり、市場の感情と外部要因によって駆動されます。 5.従来の金融市場と特定の関係を持ち、世界の株式市場、米ドルの強さなどの影響を受けています。6。長期的な傾向は強気ですが、リスクを慎重に評価する必要があります。
 2025年のトップ10の通貨取引プラットフォームのどれがトップ10の通貨取引プラットフォームの1つです
Apr 28, 2025 pm 08:12 PM
2025年のトップ10の通貨取引プラットフォームのどれがトップ10の通貨取引プラットフォームの1つです
Apr 28, 2025 pm 08:12 PM
2025年の世界の上位10の暗号通貨取引所には、Binance、Okx、Gate.io、Coinbase、Kraken、Huobi、Bitfinex、Kucoin、Bittrex、Poloniexが含まれます。これらはすべて、高い取引量とセキュリティで知られています。
 世界のトップ10の通貨取引プラットフォームのどれがトップ10の通貨取引プラットフォームの最新バージョンです
Apr 28, 2025 pm 08:09 PM
世界のトップ10の通貨取引プラットフォームのどれがトップ10の通貨取引プラットフォームの最新バージョンです
Apr 28, 2025 pm 08:09 PM
世界の上位10の暗号通貨取引プラットフォームには、Binance、Okx、Gate.io、Coinbase、Kraken、Huobi Global、Bitfinex、Bittrex、Kucoin、Poloniexが含まれます。これらはすべて、さまざまな取引方法と強力なセキュリティ対策を提供します。
 トップ10の仮想通貨取引アプリは何ですか?最新のデジタル通貨交換ランキング
Apr 28, 2025 pm 08:03 PM
トップ10の仮想通貨取引アプリは何ですか?最新のデジタル通貨交換ランキング
Apr 28, 2025 pm 08:03 PM
Binance、OKX、Gate.ioなどの上位10のデジタル通貨交換は、システムを改善し、効率的な多様化したトランザクション、厳格なセキュリティ対策を改善しました。
 復号化GATE.IO戦略のアップグレード:Memebox 2.0でCrypto Asset Managementを再定義する方法は?
Apr 28, 2025 pm 03:33 PM
復号化GATE.IO戦略のアップグレード:Memebox 2.0でCrypto Asset Managementを再定義する方法は?
Apr 28, 2025 pm 03:33 PM
Memebox 2.0は、革新的なアーキテクチャとパフォーマンスのブレークスルーを通じて、暗号資産管理を再定義します。 1)3つの主要な問題点を解決します。資産サイロ、収入の減少、セキュリティと利便性のパラドックスです。 2)インテリジェントアセットハブ、動的リスク管理およびリターンエンハンスメントエンジン、クロスチェーン移動速度、平均降伏率、およびセキュリティインシデント応答速度が向上します。 3)ユーザーに、ユーザー価値の再構築を実現し、資産の視覚化、ポリシーの自動化、ガバナンス統合を提供します。 4)生態学的なコラボレーションとコンプライアンスの革新により、プラットフォームの全体的な有効性が向上しました。 5)将来的には、スマート契約保険プール、予測市場統合、AI主導の資産配分が開始され、引き続き業界の発展をリードします。
 トップ通貨取引プラットフォームは何ですか?トップ10の最新の仮想通貨交換
Apr 28, 2025 pm 08:06 PM
トップ通貨取引プラットフォームは何ですか?トップ10の最新の仮想通貨交換
Apr 28, 2025 pm 08:06 PM
現在、上位10の仮想通貨交換にランクされています。1。Binance、2。Okx、3。Gate.io、4。CoinLibrary、5。Siren、6。HuobiGlobal Station、7。Bybit、8。Kucoin、9。Bitcoin、10。BitStamp。
 推奨される信頼できるデジタル通貨取引プラットフォーム。世界のトップ10のデジタル通貨交換。 2025
Apr 28, 2025 pm 04:30 PM
推奨される信頼できるデジタル通貨取引プラットフォーム。世界のトップ10のデジタル通貨交換。 2025
Apr 28, 2025 pm 04:30 PM
推奨される信頼できるデジタル通貨取引プラットフォーム:1。OKX、2。Binance、3。Coinbase、4。Kraken、5。Huobi、6。Kucoin、7。Bitfinex、8。Gemini、9。Bitstamp、10。Poloniex、これらのプラットフォームは、セキュリティ、ユーザーエクスペリエンス、ユーザーエクスペリエンス、ユーザーエクスペリエンス、ユーザーエクスペリエンスのデジタルエクスペリエンス、デジタルエクスペリエンスのデジタルエクスペリエンス、デジタルエクスペリエンスのために知られています。
 CでChronoライブラリを使用する方法は?
Apr 28, 2025 pm 10:18 PM
CでChronoライブラリを使用する方法は?
Apr 28, 2025 pm 10:18 PM
CでChronoライブラリを使用すると、時間と時間の間隔をより正確に制御できます。このライブラリの魅力を探りましょう。 CのChronoライブラリは、時間と時間の間隔に対処するための最新の方法を提供する標準ライブラリの一部です。 Time.HとCtimeに苦しんでいるプログラマーにとって、Chronoは間違いなく恩恵です。コードの読みやすさと保守性を向上させるだけでなく、より高い精度と柔軟性も提供します。基本から始めましょう。 Chronoライブラリには、主に次の重要なコンポーネントが含まれています。STD:: Chrono :: System_Clock:現在の時間を取得するために使用されるシステムクロックを表します。 STD :: Chron




