機械学習における 10 の非線形次元削減手法の比較概要
次元削減とは、データ セット内の特徴の数を減らしながら、データの主要な情報を可能な限り保持することを指します。次元削減アルゴリズムは教師なし学習であり、アルゴリズムはラベルなしのデータを通じてトレーニングされます。

次元削減方法には多くの種類がありますが、それらはすべて線形と非線形という 2 つの主要なカテゴリに分類できます。
線形メソッドは、高次元空間から低次元空間にデータを線形に投影します (そのため、線形投影という名前が付けられています)。例には、PCA や LDA が含まれます。
非線形手法は、非線形次元削減を実行する方法であり、元のデータの非線形構造を発見するためによく使用されます。非線形次元削減手法は、元のデータが線形に分離するのが難しい場合に特に重要です。場合によっては、非線形次元削減は多様体学習としても知られており、この方法は高次元データをより効率的に処理でき、データの基礎となる構造を明らかにするのに役立ちます。非線形次元削減を通じて、データ間の関係をより深く理解し、データ内の隠れたパターンやルールを発見し、さらなるデータ分析と応用のための強力なサポートを提供できます。
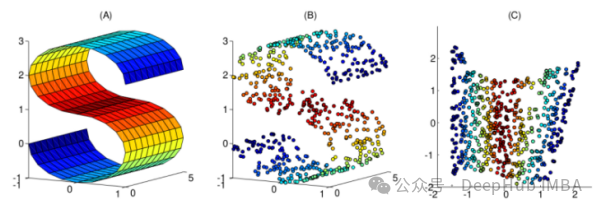
この記事では、日常業務での選択に役立つ、一般的に使用される 10 の非線形次元削減手法をまとめました。
1. カーネル PCA
線形次元削減手法である通常の PCA についてはよくご存じかもしれません。カーネル PCA は、通常の主成分分析の非線形バージョンとみなすことができます。
主成分分析とカーネル主成分分析はどちらも次元削減に使用できますが、線形分離不可能なデータを処理するにはカーネル PCA の方が効果的です。カーネル PCA の主な利点は、データ次元を削減しながら、非線形分離可能なデータを線形分離可能なデータに変換することです。カーネル PCA は、カーネル技術を導入することでデータ内の非線形構造を捕捉できるため、データの分類パフォーマンスが向上します。したがって、カーネル PCA は、複雑なデータセットを扱う場合に、より強力な表現力と一般化能力を備えています。
まず、非常に古典的なデータを作成します。
import matplotlib.pyplot as plt plt.figure(figsize=[7, 5]) from sklearn.datasets import make_moons X, y = make_moons(n_samples=100, noise=None, random_state=0) plt.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='plasma') plt.title('Linearly inseparable data')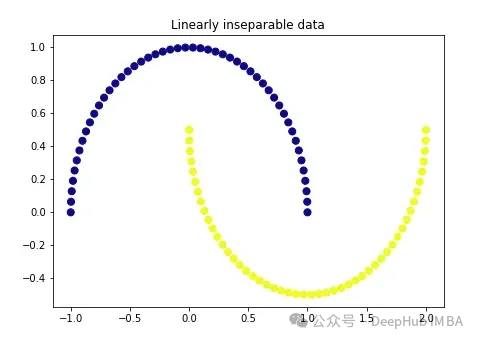
これら 2 つの色は、線形的に分離できない 2 つのカテゴリを表します。ここで直線を引いてこれら 2 つのカテゴリーを分けることは不可能です。
通常の PCA から始めます。
import numpy as np from sklearn.decomposition import PCA pca = PCA(n_components=1) X_pca = pca.fit_transform(X) plt.figure(figsize=[7, 5]) plt.scatter(X_pca[:, 0], np.zeros((100,1)), c=y, s=50, cmap='plasma') plt.title('First component after linear PCA') plt.xlabel('PC1')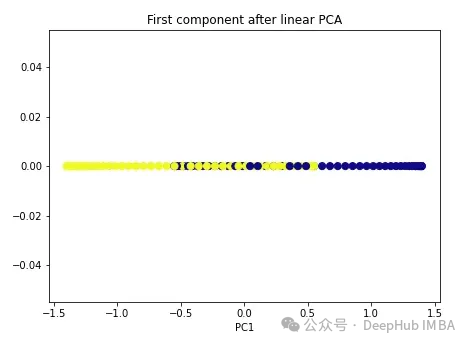
ご覧のとおり、2 つのクラスは依然として線形的に分離できません。次に、カーネル PCA を試してみましょう。
import numpy as np from sklearn.decomposition import KernelPCA kpca = KernelPCA(n_components=1, kernel='rbf', gamma=15) X_kpca = kpca.fit_transform(X) plt.figure(figsize=[7, 5]) plt.scatter(X_kpca[:, 0], np.zeros((100,1)), c=y, s=50, cmap='plasma') plt.axvline(x=0.0, linestyle='dashed', color='black', linewidth=1.2) plt.title('First component after kernel PCA') plt.xlabel('PC1')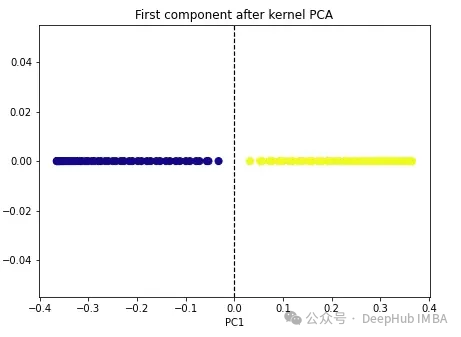
2 つのクラスは線形分離可能になり、カーネル PCA アルゴリズムは異なるカーネルを使用してデータを一方の形式からもう一方の形式に変換します。カーネル PCA は 2 段階のプロセスです。まず、カーネル関数は元のデータを、クラスが線形分離可能な高次元空間に一時的に投影します。次にアルゴリズムは、このデータを n_components ハイパーパラメータで指定されたより低い次元 (保持したい次元の数) に投影し直します。
sklearn には、linear、poly、rbf、sigmoid の 4 つのカーネル オプションがあります。カーネルを「リニア」として指定すると、通常の PCA が実行されます。他のカーネルは非線形 PCA を実行します。 rbf (放射基底関数) カーネルが最も一般的に使用されます。
2. 多次元スケーリング (MDS)
多次元スケーリングは、高次元データ ポイントと低次元データ ポイント間の距離を維持する別の非線形次元削減手法です。たとえば、元の次元でより近い点は、より低い次元のフォームでもより近くに表示されます。
Scikit-learn を使用するには、MDS() クラスを使用できます。
from sklearn.manifold import MDS mds = MDS(n_components, metric) mds_transformed = mds.fit_transform(X)
メトリック ハイパーパラメータは、メトリックと非メトリックという 2 種類の MDS アルゴリズムを区別します。 metric=Trueの場合、メトリックMDSを実行します。それ以外の場合は、非メトリック MDS を実行します。
次の非線形データに 2 種類の MDS アルゴリズムを適用します。
import numpy as np from sklearn.manifold import MDS mds = MDS(n_components=1, metric=True) # Metric MDS X_mds = mds.fit_transform(X) plt.figure(figsize=[7, 5]) plt.scatter(X_mds[:, 0], np.zeros((100,1)), c=y, s=50, cmap='plasma') plt.title('Metric MDS') plt.xlabel('Component 1')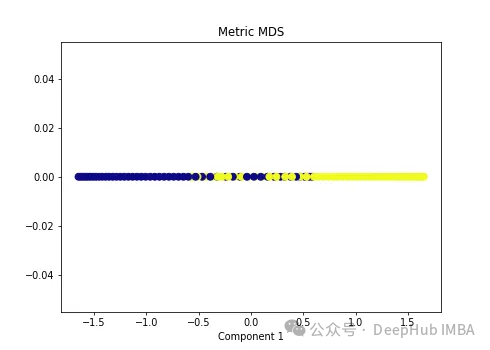
import numpy as np from sklearn.manifold import MDS mds = MDS(n_components=1, metric=False) # Non-metric MDS X_mds = mds.fit_transform(X) plt.figure(figsize=[7, 5]) plt.scatter(X_mds[:, 0], np.zeros((100,1)), c=y, s=50, cmap='plasma') plt.title('Non-metric MDS') plt.xlabel('Component 1')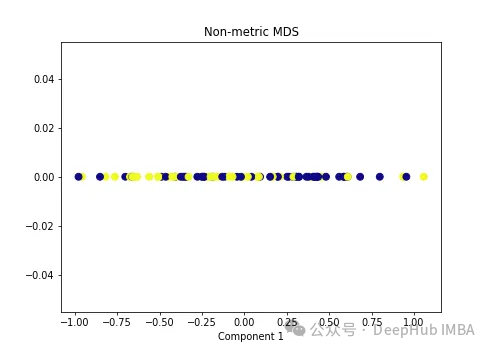
可以看到MDS后都不能使数据线性可分,所以可以说MDS不适合我们这个经典的数据集。
3、Isomap
Isomap(Isometric Mapping)在保持数据点之间的地理距离,即在原始高维空间中的测地线距离或者近似的测地线距离,在低维空间中也被保持。Isomap的基本思想是通过在高维空间中计算数据点之间的测地线距离(通过最短路径算法,比如Dijkstra算法),然后在低维空间中保持这些距离来进行降维。在这个过程中,Isomap利用了流形假设,即假设高维数据分布在一个低维流形上。因此,Isomap通常在处理非线性数据集时表现良好,尤其是当数据集包含曲线和流形结构时。
import matplotlib.pyplot as plt plt.figure(figsize=[7, 5]) from sklearn.datasets import make_moons X, y = make_moons(n_samples=100, noise=None, random_state=0) import numpy as np from sklearn.manifold import Isomap isomap = Isomap(n_neighbors=5, n_components=1) X_isomap = isomap.fit_transform(X) plt.figure(figsize=[7, 5]) plt.scatter(X_isomap[:, 0], np.zeros((100,1)), c=y, s=50, cmap='plasma') plt.title('First component after applying Isomap') plt.xlabel('Component 1')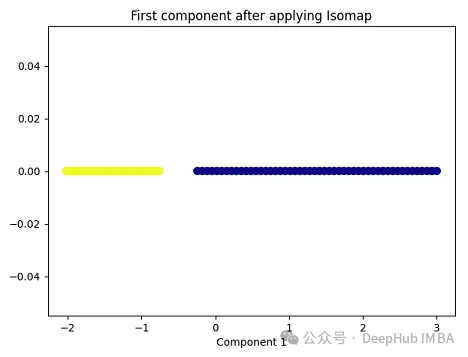
就像核PCA一样,这两个类在应用Isomap后是线性可分的!
4、Locally Linear Embedding(LLE)
与Isomap类似,LLE也是基于流形假设,即假设高维数据分布在一个低维流形上。LLE的主要思想是在局部邻域内保持数据点之间的线性关系,并在低维空间中重构这些关系。
from sklearn.manifold import LocallyLinearEmbedding lle = LocallyLinearEmbedding(n_neighbors=5,n_components=1) lle_transformed = lle.fit_transform(X) plt.figure(figsize=[7, 5]) plt.scatter(lle_transformed[:, 0], np.zeros((100,1)), c=y, s=50, cmap='plasma') plt.title('First component after applying LocallyLinearEmbedding') plt.xlabel('Component 1')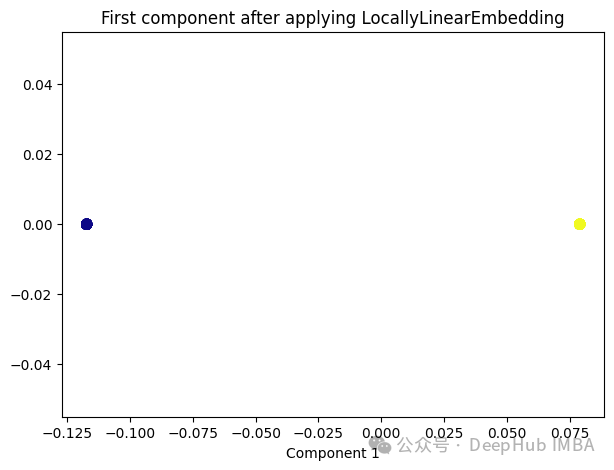
只有2个点,其实并不是这样,我们打印下这个数据
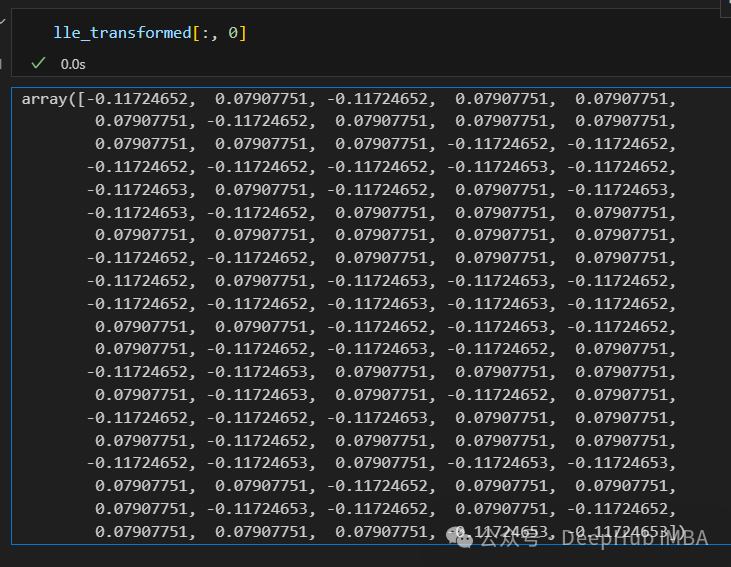
可以看到数据通过降维变成了同一个数字,所以LLE降维后是线性可分的,但是却丢失了数据的信息。
5、Spectral Embedding
Spectral Embedding是一种基于图论和谱理论的降维技术,通常用于将高维数据映射到低维空间。它的核心思想是利用数据的相似性结构,将数据点表示为图的节点,并通过图的谱分解来获取低维表示。
from sklearn.manifold import SpectralEmbedding sp_emb = SpectralEmbedding(n_components=1, affinity='nearest_neighbors') sp_emb_transformed = sp_emb.fit_transform(X) plt.figure(figsize=[7, 5]) plt.scatter(sp_emb_transformed[:, 0], np.zeros((100,1)), c=y, s=50, cmap='plasma') plt.title('First component after applying SpectralEmbedding') plt.xlabel('Component 1')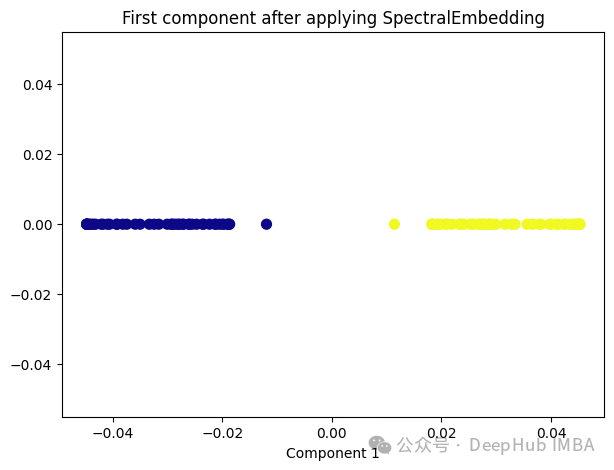
6、t-Distributed Stochastic Neighbor Embedding (t-SNE)
t-SNE的主要目标是保持数据点之间的局部相似性关系,并在低维空间中保持这些关系,同时试图保持全局结构。
from sklearn.manifold import TSNE tsne = TSNE(1, learning_rate='auto', init='pca') tsne_transformed = tsne.fit_transform(X) plt.figure(figsize=[7, 5]) plt.scatter(tsne_transformed[:, 0], np.zeros((100,1)), c=y, s=50, cmap='plasma') plt.title('First component after applying TSNE') plt.xlabel('Component 1')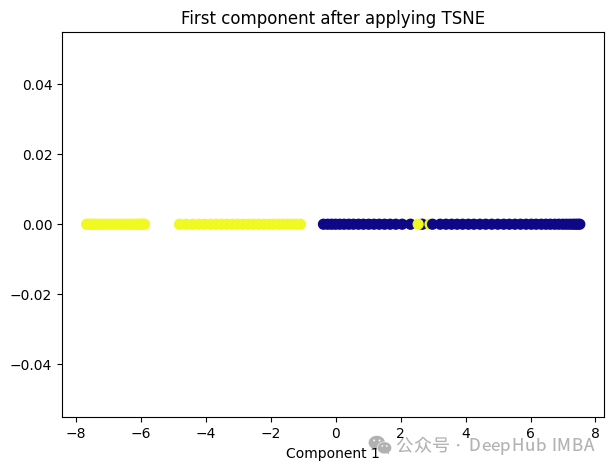
t-SNE好像也不太适合我们的数据。
7、Random Trees Embedding
Random Trees Embedding是一种基于树的降维技术,常用于将高维数据映射到低维空间。它利用了随机森林(Random Forest)的思想,通过构建多棵随机决策树来实现降维。
Random Trees Embedding的基本工作流程:
- 构建随机决策树集合:首先,构建多棵随机决策树。每棵树都是通过从原始数据中随机选择子集进行训练的,这样可以减少过拟合,提高泛化能力。
- 提取特征表示:对于每个数据点,通过将其在每棵树上的叶子节点的索引作为特征,构建一个特征向量。每个叶子节点都代表了数据点在树的某个分支上的位置。
- 降维:通过随机森林中所有树生成的特征向量,将数据点映射到低维空间中。通常使用降维技术,如主成分分析(PCA)或t-SNE等,来实现最终的降维过程。
Random Trees Embedding的优势在于它的计算效率高,特别是对于大规模数据集。由于使用了随机森林的思想,它能够很好地处理高维数据,并且不需要太多的调参过程。
RandomTreesEmbedding使用高维稀疏进行无监督转换,也就是说,我们最终得到的数据并不是一个连续的数值,而是稀疏的表示。所以这里就不进行代码展示了,有兴趣的看看sklearn的sklearn.ensemble.RandomTreesEmbedding
8、Dictionary Learning
Dictionary Learning是一种用于降维和特征提取的技术,它主要用于处理高维数据。它的目标是学习一个字典,该字典由一组原子(或基向量)组成,这些原子是数据的线性组合。通过学习这样的字典,可以将高维数据表示为一个更紧凑的低维空间中的稀疏线性组合。
Dictionary Learning的优点之一是它能够学习出具有可解释性的原子,这些原子可以提供关于数据结构和特征的重要见解。此外,Dictionary Learning还可以产生稀疏表示,从而提供更紧凑的数据表示,有助于降低存储成本和计算复杂度。
from sklearn.decomposition import DictionaryLearning dict_lr = DictionaryLearning(n_components=1) dict_lr_transformed = dict_lr.fit_transform(X) plt.figure(figsize=[7, 5]) plt.scatter(dict_lr_transformed[:, 0], np.zeros((100,1)), c=y, s=50, cmap='plasma') plt.title('First component after applying DictionaryLearning') plt.xlabel('Component 1')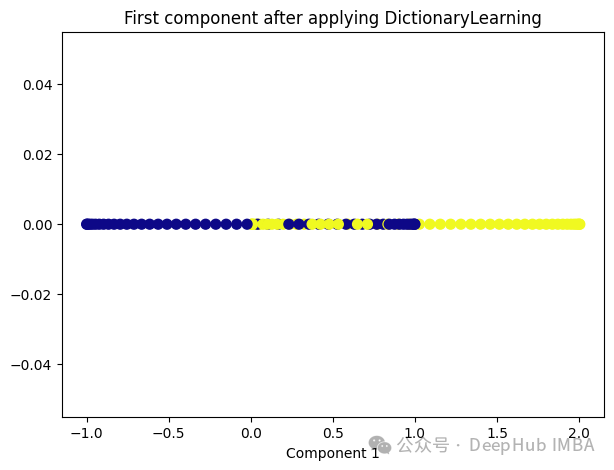
9、Independent Component Analysis (ICA)
Independent Component Analysis (ICA) 是一种用于盲源分离的统计方法,通常用于从混合信号中估计原始信号。在机器学习和信号处理领域,ICA经常用于解决以下问题:
- 盲源分离:给定一组混合信号,其中每个信号是一组原始信号的线性组合,ICA的目标是从混合信号中分离出原始信号,而不需要事先知道混合过程的具体细节。
- 特征提取:ICA可以被用来发现数据中的独立成分,提取数据的潜在结构和特征,通常在降维或预处理过程中使用。
ICA的基本假设是,混合信号中的各个成分是相互独立的,即它们的统计特性是独立的。这与主成分分析(PCA)不同,PCA假设成分之间是正交的,而不是独立的。因此ICA通常比PCA更适用于发现非高斯分布的独立成分。
from sklearn.decomposition import FastICA ica = FastICA(n_components=1, whiten='unit-variance') ica_transformed = dict_lr.fit_transform(X) plt.figure(figsize=[7, 5]) plt.scatter(ica_transformed[:, 0], np.zeros((100,1)), c=y, s=50, cmap='plasma') plt.title('First component after applying FastICA') plt.xlabel('Component 1')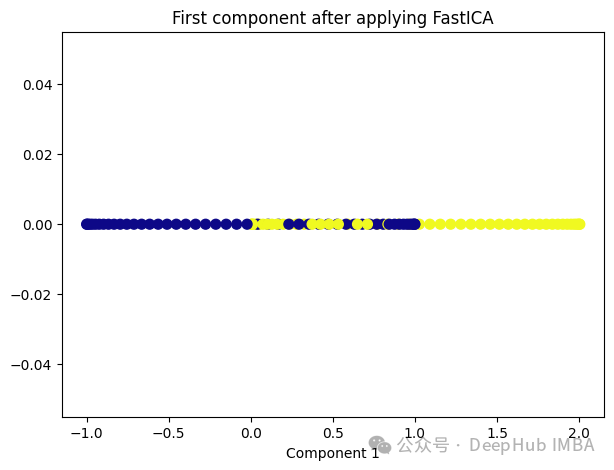
10、Autoencoders (AEs)
到目前为止,我们讨论的NLDR技术属于通用机器学习算法的范畴。而自编码器是一种基于神经网络的NLDR技术,可以很好地处理大型非线性数据。当数据集较小时,自动编码器的效果可能不是很好。
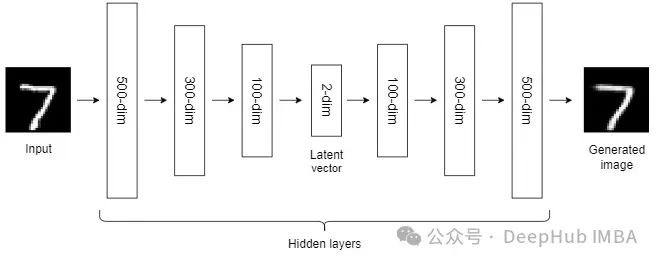
自编码器我们已经介绍过很多次了,所以这里就不详细说明了。
总结
非线性降维技术是一类用于将高维数据映射到低维空间的方法,它们通常适用于数据具有非线性结构的情况。
大多数NLDR方法基于最近邻方法,该方法要求数据中所有特征的尺度相同,所以如果特征的尺度不同,还需要进行缩放。
另外这些非线性降维技术在不同的数据集和任务中可能表现出不同的性能,因此在选择合适的方法时需要考虑数据的特征、降维的目标以及计算资源等因素。
以上が機械学習における 10 の非線形次元削減手法の比較概要の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。

ホットAIツール

Undresser.AI Undress
リアルなヌード写真を作成する AI 搭載アプリ

AI Clothes Remover
写真から衣服を削除するオンライン AI ツール。

Undress AI Tool
脱衣画像を無料で

Clothoff.io
AI衣類リムーバー

Video Face Swap
完全無料の AI 顔交換ツールを使用して、あらゆるビデオの顔を簡単に交換できます。

人気の記事

ホットツール

メモ帳++7.3.1
使いやすく無料のコードエディター

SublimeText3 中国語版
中国語版、とても使いやすい

ゼンドスタジオ 13.0.1
強力な PHP 統合開発環境

ドリームウィーバー CS6
ビジュアル Web 開発ツール

SublimeText3 Mac版
神レベルのコード編集ソフト(SublimeText3)

ホットトピック
 7681
7681
 15
15
 1639
1639
 14
14
 1393
1393
 52
52
 1286
1286
 25
25
 1229
1229
 29
29
 オープンソースの無料画像注釈ツールおすすめ 15 選
Mar 28, 2024 pm 01:21 PM
オープンソースの無料画像注釈ツールおすすめ 15 選
Mar 28, 2024 pm 01:21 PM
画像の注釈は、ラベルまたは説明情報を画像に関連付けて、画像の内容に深い意味と説明を与えるプロセスです。このプロセスは機械学習にとって重要であり、画像内の個々の要素をより正確に識別するために視覚モデルをトレーニングするのに役立ちます。画像に注釈を追加することで、コンピュータは画像の背後にあるセマンティクスとコンテキストを理解できるため、画像の内容を理解して分析する能力が向上します。画像アノテーションは、コンピュータ ビジョン、自然言語処理、グラフ ビジョン モデルなどの多くの分野をカバーする幅広い用途があり、車両が道路上の障害物を識別するのを支援したり、障害物の検出を支援したりするなど、幅広い用途があります。医用画像認識による病気の診断。この記事では主に、より優れたオープンソースおよび無料の画像注釈ツールをいくつか推奨します。 1.マケセンス
 この記事では、SHAP: 機械学習のモデルの説明について説明します。
Jun 01, 2024 am 10:58 AM
この記事では、SHAP: 機械学習のモデルの説明について説明します。
Jun 01, 2024 am 10:58 AM
機械学習とデータ サイエンスの分野では、モデルの解釈可能性が常に研究者や実務家に焦点を当ててきました。深層学習やアンサンブル手法などの複雑なモデルが広く適用されるようになったことで、モデルの意思決定プロセスを理解することが特に重要になってきました。 Explainable AI|XAI は、モデルの透明性を高めることで、機械学習モデルに対する信頼と自信を構築するのに役立ちます。モデルの透明性の向上は、複数の複雑なモデルの普及や、モデルを説明するための意思決定プロセスなどの方法によって実現できます。これらの方法には、特徴重要度分析、モデル予測間隔推定、ローカル解釈可能性アルゴリズムなどが含まれます。特徴重要度分析では、入力特徴に対するモデルの影響度を評価することで、モデルの意思決定プロセスを説明できます。モデルの予測間隔の推定
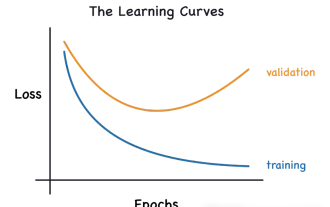 学習曲線を通じて過学習と過小学習を特定する
Apr 29, 2024 pm 06:50 PM
学習曲線を通じて過学習と過小学習を特定する
Apr 29, 2024 pm 06:50 PM
この記事では、学習曲線を通じて機械学習モデルの過学習と過小学習を効果的に特定する方法を紹介します。過小適合と過適合 1. 過適合 モデルがデータからノイズを学習するためにデータ上で過学習されている場合、そのモデルは過適合していると言われます。過学習モデルはすべての例を完璧に学習するため、未確認の新しい例を誤って分類してしまいます。過適合モデルの場合、完璧/ほぼ完璧なトレーニング セット スコアとひどい検証セット/テスト スコアが得られます。若干修正: 「過学習の原因: 複雑なモデルを使用して単純な問題を解決し、データからノイズを抽出します。トレーニング セットとしての小さなデータ セットはすべてのデータを正しく表現できない可能性があるため、2. 過学習の Heru。」
 透明!主要な機械学習モデルの原理を徹底的に分析!
Apr 12, 2024 pm 05:55 PM
透明!主要な機械学習モデルの原理を徹底的に分析!
Apr 12, 2024 pm 05:55 PM
平たく言えば、機械学習モデルは、入力データを予測された出力にマッピングする数学関数です。より具体的には、機械学習モデルは、予測出力と真のラベルの間の誤差を最小限に抑えるために、トレーニング データから学習することによってモデル パラメーターを調整する数学関数です。機械学習には、ロジスティック回帰モデル、デシジョン ツリー モデル、サポート ベクター マシン モデルなど、多くのモデルがあります。各モデルには、適用可能なデータ タイプと問題タイプがあります。同時に、異なるモデル間には多くの共通点があったり、モデル進化の隠れた道が存在したりすることがあります。コネクショニストのパーセプトロンを例にとると、パーセプトロンの隠れ層の数を増やすことで、それをディープ ニューラル ネットワークに変換できます。パーセプトロンにカーネル関数を追加すると、SVM に変換できます。これです
 宇宙探査と人類居住工学における人工知能の進化
Apr 29, 2024 pm 03:25 PM
宇宙探査と人類居住工学における人工知能の進化
Apr 29, 2024 pm 03:25 PM
1950 年代に人工知能 (AI) が誕生しました。そのとき、研究者たちは、機械が思考などの人間と同じようなタスクを実行できることを発見しました。その後、1960 年代に米国国防総省は人工知能に資金を提供し、さらなる開発のために研究所を設立しました。研究者たちは、宇宙探査や極限環境での生存など、多くの分野で人工知能の応用を見出しています。宇宙探査は、地球を超えた宇宙全体を対象とする宇宙の研究です。宇宙は地球とは条件が異なるため、極限環境に分類されます。宇宙で生き残るためには、多くの要素を考慮し、予防策を講じる必要があります。科学者や研究者は、宇宙を探索し、あらゆるものの現状を理解することが、宇宙の仕組みを理解し、潜在的な環境危機に備えるのに役立つと信じています。
 C++ での機械学習アルゴリズムの実装: 一般的な課題と解決策
Jun 03, 2024 pm 01:25 PM
C++ での機械学習アルゴリズムの実装: 一般的な課題と解決策
Jun 03, 2024 pm 01:25 PM
C++ の機械学習アルゴリズムが直面する一般的な課題には、メモリ管理、マルチスレッド、パフォーマンスの最適化、保守性などがあります。解決策には、スマート ポインター、最新のスレッド ライブラリ、SIMD 命令、サードパーティ ライブラリの使用、コーディング スタイル ガイドラインの遵守、自動化ツールの使用が含まれます。実践的な事例では、Eigen ライブラリを使用して線形回帰アルゴリズムを実装し、メモリを効果的に管理し、高性能の行列演算を使用する方法を示します。
 あなたが知らない機械学習の 5 つの流派
Jun 05, 2024 pm 08:51 PM
あなたが知らない機械学習の 5 つの流派
Jun 05, 2024 pm 08:51 PM
機械学習は人工知能の重要な分野であり、明示的にプログラムしなくてもコンピューターにデータから学習して能力を向上させる機能を提供します。機械学習は、画像認識や自然言語処理から、レコメンデーションシステムや不正行為検出に至るまで、さまざまな分野で幅広く応用されており、私たちの生活様式を変えつつあります。機械学習の分野にはさまざまな手法や理論があり、その中で最も影響力のある 5 つの手法は「機械学習の 5 つの流派」と呼ばれています。 5 つの主要な学派は、象徴学派、コネクショニスト学派、進化学派、ベイジアン学派、およびアナロジー学派です。 1. 象徴主義は、象徴主義とも呼ばれ、論理的推論と知識の表現のためのシンボルの使用を強調します。この学派は、学習は既存の既存の要素を介した逆演繹のプロセスであると信じています。
 説明可能な AI: 複雑な AI/ML モデルの説明
Jun 03, 2024 pm 10:08 PM
説明可能な AI: 複雑な AI/ML モデルの説明
Jun 03, 2024 pm 10:08 PM
翻訳者 | Li Rui によるレビュー | 今日、人工知能 (AI) および機械学習 (ML) モデルはますます複雑になっており、これらのモデルによって生成される出力はブラックボックスになっており、関係者に説明することができません。 Explainable AI (XAI) は、利害関係者がこれらのモデルがどのように機能するかを理解できるようにし、これらのモデルが実際に意思決定を行う方法を確実に理解できるようにし、AI システムの透明性、信頼性、およびこの問題を解決するための説明責任を確保することで、この問題を解決することを目指しています。この記事では、さまざまな説明可能な人工知能 (XAI) 手法を検討して、その基礎となる原理を説明します。説明可能な AI が重要であるいくつかの理由 信頼と透明性: AI システムが広く受け入れられ、信頼されるためには、ユーザーは意思決定がどのように行われるかを理解する必要があります




