Editor | Violet
단백질, 약물 및 기타 생체분자 간의 상호작용은 다양한 생물학적 과정에서 중요한 역할을 합니다. 이러한 상호 작용을 이해하는 것은 생물학적 과정의 기초가 되는 분자 메커니즘을 해독하고 새로운 치료 전략을 개발하는 데 중요합니다. 단백질은 세포에서 가장 중요한 분자 중 하나이며 세포 내에서 다양한 기능을 수행합니다. 약물은 종종 특정 단백질과 상호작용하여 생리학적 과정을 조절합니다. 이러한 상호 작용은 특정 분자 신호 전달 경로를 촉진하거나 억제할 수 있습니다. 현재의 다중 규모 계산 방법은 종종 단일 규모에 너무 많이 의존하고 다른 규모에 적합하지 않습니다. 이는 고르지 못한 다중 규모 방향성 및 다중 규모 학습에 내재된 탐욕과 관련이 있을 수 있습니다.
최적화 불균형을 완화하기 위해 Sun Yat-sen University와 Shanghai Jiao Tong University의 연구자들은 학습을 위해 다중 규모 정보를 효과적으로 통합할 수 있는 변수 기대 최대화 기반 다중 규모 표현 학습 프레임워크 MUSE를 제안했습니다. 이 전략은 상호 감독 및 반복 최적화를 통해 원자 구조와 분자 네트워크 규모 간의 다중 규모 정보를 효과적으로 융합합니다. 이 접근 방식을 사용하면 더 많은 정보 전달 및 학습이 가능합니다. 이 전략은 상호 감독 및 반복 최적화를 통해 원자 구조와 분자 네트워크 규모 간의 다중 규모 정보를 효과적으로 융합합니다.
MUSE+는 분자 상호 작용(단백질-단백질, 약물-단백질 및 약물) 작업에 대한 현재 최첨단 모델보다 성능이 뛰어날 뿐만 아니라 단백질 인터페이스 예측에 대한 현재 최첨단 모델보다 성능이 뛰어납니다. 원자 구조 규모에서. 더 중요한 것은 다중 규모 학습 프레임워크가 다른 규모의 컴퓨팅 약물 발견으로 확장될 수 있다는 것입니다.
"
단백질과 약물 상호작용의 균형 잡힌 다중 규모 학습을 위한 변이적 기대-최대화 프레임워크"라는 제목의 이번 연구는 "Nature Communications"에 5월 25일 게재되었습니다.
 논문 링크:
논문 링크:
순전히 구조를 통해 이러한 상호 작용을 예측하는 것은 구조 생물학에서 가장 중요한 과제 중 하나입니다. 현재의 계산 방법은 대부분 분자 네트워크나 구조 정보를 기반으로 상호 작용을 예측하며 이를 통합된 다중 규모 프레임워크에 통합하지 않습니다.
일부 다중 뷰 학습 방법은 다중 규모 정보를 융합하려고 노력하지만 다중 규모 표현을 학습하는 직관적인 방법은 분자 그래프를 상호 작용 네트워크와 결합하여 공동으로 최적화하는 것입니다. 그러나 다중 규모 학습의 불균형과 본질적인 탐욕적 특성으로 인해 이러한 모델은 단일 규모에 크게 의존하는 경우가 많습니다. 모든 규모와 일반화에 관련된 정보를 효과적으로 활용하지 못하는 것은 좋지 않습니다.
또한 효과적인 다중 규모 프레임워크는 다양한 규모 내에서 풍부한 정보를 캡처해야 할 뿐만 아니라 이들 간의 기본 관계를 잘 보존해야 합니다.
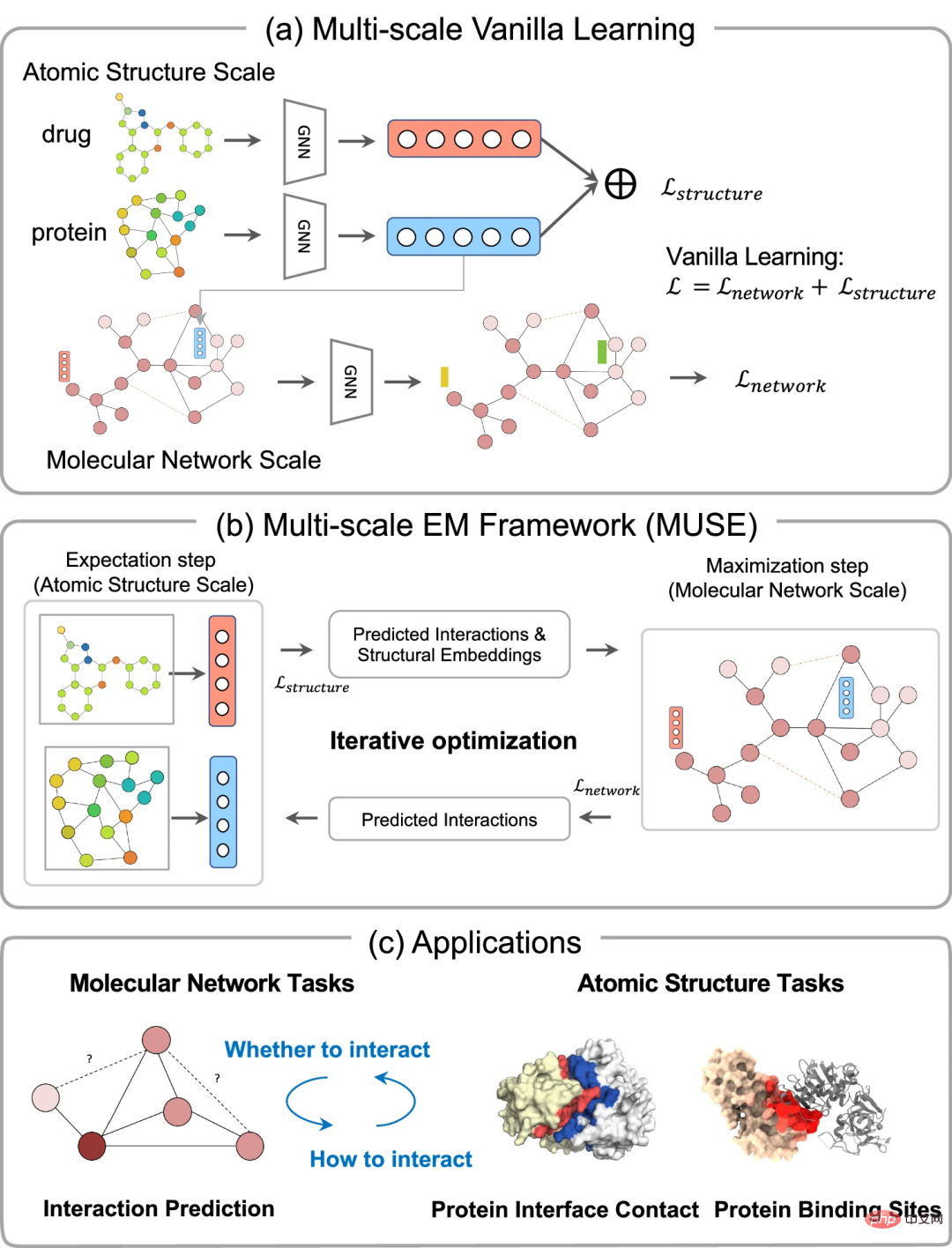
단백질과 약물의 다단계 정보 학습을 위한 MUSEMUSE는 변형 기대 최대화(EM) 프레임워크를 통해 분자 구조 모델링과 단백질-약물 상호 작용 네트워크 학습을 결합한 다중 규모 학습 방법입니다. EM 프레임워크는 여러 반복의 교번 프로세스에서 기대 단계(E-step)와 최대화 단계(M-step)라는 두 모듈을 최적화합니다.
E-단계 동안 MUSE는 각 생체 분자의 구조 정보를 활용하여 알려진 상호 작용 및 강화 샘플을 사용하여 M-단계 훈련을 위한 효과적인 구조적 표현을 학습합니다. 이는 입력 단백질과 약물 쌍 및 원자 수준의 구조 정보로 M 단계 예측 상호 작용에 의해 강화됩니다. M-step은 분자 수준 상호 작용 네트워크, 구조적 임베딩 및 E-step의 예측된 상호 작용을 입력으로 사용하여 예측된 상호 작용을 출력합니다. E-step과 M-step 간의 반복적 최적화는 두 가지 규모에서 서로 다른 학습 속도로 분자 구조와 네트워크 정보의 대화형 캡처를 보장합니다.
상호 감독을 통해 각 규모 모델이 적절한 방식으로 학습할 수 있으므로 다양한 규모에서 효과적인 정보를 활용할 수 있습니다. 이 프레임워크는 단백질과 약물 간의 다양한 규모의 상호작용을 통해 입증될 것입니다. MUSE는 다중 규모 학습의 불균형 특성을 완화하고 다양한 규모의 계층적, 보완적 정보를 효과적으로 통합하는 것으로 분석됩니다.
원자 구조 정보를 활용하여 분자 네트워크 규모에서 예측을 개선합니다.
접근 방식을 평가하려면 먼저 연구자들은 MUSE를 사용하여 원자 구조 정보를 통합하여 분자 네트워크 규모 예측을 개선했습니다. MUSE는 단백질-단백질 상호작용(PPI), 약물-단백질 상호작용(DPI) 및 약물-약물 상호작용(DDI)의 세 가지 다중 규모 상호작용 예측 작업에서 최첨단 성능을 달성합니다.
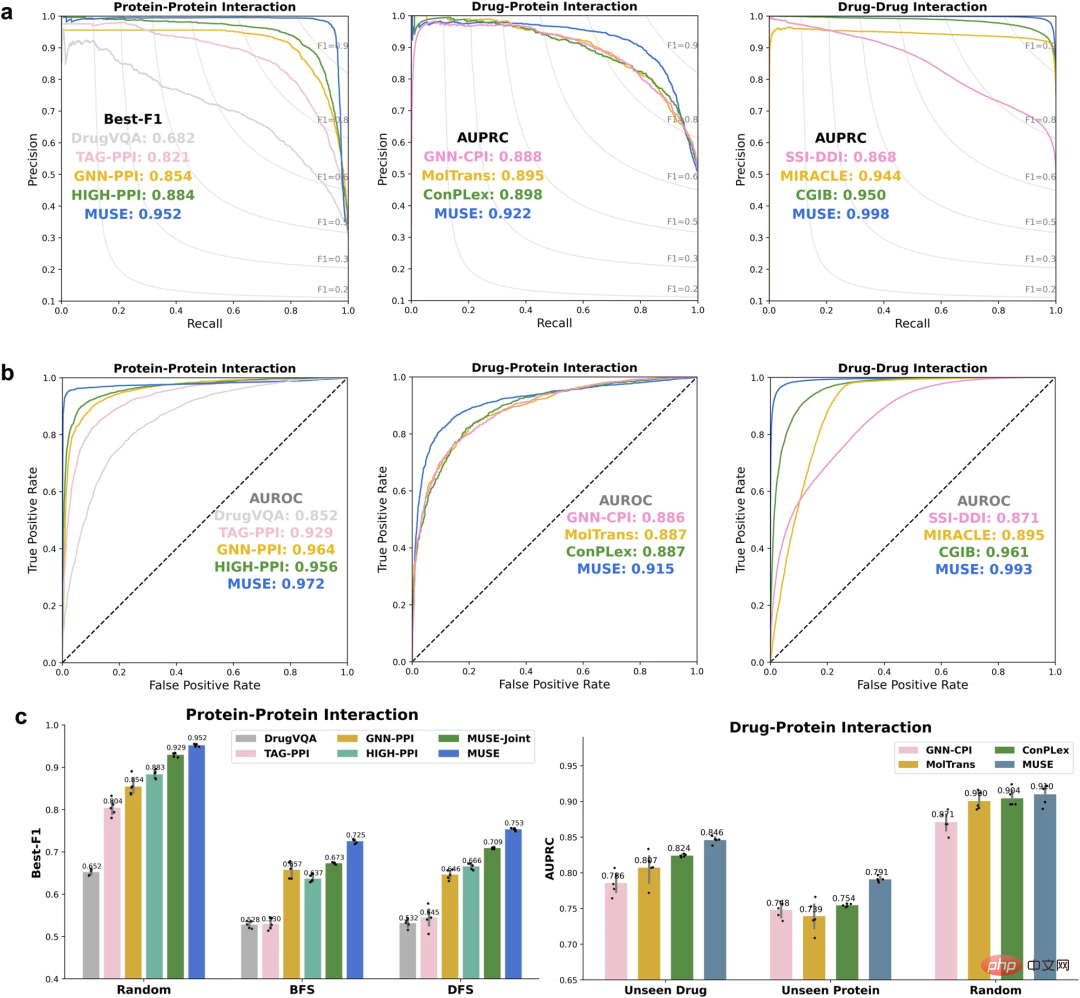
분자 네트워크 규모에서 원자 구조 규모 예측 개선
원자 구조 정보를 사용하여 분자 네트워크 규모 예측을 개선하는 것 외에도 연구원들은 원자 구조에서 구조를 학습하고 예측하는 MUSE의 능력을 추가로 조사했습니다. 규모 PPI와 관련된 계면 접촉 및 결합 부위 예측을 포함하여 특성화하는 능력입니다.
단백질 간 접촉 예측을 평가하기 위해 MUSE를 DIPS-Plus 벤치마크에서 최첨단 방법과 비교했습니다. MUSE는 다른 모든 방법보다 지속적으로 뛰어난 성능을 발휘하여 원자 구조 예측의 효율성과 적응성을 검증합니다.
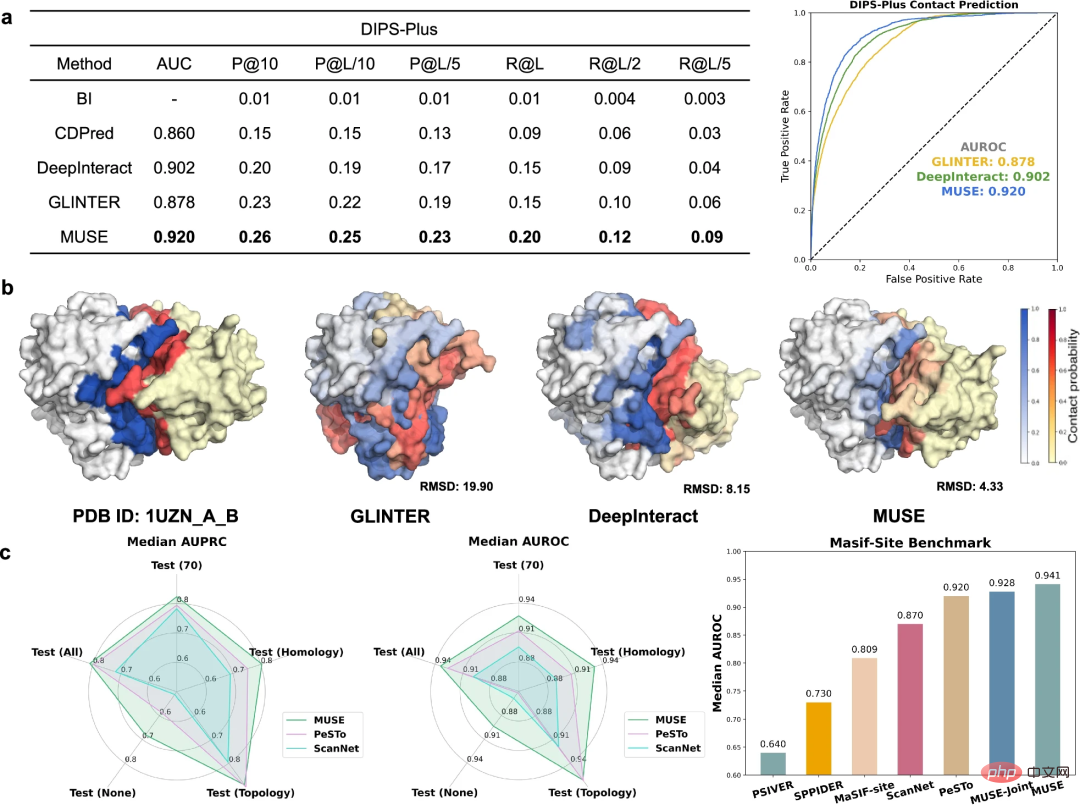
MUSE는 잔류물이 단백질-단백질 상호작용에 직접적으로 관여하는지 여부를 예측하기 위해 추가로 평가되었습니다. 결과는 MUSE의 분자 네트워크 규모 학습이 원자 구조 규모 예측에 대한 귀중한 통찰력을 제공할 수 있음을 보여줍니다.
반복 최적화를 통해 다중 규모 학습의 불균형 특성 완화
MUSE가 다중 규모 표현에서 우수한 성능을 달성할 수 있는 이유를 알아보기 위해 연구자들은 다중 규모 학습의 불균형 특성을 기반으로 MUSE의 학습 능력을 분석했습니다. 학습.
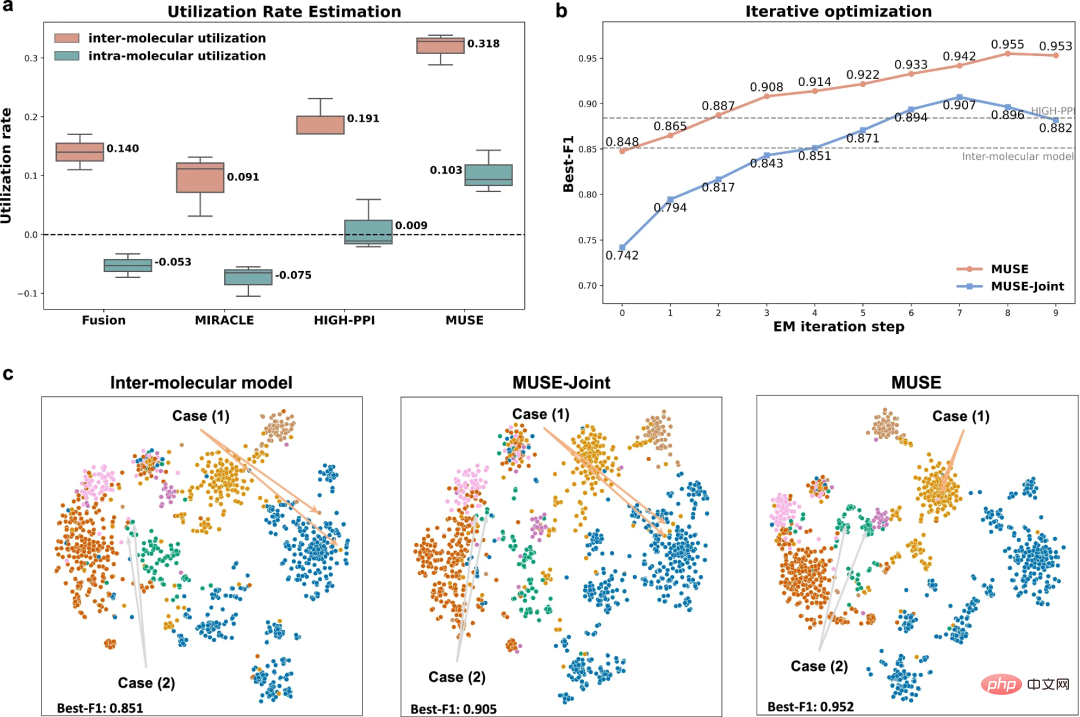
결과에 따르면 MUSE는 다중 규모 학습의 불균형 특성과 욕심 많은 학습을 효과적으로 완화하여 훈련 과정에서 다양한 규모의 정보를 포괄적으로 활용할 수 있음을 보여줍니다. 또한 활용률 분석 실험을 통해 연구자들은 모델이 학습한 내용을 구체적으로 이해할 수 있었고 MUSE를 사용하여 다양한 규모에서 모델 학습의 균형을 유지하면 일반화 기능을 향상시킬 수 있음을 입증했습니다.
학습된 다중 스케일 표현의 시각화 및 해석
학습된 다중 스케일 표현을 더 잘 이해하기 위해 연구자들은 MUSE가 학습한 다중 스케일 표현을 다음과 같은 다양한 각도에서 연구했습니다. (1) MUSE 캡처 원자의 능력 PPI와 관련된 구조 정보(즉, 구조적 모티프 및 임베딩), (2) 학습된 원자 구조와 분자 네트워크 표현 간의 상호 감독.
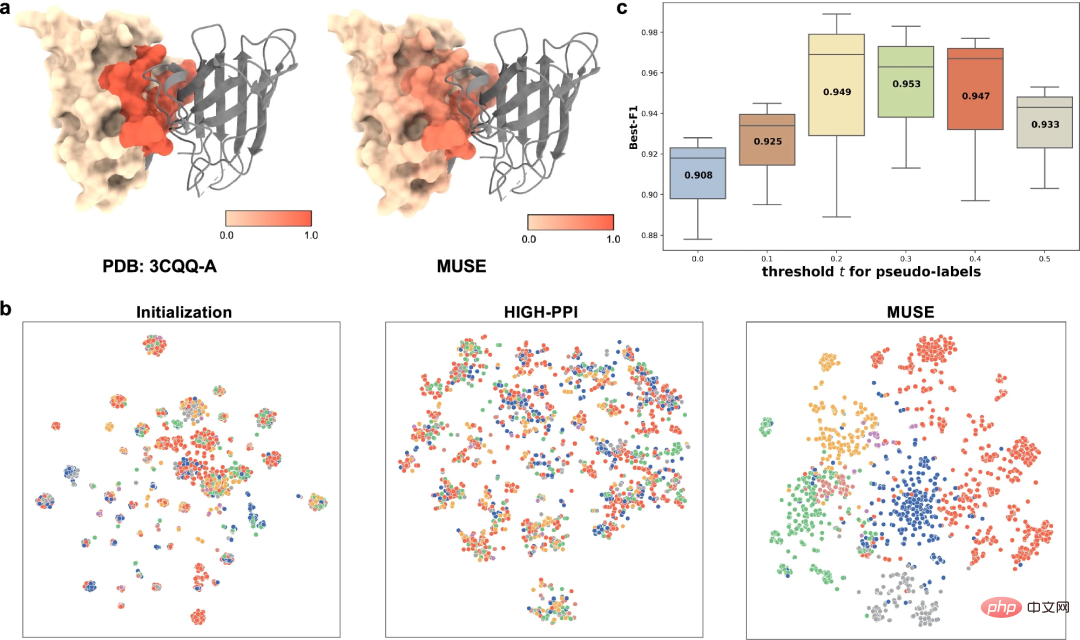
결합 부위 예측(PDB id: 3CQQ-A)의 예로 MUSE는 97.7%의 정확도로 결합 부위에 속하는 잔류물을 정확하게 식별할 수 있습니다. 이는 MUSE의 상호 감독이 원자 구조 규모 모델이 상호 작용과 관련된 주요 하위 구조를 학습하는 데 도움이 된다는 것을 의미합니다.
마지막으로 연구원들은 원자 구조 규모에서 예측된 유사 라벨이 분자 네트워크 규모에 미치는 영향을 연구하기 위해 절제 연구도 수행했습니다.
MUSE는 벤치마크에서 최첨단 성능을 보여주지만 시끄럽고 불완전한 다중 규모 다운스트림 작업을 처리하는 능력을 향상시키는 것은 여전히 가능합니다. 이는 지식 그래프와 설명 가능한 AI 기술을 통해 사전 지식을 결합함으로써 달성될 수 있습니다. 한편, 이 개념적 다중 규모 프레임워크는 다른 규모의 전산 약물 발견으로 확장될 수도 있습니다.
위 내용은 SOTA 성능, 다중 규모 학습, Sun Yat-sen University에서 단백질-약물 상호 작용 AI 프레임워크 제안의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!