
칭화대학교 AIR, 북경대학교, 난징대학교 연구팀이 ESM-AA 모델을 제안했습니다. 이 모델은 단백질 언어 모델링 분야에서 중요한 진전을 이루었으며, 다중 규모 정보를 통합하는 통합 모델링 솔루션을 제공합니다.
아미노산 정보와 원자 정보를 모두 처리할 수 있는 최초의 단백질 사전 학습 언어 모델입니다. 모델의 뛰어난 성능은 기존 한계를 극복하고 새로운 기능을 잠금 해제할 수 있는 다중 규모 통합 모델링의 큰 잠재력을 보여줍니다.
기본 모델인 ESM-AA는 많은 학자들의 관심과 폭넓은 논의를 받아왔으며(아래 스크린샷 참조), AlphaFold3 및 RoseTTAFold All-Atom과 경쟁할 수 있는 ESM-AA 기반 모델 개발 가능성이 있는 것으로 간주됩니다. 이는 다양한 생물학적 구조 간의 상호작용을 연구하는 새로운 길을 열어줍니다. 현재 논문은 ICML 2024에 승인되었습니다.
단백질은 다양한 생활 활동의 핵심 실행자입니다. 단백질과 다른 생물학적 구조와의 상호 작용에 대한 심층적인 이해는 생물학의 핵심 문제이며, 이는 표적 약물 스크리닝, 효소 공학 및 기타 분야에서 실질적으로 중요한 의미를 갖습니다.
따라서 단백질을 더 잘 이해하고 모델링하는 방법은 AI4Science 분야의 연구 핫스팟이 되었습니다.
최근 딥마인드, 워싱턴대학교 베이커그룹 등 주요 첨단 연구기관에서도 단백질 전원자 모델링 문제에 대해 심층적인 연구를 진행하고 있으며, AlphaFold 3, RoseTTAFold 등 방법론을 제안하고 있습니다. 단백질 및 기타 생명 활동에 대한 All-Atom 등 관련 분자의 전원자 규모 모델링 모델은 단백질 구조, 분자 구조, 수용체-리간드 구조 및 기타 전원자 규모를 높은 정확도로 정확하게 예측할 수 있습니다.
이러한 모델은 전원자 규모의 구조 모델링에서 상당한 진전을 이루었지만 현재의 주류 단백질 언어 모델은 여전히 전원자 규모에서 단백질 이해 및 표현 학습을 달성하지 못합니다.
ESM-2로 대표되는 단백질 표현 학습 모델은 아미노산을 유일한 스케일로 사용하여 모델을 구축하는 상황에 적합합니다. 단백질을 가공하는 것은 합리적인 접근 방식입니다.
그러나 단백질의 본질을 완전히 이해하는 열쇠는 작은 분자, DNA, RNA 등과 같은 다른 생물학적 구조와의 상호 작용을 해명하는 데 있습니다.
이러한 요구에 직면하여 서로 다른 구조 간의 복잡한 상호 작용을 설명해야 하며 단일 규모 모델링 전략으로는 효과적이고 포괄적인 적용 범위를 제공하기가 어렵습니다.
이러한 단점을 극복하기 위해 단백질 모델은 다중 규모 모델로의 획기적인 혁신을 겪고 있습니다. 일례로 지난 5월 초 사이언스 매거진에 게재된 RoseTTAFold All-Atom 모델은 RoseTTAFold의 후속 제품으로 멀티 스케일 개념을 도입했습니다.
이 모델은 단백질 구조 예측에만 국한되지 않고 단백질 및 분자/핵산 도킹, 단백질 번역 후 변형 등과 같은 더 넓은 연구 분야로 확장됩니다.
동시에 DeepMind가 새로 출시한 AlphaFold3는 다중 단백질 복합체의 구조 예측을 지원하기 위해 다중 규모 모델링 전략을 채택했으며 그 성능은 인상적이며 의심할 여지 없이 인공 지능 및 생물학 분야에 큰 영향을 미칠 것입니다. .
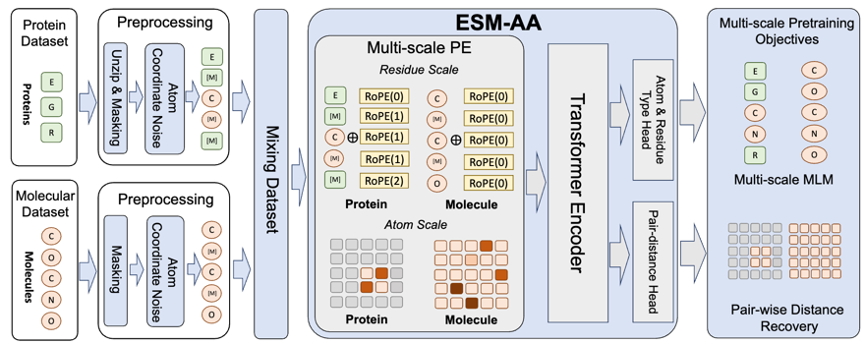
RoseTTAFold All-Atom과 AlphaFold3을 멀티 스케일에 성공적으로 적용 scale 개념에서 영감을 얻었습니다. 중요한 생각은 단백질 기본 모델인 단백질 언어 모델이 어떻게 다중 규모 기술을 채택해야 하는지입니다. 이를 기반으로 연구팀은 다중 규모 단백질 언어 모델 ESM All-Atom(ESM-AA)을 제안했다.
간단히 ESM-AA는 일부 아미노산을 해당 원자 구성에 "압축 해제"하여 다중 규모 개념을 도입합니다. 그 후, 단백질 데이터와 분자 데이터를 혼합하여 사전 훈련을 수행하여 모델이 다양한 규모의 생물학적 구조를 동시에 처리할 수 있는 능력을 부여했습니다.
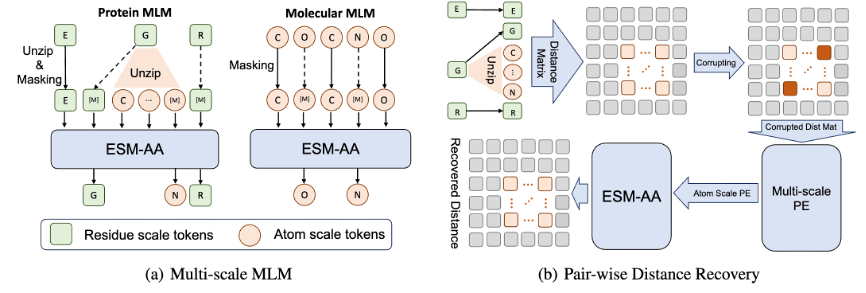
또한 모델이 고품질 원자 수준 정보를 더 잘 학습할 수 있도록 ESM-AA에서는 원자 수준의 분자 구조 데이터도 학습에 사용합니다. 또한 그림 2에 표시된 다중 스케일 위치 인코딩 메커니즘을 도입함으로써 ESM-AA 모델은 다양한 스케일의 정보를 잘 구분할 수 있으므로 모델이 잔류물 수준과 원자 수준에서 위치 및 구조 정보를 정확하게 이해할 수 있습니다.
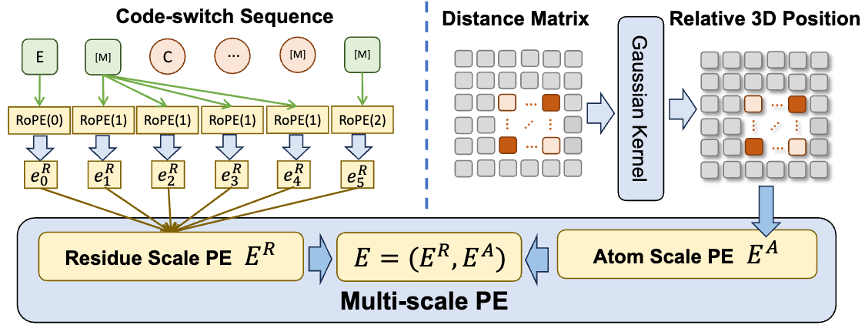
모델이 다중 규모 정보를 학습할 수 있도록 팀에서는 ESM-AA 모델에 대한 다양한 사전 학습 목표를 설계했습니다. ESM-AA의 다중 규모 사전 훈련 목표에는 MLM(Masked Language Modeling) 및 PDR(Pairwise Distance Recovery)이 포함됩니다. 그림 3(a)에 표시된 것처럼 MLM에서는 아미노산과 원자를 마스킹하여 주변 상황을 기반으로 예측을 수행해야 합니다. 이 훈련 작업은 아미노산 및 원자 규모 모두에서 수행될 수 있습니다. PDR은 모델이 원자 수준의 구조 정보를 이해하도록 훈련하기 위해 모델이 서로 다른 원자 사이의 유클리드 거리를 정확하게 예측해야 합니다(그림 3(b) 참조).
ESM-AA 모델은 효소를 포함한 여러 단백질-소분자 벤치마크 작업에서 미세 조정 및 평가되었습니다. -기질 물질 친화성 회귀 작업(결과는 그림 4에 표시됨), 효소-기질 쌍 분류 작업(결과는 그림 4에 표시됨) 및 약물-표적 친화성 회귀 작업(결과는 그림 5에 표시됨).
결과는 ESM-AA가 이러한 작업에서 이전 모델보다 성능이 뛰어나다는 것을 보여 주며, 이는 아미노산 및 원자 규모에서 단백질 사전 훈련된 언어 모델의 잠재력을 완전히 실현했음을 나타냅니다.
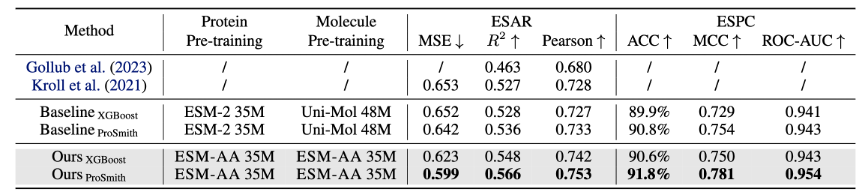
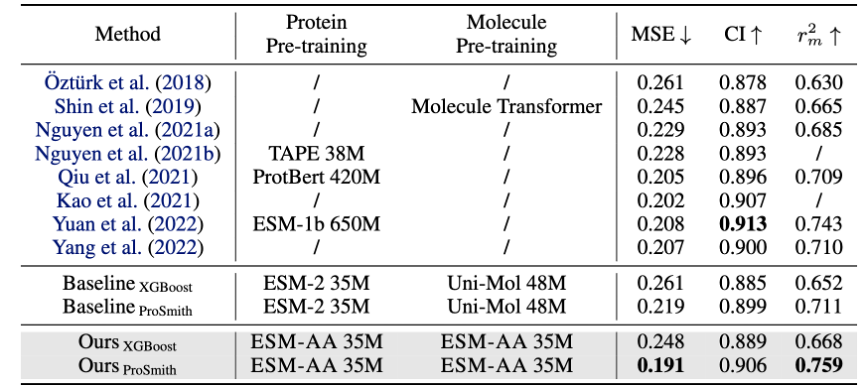
그림 5: 약물-표적 친화성 회귀 작업의 성능 비교
또한 ESM-AA 모델 또한 단백질 접촉 예측, 단백질 기능 분류, 분자 특성 예측 등의 작업에 대한 성능을 테스트했습니다.
결과에 따르면 단백질만 관련된 작업을 처리할 때 ESM-AA는 분자 작업에서 ESM-2와 동등한 성능을 발휘하며 대부분의 벤치마크 모델보다 성능이 뛰어나며 Uni-Mol과 유사합니다.
이것은 ESM-AA가 강력한 분자 지식을 획득하는 과정에서 단백질을 이해하는 능력을 희생하지 않는다는 것을 보여줍니다. 또한 ESM-AA 모델이 처음부터 시작하지 않고도 ESM-2 모델의 지식을 성공적으로 재사용한다는 것을 보여줍니다. 스크래치 개발을 통해 모델 학습 비용을 대폭 절감합니다.
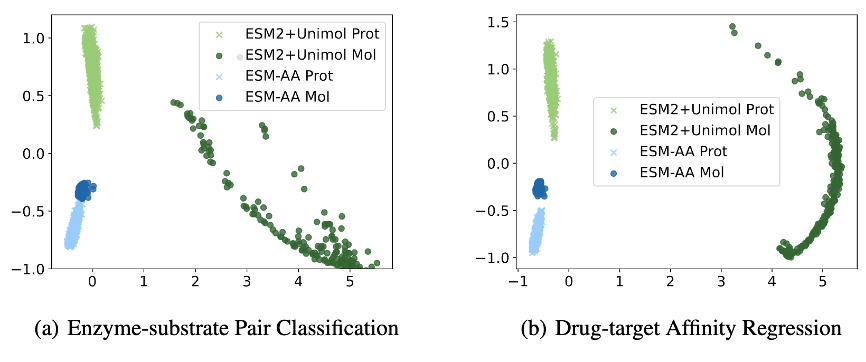
ESM-AA가 단백질-소분자 벤치마크 작업에서 좋은 성능을 보이는 이유를 추가로 분석하기 위해 본 논문에서는 ESM-AA 모델과 ESM-2+Uni-Mol의 추출을 보여줍니다. 이 작업의 모델 조합 표본 표현 분포의 시각화.
그림 6에서 볼 수 있듯이 ESM-AA 모델에서 학습한 단백질 및 소분자 표현은 더 컴팩트하며 이는 두 가지가 동일한 표현 공간에 있음을 나타냅니다. 이것이 ESM-AA 모델이 ESM보다 나은 이유입니다. -2+Uni-Mol 모델은 다중 규모 통합 분자 모델링의 장점을 더욱 잘 보여줍니다.
결론
Tsinghua AIR 팀이 개발한 ESM-AA는 아미노산과 원자 정보 처리를 통합한 최초의 단백질 사전 학습 언어 모델입니다. 이 모델은 다중 규모 정보를 통합하여 강력하고 우수한 성능을 보여주며 생물학적 구조 간의 상호 작용 문제를 해결하는 새로운 방법을 제공합니다.
ESM-AA는 단백질에 대한 더 깊은 이해를 촉진할 뿐만 아니라 여러 생체분자 작업에서도 우수한 성능을 발휘하여 단백질 이해 기능을 유지하면서 분자 수준의 지식을 효과적으로 통합할 수 있음을 입증하여 모델 훈련 비용을 절감합니다. AI를 활용한 생물학 연구의 새로운 방향.
논문 제목: ESM All-Atom: 통합 분자 모델링을 위한 다중 규모 단백질 언어 모델
위 내용은 Tsinghua AIR 등은 아미노산에서 원자 규모까지 최초의 단백질 언어 모델인 ESM-AA를 제안했습니다.의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!