
Éditeur | Feuille de chou
Le modèle de base pré-entraîné à grande échelle a connu un grand succès dans les domaines non médicaux. Cependant, la formation de ces modèles nécessite souvent des ensembles de données volumineux et complets, contrairement aux ensembles de données plus petits et plus spécialisés courants en imagerie biomédicale.
Des chercheurs de l'Institut Fraunhofer de médecine numérique MEVIS en Allemagne ont proposé une stratégie d'apprentissage multitâche qui sépare le nombre de tâches de formation des besoins en mémoire.
Ils ont formé un modèle biomédical pré-entraîné universel (UMedPT) sur une base de données multitâche comprenant des images de tomographie, de microscopie et de rayons X et ont utilisé diverses stratégies d'étiquetage telles que la classification, la segmentation et la détection d'objets. Le modèle de base UMedPT surpasse les modèles STOA pré-entraînés ImageNet et précédents.
Dans le cadre d'une validation externe indépendante, il a été prouvé que les caractéristiques d'imagerie extraites à l'aide de l'UMedPT établissent une nouvelle norme en matière de transférabilité intercentrique.
L'étude s'intitulait « Surmonter la rareté des données en imagerie biomédicale avec un modèle multitâche fondamental » et a été publiée dans « Nature Computational Science » le 19 juillet 2024.

Le deep learning révolutionne progressivement l'analyse d'images biomédicales grâce à sa capacité à apprendre et à extraire des représentations d'images utiles.
La méthode générale consiste à pré-entraîner le modèle sur un ensemble de données d'images naturelles à grande échelle (comme ImageNet ou LAION), puis à l'affiner pour des tâches spécifiques ou à utiliser directement les fonctionnalités pré-entraînées. Mais le réglage fin nécessite davantage de ressources informatiques.
Dans le même temps, le domaine de l’imagerie biomédicale nécessite une grande quantité de données annotées pour une pré-formation efficace en apprentissage profond, mais ces données sont souvent rares.
L'apprentissage multitâche (MTL) fournit une solution à la rareté des données en entraînant un modèle pour résoudre plusieurs tâches simultanément. Il exploite de nombreux ensembles de données de petite et moyenne taille en imagerie biomédicale pour pré-entraîner des représentations d'images adaptées à toutes les tâches et convient aux domaines où les données sont rares.
MTL a été appliqué à l'analyse d'images biomédicales de diverses manières, notamment en s'entraînant à partir de plusieurs ensembles de données de petite et moyenne taille pour différentes tâches et en utilisant plusieurs types d'étiquettes sur une seule image, démontrant que les fonctionnalités partagées peuvent améliorer les performances des tâches.
Dans les dernières recherches, afin de combiner plusieurs ensembles de données avec différents types d'étiquettes pour une pré-formation à grande échelle, des chercheurs de l'Institut MEVIS ont introduit une stratégie de formation multitâche et une architecture de modèle correspondante, notamment grâce à l'apprentissage de représentations polyvalentes selon différentes modalités. , maladies et types d'étiquettes pour remédier à la rareté des données en imagerie biomédicale.
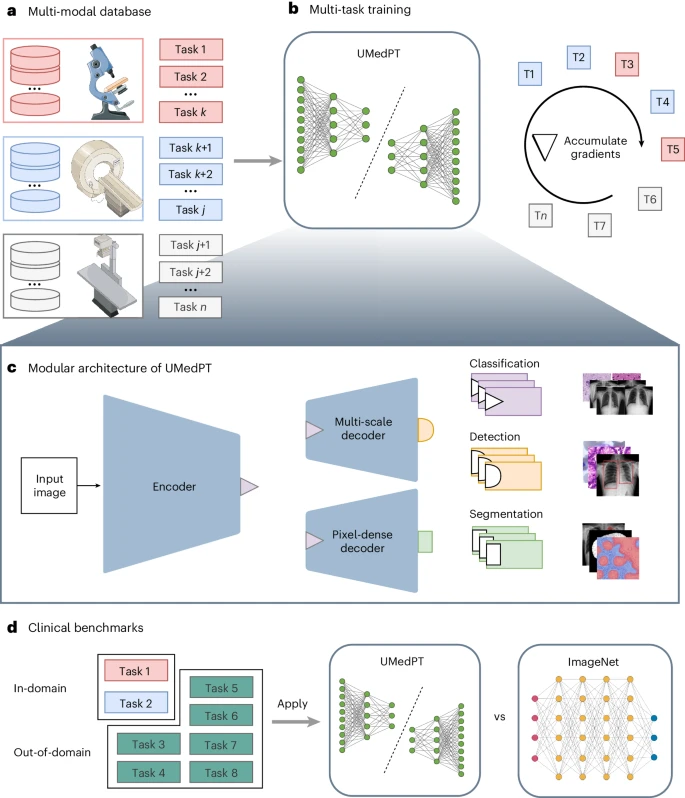
Pour faire face aux contraintes de mémoire rencontrées dans l'apprentissage multitâche à grande échelle, cette méthode adopte une boucle d'entraînement basée sur l'accumulation de gradient, dont l'expansion est presque illimitée par le nombre de tâches d'entraînement.
Sur cette base, les chercheurs ont formé un modèle de base d'imagerie biomédicale entièrement supervisé appelé UMedPT en utilisant 17 tâches et leurs annotations originales.
L'image ci-dessous montre l'architecture du réseau neuronal de l'équipe, qui se compose de blocs partagés comprenant un encodeur, un décodeur de segmentation et un décodeur de localisation, ainsi que des têtes spécifiques à des tâches. Les blocs partagés sont formés pour être applicables à toutes les tâches de pré-formation, aidant ainsi à extraire les caractéristiques communes, tandis que les superviseurs spécifiques aux tâches gèrent les calculs et les prévisions de pertes spécifiques aux étiquettes.
Les tâches définies comprennent trois types d'étiquettes supervisées : détection d'objets, segmentation et classification. Par exemple, les tâches de classification peuvent modéliser des biomarqueurs binaires, les tâches de segmentation peuvent extraire des informations spatiales et les tâches de détection d'objets peuvent être utilisées pour former des biomarqueurs basés sur le nombre de cellules.
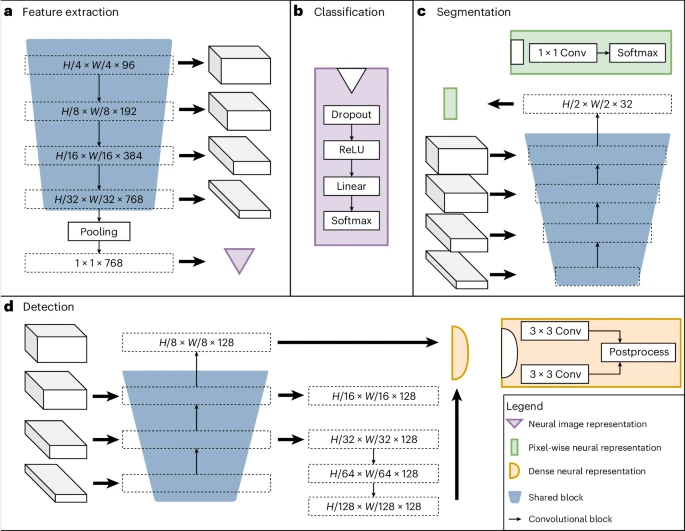
Illustration : L’architecture de l’UMedPT. (Source : article)
UMedPT correspond ou surpasse systématiquement les réseaux ImageNet pré-entraînés sur les tâches dans le domaine et hors domaine, tout en conservant de solides performances en utilisant moins de données d'entraînement lors de l'application directe de la représentation d'image (gel) et du réglage fin des paramètres.
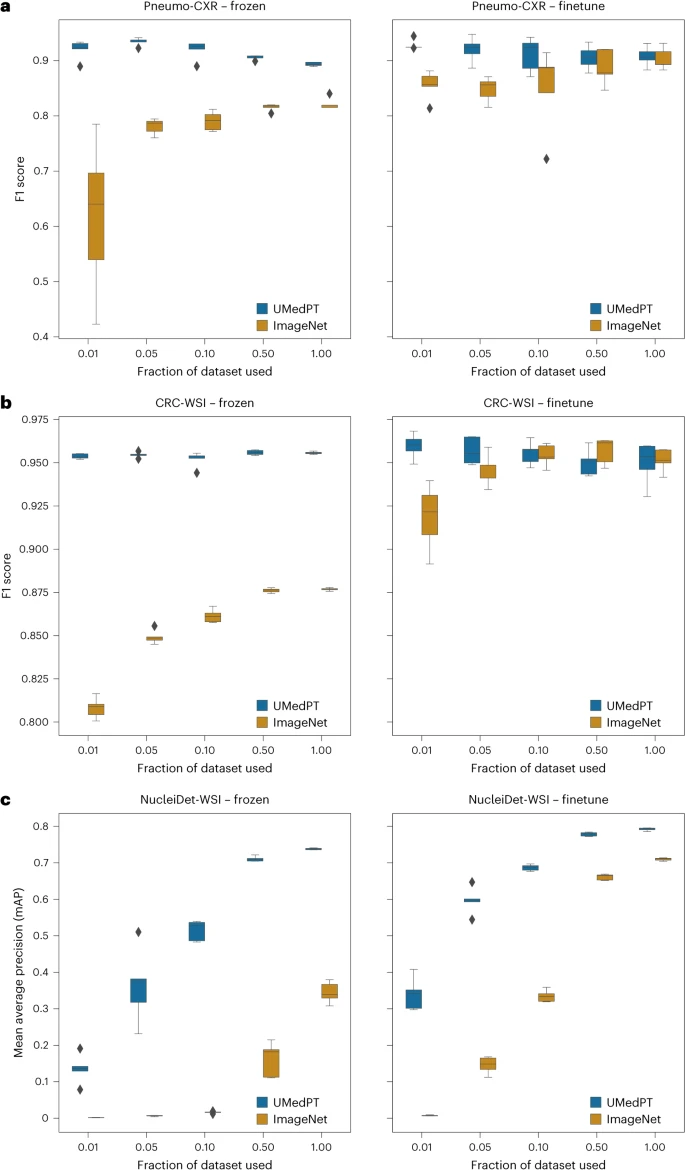
Illustration : Résultats des tâches au sein du domaine. (Source : article)
Pour les tâches de classification associées aux bases de données pré-entraînées, UMedPT est capable d'obtenir les meilleures performances de la base ImageNet sur toutes les configurations en utilisant seulement 1 % des données d'entraînement d'origine. Ce modèle atteint des performances supérieures en utilisant des encodeurs gelés par rapport au modèle utilisant un réglage fin.
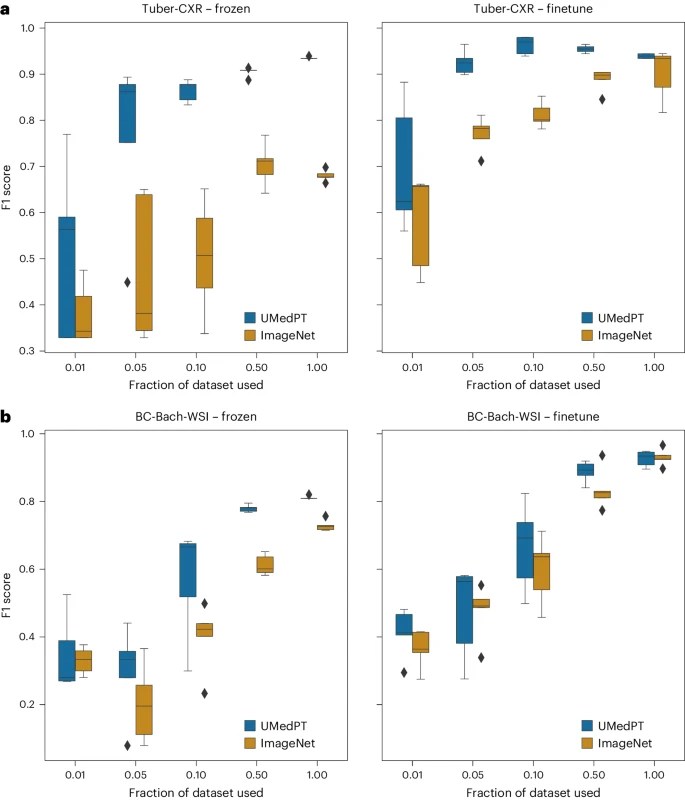
그림: 도메인 외부 작업에 대한 결과(출처: 논문)
도메인 외부 작업의 경우 UMedPT는 미세한 데이터 사용에도 불구하고 50% 이하의 데이터만 사용하여 ImageNet의 성능을 일치시킬 수 있습니다. 튜닝이 적용되었습니다.
또한 연구원들은 UMedPT의 성능을 문헌에 보고된 결과와 비교했습니다. 고정 인코더 구성을 사용할 때 UMedPT는 대부분의 작업에서 외부 참조 결과를 초과했습니다. 이 설정에서는 MedMNIST 데이터베이스 16의 곡선 아래 평균 면적(AUC)보다 성능도 뛰어납니다.
UMedPT의 동결 적용이 참조 결과를 능가하지 못한 작업이 도메인 외부에 있다는 점은 주목할 가치가 있습니다(유방암 분류를 위한 BC-Bach-WSI 및 CNS 종양 진단을 위한 CNS-MRI). 미세 조정을 통해 UMedPT를 사용한 사전 훈련은 모든 작업에서 외부 참조 결과보다 성능이 뛰어납니다.

그림: UMedPT가 다양한 이미징 영역의 작업에서 최첨단 성능을 달성하는 데 필요한 데이터의 양. (출처: 논문)
데이터가 부족한 분야의 향후 개발을 위한 기반으로서 UMedPT는 희귀 질환 및 소아 영상 촬영과 같이 대량의 데이터 수집이 특히 어려운 의료 분야에서 딥 러닝 애플리케이션의 가능성을 열어줍니다.
논문 링크:https://www.nature.com/articles/s43588-024-00662-z
관련 내용:https://www.nature.com/articles/s43588-024-00658- 9
위 내용은 AI 이미징의 새로운 표준, 원본 데이터의 1%만이 최고의 성능 달성 가능, 일반 의료 기본 모델 네이처(Nature) 저널에 게재의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!