오픈 소스! 홍콩 중국인, MIT, 푸단이 최초의 RNA 초석 모델 제안
단백질 분야와 달리 RNA 분야의 연구는 충분한 주석 데이터가 부족한 경우가 많습니다. 예를 들어 3D 데이터에는 1,000개 이상의 RNA만 있습니다. 이는 RNA 구조-기능 예측 작업에서 기계 학습 방법의 개발을 크게 제한합니다.
주석이 있는 데이터의 부족함을 보완하기 위해 이 기사에서는 다양한 유형의 RNA 연구에 풍부한 구조적, 기능적 지식을 제공할 수 있는 초석 모델인 RNA 기초 모델(RNA-FM)을 보여줍니다. 2,300만 개의 라벨이 지정되지 않은 RNA 서열을 기반으로 감독되지 않은 방식으로 훈련된 세계 최초의 RNA 초석 모델인 RNA-FM은 RNA 서열에 포함된 진화 및 구조적 패턴을 마이닝합니다.
RNA-FM은 단순한 다운스트림 모델만 일치시키거나 임베딩만 제공하면 되며 2차 구조 예측 20% 개선, 거리 개선 등 많은 다운스트림 작업에서 SOTA보다 훨씬 더 나은 성능을 달성할 수 있다는 점은 주목할 가치가 있습니다. 지도 예측은 30% 향상될 수 있습니다. 대규모 실험을 통해 이 모델이 고도로 일반화 가능하며 코로나19 및 mRNA의 조절 단편에도 사용될 수 있음이 입증되었습니다.

- 논문 사전 인쇄: https://arxiv.org/abs/2204.00300
- 코드 및 모델: https://github.com/ml4bio/RNA - FM
- Server: https://proj.cse.cuhk.edu.hk/rnafm
Introduction
최근 몇 년 동안 딥러닝을 기반으로 한 생물학적 컴퓨팅 방법이 이 분야에서 획기적인 발전을 이루었습니다. 가장 유명한 이정표는 Google DeepMind 팀이 개발한 엔드투엔드 단백질 3D 구조 예측 프레임워크 AlphaFold2입니다. 그러나 단백질은 많은 생물학적 분자 중 한 유형일 뿐이며, 단백질 생산의 원천인 유전자(DNA/RNA)는 후자보다 더 많은 기본 정보를 포함하고 있으며 더 중요한 연구 가치를 가지고 있습니다.
일반적으로 단백질은 코딩에 사용되는 RNA, 즉 고정된 mRNA가 고정된 단백질 서열로 번역되는 산물입니다. 실제로 코딩 RNA의 이 부분은 전체 RNA 서열의 2%만을 차지하고 나머지 98%는 비코딩 RNA(ncRNA)입니다. ncRNA는 단백질로 직접 "번역"되지는 않지만 특정 기능을 가진 3차 구조로 접히고 mRNA의 번역 과정이나 기타 생물학적 기능에서 조절 역할을 합니다. 따라서 ncRNA의 구조와 기능을 분석하는 것은 단백질 분석보다 더 기본적이고 복잡한 연구이다.
그러나 계산 방법이 보다 성숙한 단백질 분야에 비해 RNA 기반의 구조 및 기능 예측은 아직 초기 단계이며, 원래 단백질 분야에 적용 가능한 계산 방법은 RNA 분야로 직접 이전하기가 어렵습니다. . 이러한 계산 방법의 주요 한계는 일반적으로 RNA 데이터의 주석을 얻기가 어렵고, 적은 양의 데이터에 대한 주석을 완료하는 데 많은 실험 자원과 시간이 필요하다는 것입니다. 감독을 통해 높은 성과를 달성합니다. 비록 주석이 달린 데이터는 많지 않지만, RNA 분야에서는 실제로 주석이 없는 서열 데이터가 많이 축적되어 있다. 이 문서의 방법은 이러한 레이블이 지정되지 않은 데이터를 사용하여 다양한 다운스트림 작업에 대한 효과적인 추가 정보를 제공하는 것입니다.
이러한 고려 사항을 바탕으로 홍콩 중국인, MIT, 푸단 및 상하이 인공 지능 연구소 팀은 2,300만 개의 라벨이 지정되지 않은 순수 RNA 서열을 감독되지 않은 방식으로 훈련시킨 기초 모델 RNA 기초 모델(RNA-FM)을 제안했습니다. . 데이터가 훈련 과정 동안 주석 정보를 제공하지 않지만, RNA-FM은 여전히 감독되지 않은 방식으로 이러한 RNA 서열에 포함된 진화 및 구조적 패턴을 마이닝합니다.
RNA-FM이 다운스트림 RNA 구조 및 기능 예측 작업에 효과적으로 적용될 수 있다면 이러한 컴퓨팅 방법은 RNA-FM으로 요약된 지식의 이점을 확실히 활용하여 성능을 향상시킬 수 있습니다. RNA-FM의 업스트림 사전 훈련과 다운스트림 마이그레이션 및 적용 프레임워크는 아래 그림에 나와 있습니다.
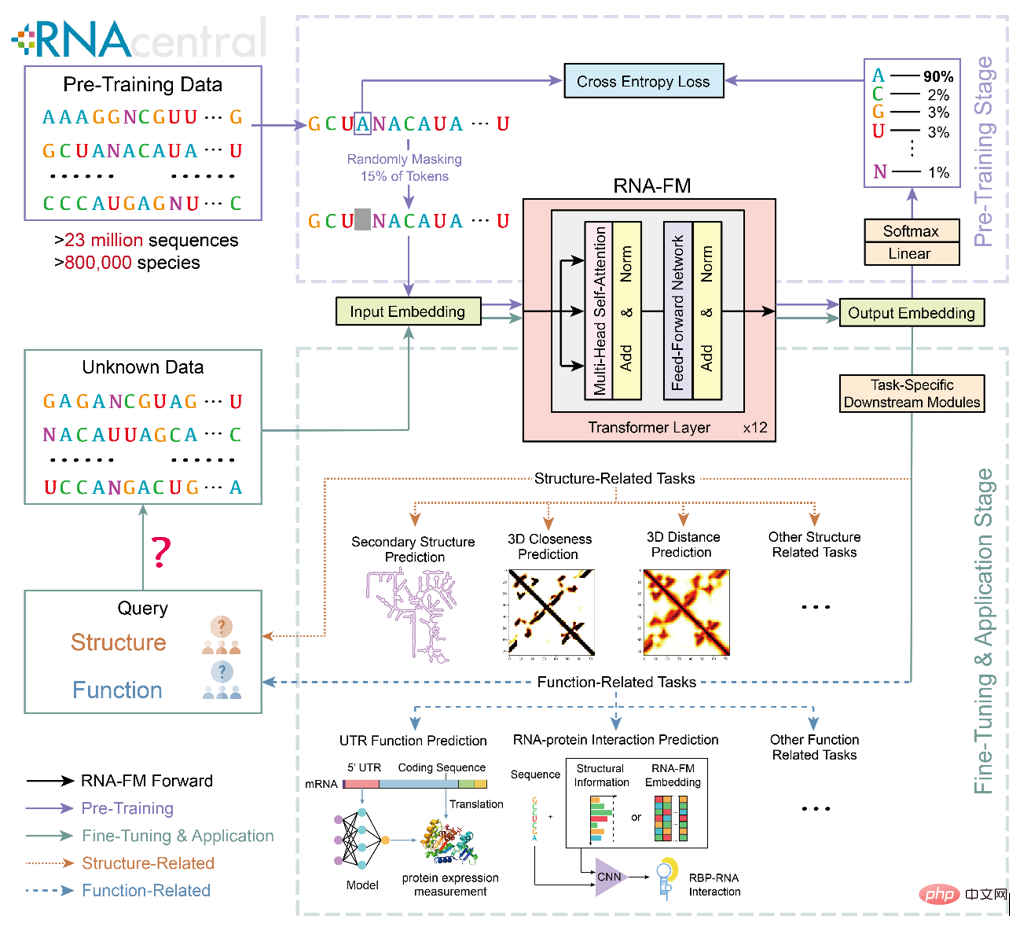
연구 개요
미리 훈련된 RNA-FM이 라벨링되지 않은 대량의 데이터로부터 '지식'을 학습했는지, 어떤 '지식'을 학습했는지 확인하기 위해 기사 일련의 임베딩 테스트 분석 을 수행했습니다.
먼저 다양한 특징에 대한 간단한 클러스터링 비교를 UMAP을 통해 직접 수행한 결과, 사전 훈련된 RNA-FM의 임베딩이 다른 임베딩보다 더 명확한 RNA 종 클러스터를 형성하는 것으로 나타났습니다. 이는 RNA-FM의 삽입이 RNA 종을 구별하기 위한 구조적 또는 기능적 정보를 포함하고 있음을 의미합니다.
그런 다음, 기사에서는 궤적 추론을 사용하여 RNA-FM 임베딩을 통해 다양한 종의 lncRNA 진화를 예측합니다. 아래 흐름도에서 예측된 종 간 진화의 유사 시간은 실제 종의 진화 정보와 대략 일치하며, 이는 RNA-FM 임베딩에도 진화 정보의 일부가 포함되어 있음을 나타냅니다.
RNA 종의 공동체 정보이든 lncRNA의 진화 정보이든, RNA-FM은 훈련 중에 이러한 라벨에 직접 노출되지 않았다는 점에 주목할 가치가 있습니다. RNA-FM은 완전히 자기 감독 방식으로 순수 서열에서 구조, 기능 및 진화와 관련된 패턴을 발견합니다.
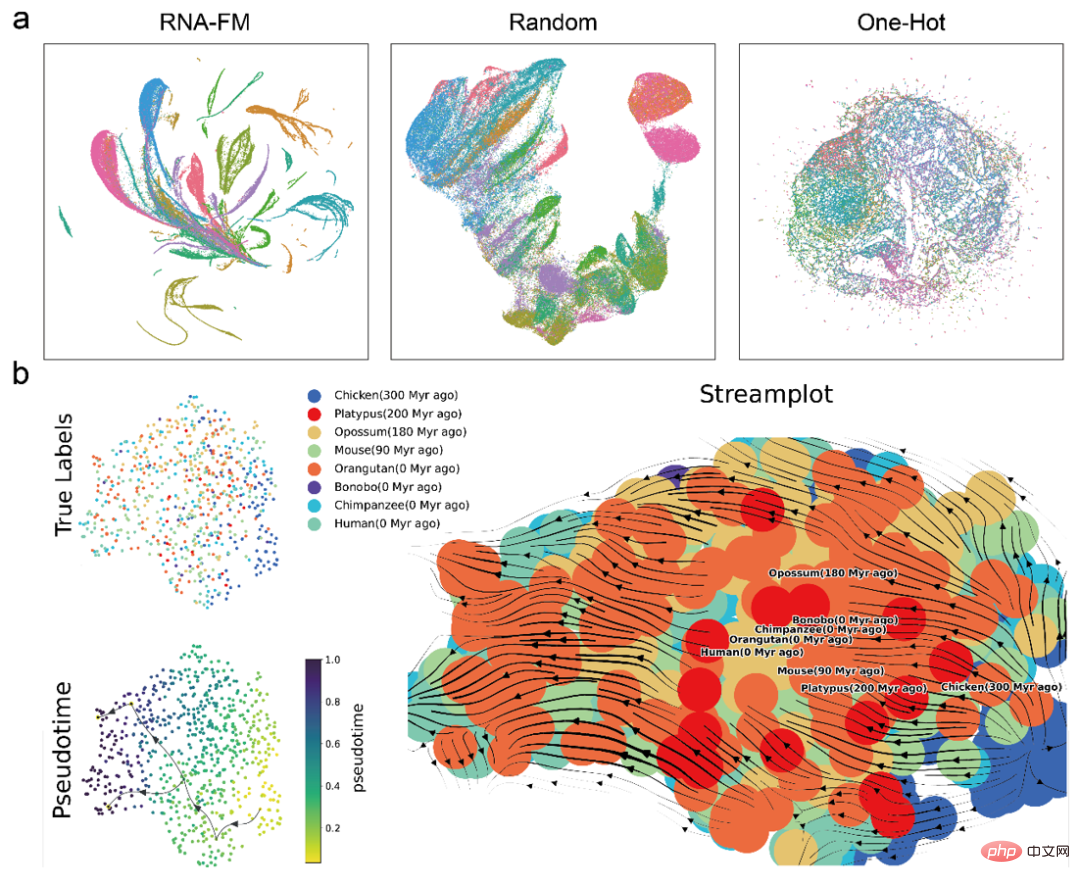
더 많은 실험 결과
RNA-FM의 삽입을 직접 분석하는 것 외에도 이 기사에서는 2차 구조, 접촉 예측, 거리 예측, 3차 구조 예측이 모두 상당한 개선을 이루었습니다.
특히 2차 구조 예측에서 기사는 RNA-FM을 백본으로 사용하고 간단한 ResNet 네트워크만 다운스트림 모델로 사용하여 두 개의 공개 데이터 세트에서 다른 12개의 최신 상태를 능가합니다. F1score에서 최고의 UFold보다 3~5% 포인트 더 좋습니다. UFold와의 정면 비교에서 RNA-FM은 대부분의 RNA 범주에서 UFold를 능가합니다. RNA-FM을 E2Efold와 결합하면 추가로 5% 성능 향상을 달성할 수 있습니다.

모델의 실제 적용 가치를 검증하기 위해 기사 에서는 RNA-FM을 사용하여 코로나19를 정확하게 예측하기 위해 RNA-FM을 사용하는 것을 포함하여 코로나19 데이터에 대한 완전한 분석을 수행합니다. -19 참조 게놈(29870nt) 주요 조절 요소를 사용하고 RNA-FM 임베딩을 사용하여 주요 코로나19 변종의 진화 추세를 대략적으로 예측합니다. 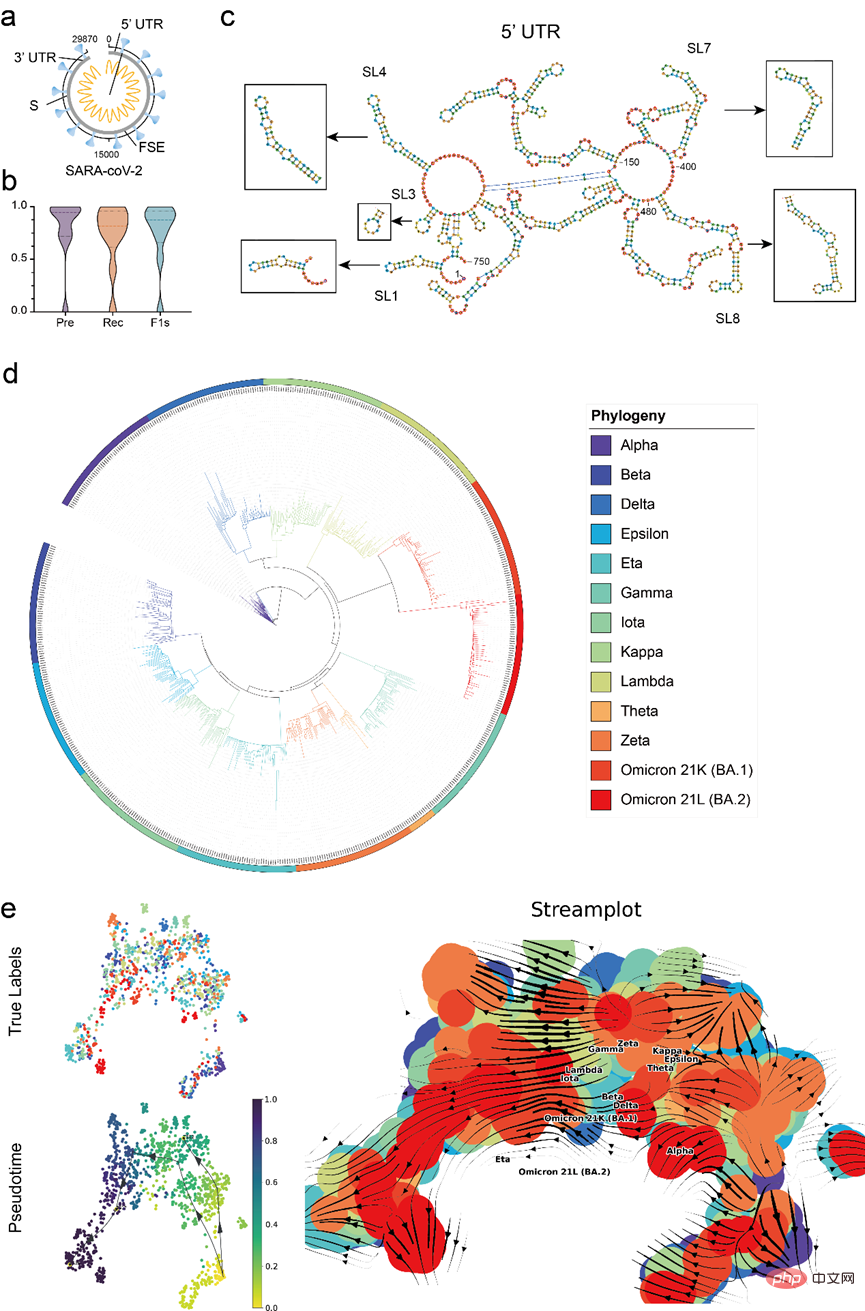
일반적으로 말하면, 분자의 구조가 그 기능을 결정합니다. RNA-FM은 RNA 구조 예측 작업을 훌륭하게 완료할 수 있는데, RNA-FM을 사용하여 기능 예측 결과도 향상시킬 수 있나요?
따라서 기사 에서는 RNA-FM 임베딩을 사용하여 RNA-단백질 역할을 예측하는 등 다운스트림 RNA 기능 예측 작업에 RNA-FM을 도입하려고 추가로 시도합니다.
RNA-FM 임베딩의 도입으로 모델의 성능이 향상되었음을 실험을 통해 입증했으며, 경우에 따라 실제 2차 구조 정보를 입력과 일치하는 예측 결과를 달성하기도 했습니다.
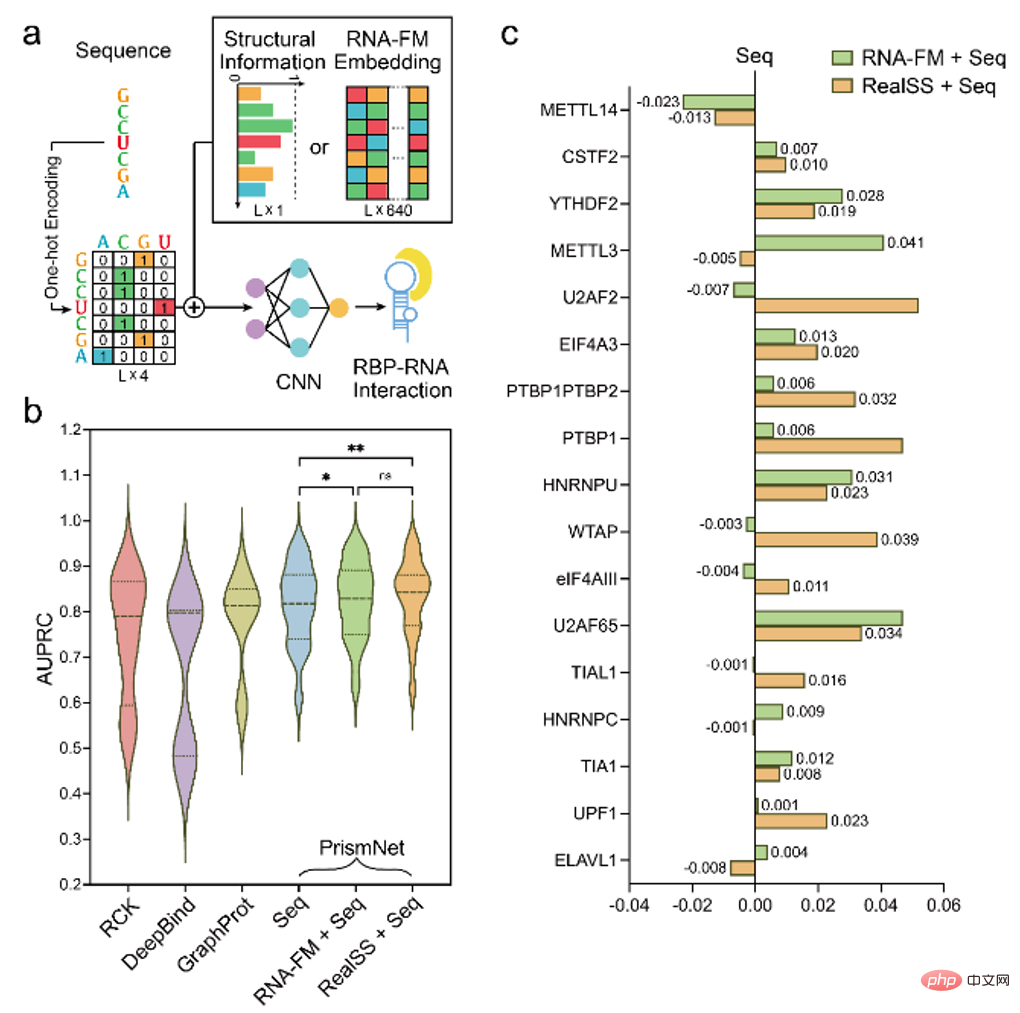
ncRNA 훈련을 기반으로 한 RNA-FM이 다른 RNA에도 일반화될 수 있는지 알아보기 위해 기사에서는 마침내 RNA-FM을 사용하여 5'UTR을 기반으로 한 단백질 발현 기능을 예측하려고 시도합니다. mRNA에. mRNA는 ncRNA에 속하지 않지만 그 위에 있는 5'UTR은 번역되지는 않지만 조절 기능을 하는 영역으로, 이는 ncRNA의 특성과 일치하며 훈련 데이터에는 나타나지 않습니다.
아래 그림에서 볼 수 있듯이 RNA-FM 임베딩을 포함하는 모델은 그렇지 않은 모델보다 항상 우수합니다. 성능 향상은 상대적으로 제한적이지만 RNA-FM도 비-ncRNA 데이터에 대한 특정 일반화 능력을 가지고 있음을 부분적으로 보여줍니다.

결론
일반적으로 이 기사에서는 레이블이 지정되지 않은 RNA 서열 데이터로 언어 모델 RNA-FM을 사전 훈련하고 직접 또는 간접적인 방법을 통해 구조나 기능과 같은 일련의 다양한 작업에 대해 포괄적인 검증을 수행하여 RNA가임을 증명합니다. -FM은 실제로 다운스트림 작업에서 컴퓨팅 방법의 성능을 효과적으로 향상시킬 수 있습니다.
RNA-FM의 출현으로 RNA 주석이 달린 데이터의 현재 상황이 어느 정도 완화되었으며, 다른 연구자들이 라벨이 없는 대량의 데이터에 접근할 수 있는 편리한 인터페이스를 제공하게 되면서 RNA 분야의 기본 모델이 될 것입니다. . 이 분야의 다양한 연구에 대한 강력한 지원과 도움을 제공합니다.
저자 소개
이 글의 공동 제1저자는 2명입니다. Chen Jiayang은 홍콩중문대학교의 연구 조교입니다. Hu Zhihang은 홍콩 중문 대학교의 박사 과정 후보자입니다.
이 기사에는 교신저자가 2명 있습니다. 푸단대학교 지능형 복합 시스템 연구소 및 상하이 인공 지능 연구소의 젊은 연구원 Sun Siqi, 홈페이지 https://intersun.github.io.
Li Yu, 홍콩 중문 대학교 조교수, MIT James Collins 연구소 객원 조교수, MIT 및 Harvard의 Broad Institute 연구 과학자, Harvard University Wyss Institute 객원 학자, Forbes 30 Under 30 아시아 목록 – 2022년 클래스, 의료 및 과학. 홈페이지: https://liyu95.com.
위 내용은 오픈 소스! 홍콩 중국인, MIT, 푸단이 최초의 RNA 초석 모델 제안의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!

핫 AI 도구

Undresser.AI Undress
사실적인 누드 사진을 만들기 위한 AI 기반 앱

AI Clothes Remover
사진에서 옷을 제거하는 온라인 AI 도구입니다.

Undress AI Tool
무료로 이미지를 벗다

Clothoff.io
AI 옷 제거제

AI Hentai Generator
AI Hentai를 무료로 생성하십시오.

인기 기사

뜨거운 도구

메모장++7.3.1
사용하기 쉬운 무료 코드 편집기

SublimeText3 중국어 버전
중국어 버전, 사용하기 매우 쉽습니다.

스튜디오 13.0.1 보내기
강력한 PHP 통합 개발 환경

드림위버 CS6
시각적 웹 개발 도구

SublimeText3 Mac 버전
신 수준의 코드 편집 소프트웨어(SublimeText3)

뜨거운 주제
 7530
7530
 15
15
 1379
1379
 52
52
 82
82
 11
11
 54
54
 19
19
 21
21
 76
76
 세계에서 가장 강력한 오픈 소스 MoE 모델이 여기에 있습니다. 중국의 기능은 GPT-4와 비슷하며 가격은 GPT-4-Turbo의 거의 1%에 불과합니다.
May 07, 2024 pm 04:13 PM
세계에서 가장 강력한 오픈 소스 MoE 모델이 여기에 있습니다. 중국의 기능은 GPT-4와 비슷하며 가격은 GPT-4-Turbo의 거의 1%에 불과합니다.
May 07, 2024 pm 04:13 PM
기존 컴퓨팅을 능가할 뿐만 아니라 더 낮은 비용으로 더 효율적인 성능을 달성하는 인공 지능 모델을 상상해 보세요. 이것은 공상과학 소설이 아닙니다. DeepSeek-V2[1], 세계에서 가장 강력한 오픈 소스 MoE 모델이 여기에 있습니다. DeepSeek-V2는 경제적인 훈련과 효율적인 추론이라는 특징을 지닌 전문가(MoE) 언어 모델의 강력한 혼합입니다. 이는 236B 매개변수로 구성되며, 그 중 21B는 각 마커를 활성화하는 데 사용됩니다. DeepSeek67B와 비교하여 DeepSeek-V2는 더 강력한 성능을 제공하는 동시에 훈련 비용을 42.5% 절감하고 KV 캐시를 93.3% 줄이며 최대 생성 처리량을 5.76배로 늘립니다. DeepSeek은 일반 인공지능을 연구하는 회사입니다.
 AI가 수학적 연구를 전복시킨다! 필즈상 수상자이자 중국계 미국인 수학자, Terence Tao가 좋아하는 11개 논문 발표 |
Apr 09, 2024 am 11:52 AM
AI가 수학적 연구를 전복시킨다! 필즈상 수상자이자 중국계 미국인 수학자, Terence Tao가 좋아하는 11개 논문 발표 |
Apr 09, 2024 am 11:52 AM
AI는 실제로 수학을 변화시키고 있습니다. 최근 이 문제에 주목하고 있는 타오저쉬안(Tao Zhexuan)은 '미국수학회지(Bulletin of the American Mathematical Society)' 최신호를 게재했다. '기계가 수학을 바꿀 것인가?'라는 주제를 중심으로 많은 수학자들이 그들의 의견을 표현했습니다. 저자는 필즈상 수상자 Akshay Venkatesh, 중국 수학자 Zheng Lejun, 뉴욕대학교 컴퓨터 과학자 Ernest Davis 등 업계의 유명 학자들을 포함해 강력한 라인업을 보유하고 있습니다. AI의 세계는 극적으로 변했습니다. 이 기사 중 상당수는 1년 전에 제출되었습니다.
 Google은 열광하고 있습니다. JAX 성능이 Pytorch와 TensorFlow를 능가합니다! GPU 추론 훈련을 위한 가장 빠른 선택이 될 수 있습니다.
Apr 01, 2024 pm 07:46 PM
Google은 열광하고 있습니다. JAX 성능이 Pytorch와 TensorFlow를 능가합니다! GPU 추론 훈련을 위한 가장 빠른 선택이 될 수 있습니다.
Apr 01, 2024 pm 07:46 PM
Google이 추진하는 JAX의 성능은 최근 벤치마크 테스트에서 Pytorch와 TensorFlow를 능가하여 7개 지표에서 1위를 차지했습니다. 그리고 JAX 성능이 가장 좋은 TPU에서는 테스트가 이루어지지 않았습니다. 개발자들 사이에서는 여전히 Tensorflow보다 Pytorch가 더 인기가 있습니다. 그러나 앞으로는 더 큰 모델이 JAX 플랫폼을 기반으로 훈련되고 실행될 것입니다. 모델 최근 Keras 팀은 기본 PyTorch 구현을 사용하여 세 가지 백엔드(TensorFlow, JAX, PyTorch)와 TensorFlow를 사용하는 Keras2를 벤치마킹했습니다. 첫째, 그들은 주류 세트를 선택합니다.
 안녕하세요, 일렉트릭 아틀라스입니다! 보스턴 다이나믹스 로봇 부활, 180도 이상한 움직임에 겁먹은 머스크
Apr 18, 2024 pm 07:58 PM
안녕하세요, 일렉트릭 아틀라스입니다! 보스턴 다이나믹스 로봇 부활, 180도 이상한 움직임에 겁먹은 머스크
Apr 18, 2024 pm 07:58 PM
Boston Dynamics Atlas가 공식적으로 전기 로봇 시대에 돌입했습니다! 어제 유압식 Atlas가 역사의 무대에서 "눈물을 흘리며" 물러났습니다. 오늘 Boston Dynamics는 전기식 Atlas가 작동 중이라고 발표했습니다. 상업용 휴머노이드 로봇 분야에서는 보스턴 다이내믹스가 테슬라와 경쟁하겠다는 각오를 다진 것으로 보인다. 새 영상은 공개된 지 10시간 만에 이미 100만 명이 넘는 조회수를 기록했다. 옛 사람들은 떠나고 새로운 역할이 등장하는 것은 역사적 필연이다. 올해가 휴머노이드 로봇의 폭발적인 해라는 것은 의심의 여지가 없습니다. 네티즌들은 “로봇의 발전으로 올해 개막식도 인간처럼 생겼고, 자유도도 인간보다 훨씬 크다. 그런데 정말 공포영화가 아닌가?”라는 반응을 보였다. 영상 시작 부분에서 아틀라스는 바닥에 등을 대고 가만히 누워 있는 모습입니다. 다음은 입이 떡 벌어지는 내용이다
 MLP를 대체하는 KAN은 오픈소스 프로젝트를 통해 컨볼루션으로 확장되었습니다.
Jun 01, 2024 pm 10:03 PM
MLP를 대체하는 KAN은 오픈소스 프로젝트를 통해 컨볼루션으로 확장되었습니다.
Jun 01, 2024 pm 10:03 PM
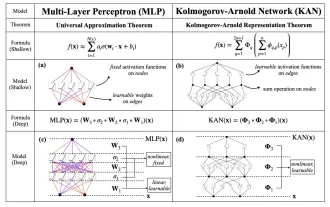
이달 초 MIT와 기타 기관의 연구자들은 MLP에 대한 매우 유망한 대안인 KAN을 제안했습니다. KAN은 정확성과 해석성 측면에서 MLP보다 뛰어납니다. 그리고 매우 적은 수의 매개변수로 더 많은 수의 매개변수를 사용하여 실행되는 MLP보다 성능이 뛰어날 수 있습니다. 예를 들어 저자는 KAN을 사용하여 더 작은 네트워크와 더 높은 수준의 자동화로 DeepMind의 결과를 재현했다고 밝혔습니다. 구체적으로 DeepMind의 MLP에는 약 300,000개의 매개변수가 있는 반면 KAN에는 약 200개의 매개변수만 있습니다. KAN은 MLP와 같이 강력한 수학적 기반을 가지고 있으며, KAN은 Kolmogorov-Arnold 표현 정리를 기반으로 합니다. 아래 그림과 같이 KAN은
 권장 사항: 우수한 JS 오픈 소스 얼굴 감지 및 인식 프로젝트
Apr 03, 2024 am 11:55 AM
권장 사항: 우수한 JS 오픈 소스 얼굴 감지 및 인식 프로젝트
Apr 03, 2024 am 11:55 AM
얼굴 검출 및 인식 기술은 이미 상대적으로 성숙하고 널리 사용되는 기술입니다. 현재 가장 널리 사용되는 인터넷 응용 언어는 JS입니다. 웹 프런트엔드에서 얼굴 감지 및 인식을 구현하는 것은 백엔드 얼굴 인식에 비해 장점과 단점이 있습니다. 장점에는 네트워크 상호 작용 및 실시간 인식이 줄어 사용자 대기 시간이 크게 단축되고 사용자 경험이 향상된다는 단점이 있습니다. 모델 크기에 따라 제한되고 정확도도 제한됩니다. js를 사용하여 웹에서 얼굴 인식을 구현하는 방법은 무엇입니까? 웹에서 얼굴 인식을 구현하려면 JavaScript, HTML, CSS, WebRTC 등 관련 프로그래밍 언어 및 기술에 익숙해야 합니다. 동시에 관련 컴퓨터 비전 및 인공지능 기술도 마스터해야 합니다. 웹 측면의 디자인으로 인해 주목할 가치가 있습니다.
 공장에서 일하는 테슬라 로봇, 머스크 : 올해 손의 자유도가 22도에 달할 것!
May 06, 2024 pm 04:13 PM
공장에서 일하는 테슬라 로봇, 머스크 : 올해 손의 자유도가 22도에 달할 것!
May 06, 2024 pm 04:13 PM
테슬라의 로봇 옵티머스(Optimus)의 최신 영상이 공개됐는데, 이미 공장에서 작동이 가능한 상태다. 정상 속도에서는 배터리(테슬라의 4680 배터리)를 다음과 같이 분류합니다. 공식은 또한 20배 속도로 보이는 모습을 공개했습니다. 작은 "워크스테이션"에서 따고 따고 따고 : 이번에 출시됩니다. 영상에는 옵티머스가 공장에서 이 작업을 전 과정에 걸쳐 사람의 개입 없이 완전히 자율적으로 완료하는 모습이 담겨 있습니다. 그리고 Optimus의 관점에서 보면 자동 오류 수정에 중점을 두고 구부러진 배터리를 집어 넣을 수도 있습니다. NVIDIA 과학자 Jim Fan은 Optimus의 손에 대해 높은 평가를 했습니다. Optimus의 손은 세계의 다섯 손가락 로봇 중 하나입니다. 가장 능숙합니다. 손은 촉각적일 뿐만 아니라
 FisheyeDetNet: 어안 카메라를 기반으로 한 최초의 표적 탐지 알고리즘
Apr 26, 2024 am 11:37 AM
FisheyeDetNet: 어안 카메라를 기반으로 한 최초의 표적 탐지 알고리즘
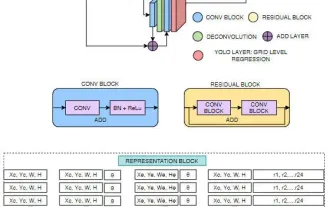
Apr 26, 2024 am 11:37 AM
표적 탐지는 자율주행 시스템에서 상대적으로 성숙한 문제이며, 그 중 보행자 탐지는 가장 먼저 배포되는 알고리즘 중 하나입니다. 대부분의 논문에서 매우 포괄적인 연구가 수행되었습니다. 그러나 서라운드 뷰를 위한 어안 카메라를 사용한 거리 인식은 상대적으로 덜 연구되었습니다. 큰 방사형 왜곡으로 인해 표준 경계 상자 표현은 어안 카메라에서 구현하기 어렵습니다. 위의 설명을 완화하기 위해 확장된 경계 상자, 타원 및 일반 다각형 디자인을 극/각 표현으로 탐색하고 인스턴스 분할 mIOU 메트릭을 정의하여 이러한 표현을 분석합니다. 제안된 다각형 형태의 모델 fisheyeDetNet은 다른 모델보다 성능이 뛰어나며 동시에 자율 주행을 위한 Valeo fisheye 카메라 데이터 세트에서 49.5% mAP를 달성합니다.




