하나의 AI로 9,808명의 암환자의 약물에 대한 임상 반응을 완전히 예측할 수 있습니다.
그리고 결과는 임상 관찰과 일치합니다.
뉴욕 시립대학교 Lei Xie 팀이 가져온 CODE-AE(context-aware deconfounding autoencoder)의 최신 결과입니다.

다양한 환자의 약물에 대한 구체적인 반응을 예측할 수 있는 새로운 상황별 자동 인코딩 모델을 제안합니다.
이는 신약 개발과 임상시험에 큰 영향을 미칠 것입니다.
기존 모델에서는 신약이 개발, 테스트되고 완전히 출시되기까지 거의 10년이 걸리며, 소비되는 자금은 전례 없이 거대하여 쉽사리 10억 달러에 달한다는 점을 아셔야 합니다.
신약의 인체 내 반응은 예측하기 어렵기 때문에 주기가 너무 길고, 테스트를 위해서는 반복적인 시도가 필요한 경우가 많습니다.
그리고 AI가 데이터를 사용하여 예측을 할 수 있다면 신약 출시 시간을 크게 단축하고 비용을 절감할 수 있습니다.
현재 이 연구는 네이처(Nature) 부저널 '네이처 머신 인텔리전스(Nature Machine Intelligence)'에 게재됐다.
간단히 말하면 CODE-AE는 신약의 체외 세포 검증 데이터를 사용하여 인체 내 약물의 반응을 예측합니다.
이렇게 하면 AI 모델 훈련이 환자 임상 데이터에 의존하는 것을 방지할 수 있습니다.
과거 AI가 임상 반응 예측에 그다지 효과적이지 못한 가장 큰 이유는 대규모의 지속적인 임상 반응 데이터를 수집하기가 너무 어렵다는 것입니다.
메커니즘 관점에서 연구자들은 약물 바이오마커를 소스 도메인과 타겟 도메인으로 구분합니다.
소스 도메인은 테스트 샘플과 다른 도메인을 나타내지만 감독 정보가 풍부해 체외 세포 검증 데이터로 이해할 수 있습니다.
대상 도메인은 테스트 샘플이 위치한 도메인입니다. 라벨이 없거나 몇 개만 있는, 즉 환자 데이터입니다.
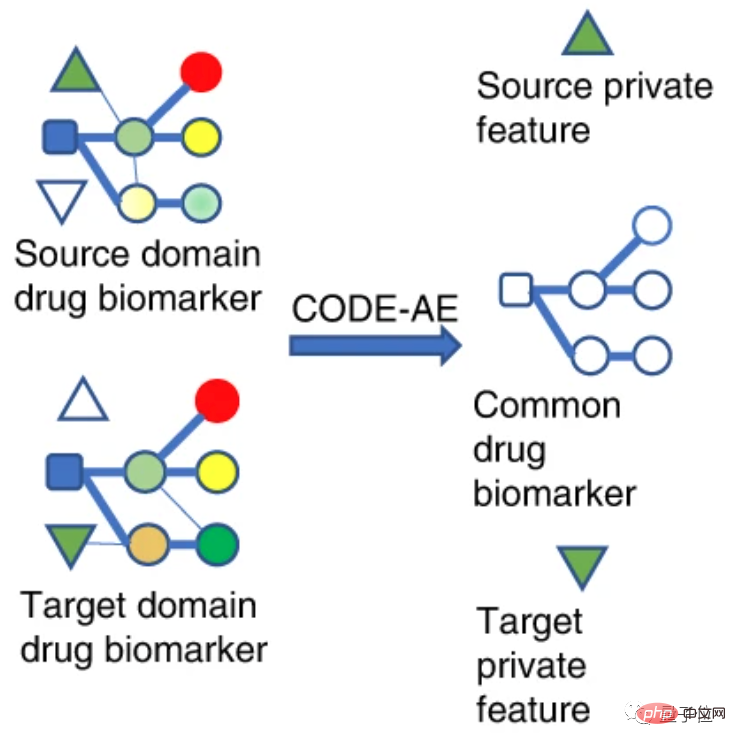
다른 필드의 데이터 기능을 동일한 기능 공간에 매핑하여 이 공간에서의 거리가 최대한 가까워지도록 합니다.
따라서 특징 공간의 소스 도메인에서 훈련된 목적 함수를 대상 도메인으로 전송하여 대상 도메인의 정확도를 향상시킬 수 있습니다.
본 연구의 맥락에서 소스 도메인과 타겟 도메인은 모두 약물 바이오마커의 데이터 특성, 즉 약물 타겟의 데이터 특성입니다.
모델 프레임워크를 구체적으로 살펴보면 크게 사전 학습, 미세 조정, 추론의 세 부분으로 나뉩니다.
사전 훈련은 주로 자기 지도 학습을 사용하여 체외 세포 데이터와 환자 데이터의 레이블이 지정되지 않은 유전자 발현 프로필을 임베딩 공간에 매핑하는 기능 인코딩 모듈을 구축합니다. 이러한 방식으로 일부 교란 요인을 제거할 수 있으며 두 데이터의 잠재 분포를 일관되게 유지하여 체계적인 편향을 제거할 수 있습니다.
미세 조정 단계는 사전 훈련을 기반으로 지도 모델을 추가하고 레이블이 지정된 시험관 내 세포 데이터를 훈련에 사용하는 것입니다.
마지막으로 추론 단계에서는 사전 훈련을 통해 얻은 환자를 먼저 명확화하고 내장한 다음 조정된 모델을 사용하여 약물에 대한 환자의 반응을 예측합니다.

이 모드에서 CODE-AE에는 두 가지 기능이 있습니다.
첫째, 일관되지 않은 샘플에서 일반적인 생물학적 신호와 개인 표현을 추출하여 다양한 데이터 패턴으로 인한 간섭을 제거할 수 있습니다.
둘째, 약물 반응 신호와 교란 요인을 분리한 후 국소 정렬도 달성할 수 있습니다.
요약하면 CODE-AE는 레이블이 있는 데이터와 레이블이 없는 데이터의 불일치 데이터 패턴 임베딩 공간에서 고유한 특징을 선택하는 프로세스로 이해될 수 있습니다.
모델의 효과를 입증하기 위해 연구자들은 9,808명의 암 환자의 약물 적합성을 예측했습니다.
환자의 상태에 대한 모델이 예측한 현장 결과가 그가 사용하는 약물 타겟과 관련이 있으면 예측이 정확하다는 것을 증명합니다.
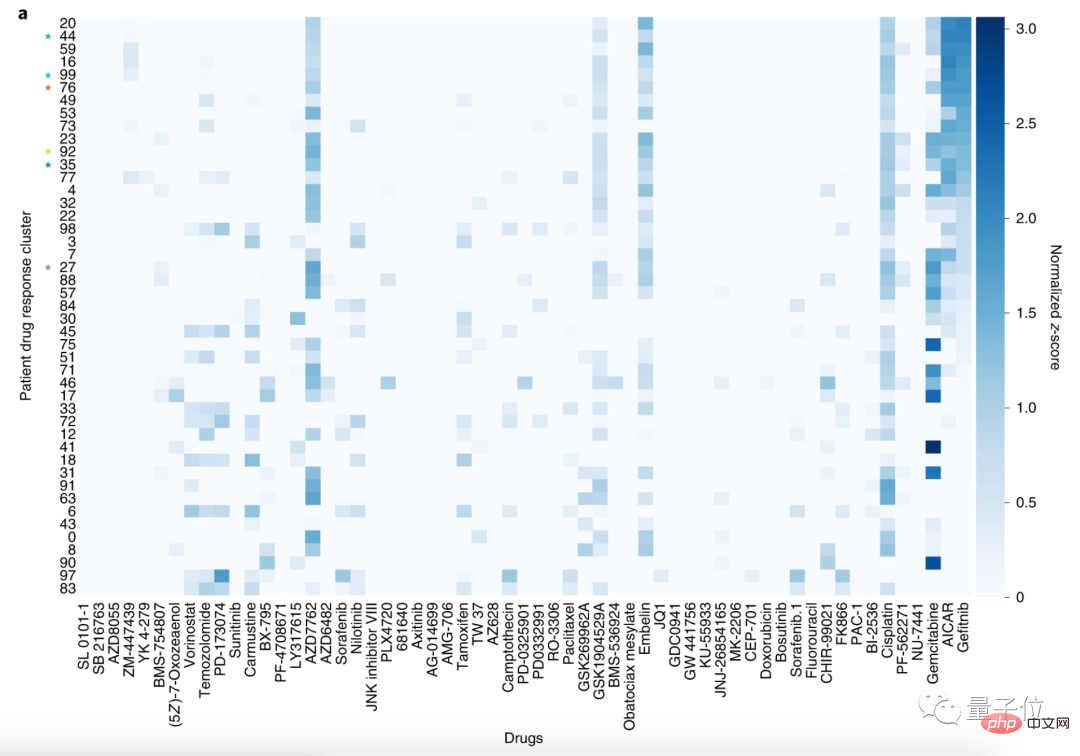
그런 다음 연구원들은 환자를 100개의 클러스터로 나누고 59개의 약물을 30개의 클러스터로 나누었습니다.
이 분석 방법을 통해 유사한 약물 반응 프로파일을 가진 환자를 그룹화할 수 있습니다.
여기에서는 폐편평세포암종(LSCC)과 비소세포폐암(NSCLC) 환자의 군집을 예로 들어보겠습니다.
59개 약물 중 LSCC에 가장 민감한 약물은 게피티닙, AICAR, 젬시타빈입니다.
게피티닙과 AICAR의 표적은 모두 표피성장인자수용체(EGFR)이며, 젬시타빈은 EGFR 돌연변이가 없는 비소세포폐암 치료에 자주 사용됩니다.
논문에서는 이러한 약물의 작용 모드와 일치하여 CODE-AE가 게피티닙과 AICAR을 사용하는 환자가 유사한 약물 반응 프로필을 가지고 있음을 발견했다고 밝혔습니다.
즉, CODE-AE는 환자 치료에 대한 올바른 타겟, 즉 적용 가능한 약물을 예측할 수 있다는 의미입니다.
위 연구팀은 뉴욕시립대학교 출신입니다.
교신 저자는 중국 과학 기술 대학교에서 고분자 물리학 학사 학위를 취득한 Lei Xie입니다.
Rutgers University에서 컴퓨터 과학 석사 학위를 취득했지만 화학 학위도 받았습니다.

연구팀의 다음 단계는 신약의 임상 반응의 농도 및 대사 측면에서 CODE-AE의 예측 기능을 개발하는 것으로 이해됩니다.
연구원들은 AI 모델이 약물이 인체에 미치는 부작용을 예측하는 데에도 적용될 수 있다고 말했습니다.
네이처의 하위저널인 '네이처 머신 인텔리전스(Nature Machine Intelligence)'는 인공지능과 생명과학 분야의 학제간 응용 연구를 전문으로 하며, 매년 평균 포함되는 논문 수가 약 60편 정도라는 점을 언급할 가치가 있습니다.
논문주소: https://www.nature.com/articles/s42256-022-00541-0
참고링크: https://phys.org/news/2022-10-ai-accurately-human-response See More -drug.html
위 내용은 중국 팀이 암 환자에게 적합한 약물을 예측하는 AI를 성공적으로 개발했으며 그 결과는 네이처(Nature) 저널에 게재되었습니다.의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!