세계 최초: 단백질 측쇄 예측 및 서열 설계 문제를 극복하기 위한 Molecular Heart의 오픈 소스 새로운 AI 알고리즘
단백질의 구조와 기능의 형성은 곁사슬 원자 간의 상호작용에 크게 좌우됩니다. 따라서 정확한 단백질 곁사슬 예측(PSCP)은 단백질 구조 예측과 단백질 설계 문제를 해결하는 핵심 고리입니다. 그러나 기존의 단백질 구조 예측은 대부분 주쇄 구조에 집중되어 있었고, 측쇄 구조 예측은 항상 완전히 해결되지 않은 어려운 문제였습니다.
최근 Molecular Heart Xu Jinbo 팀은 새로운 PSCP 심층 아키텍처 AttnPacker를 출시했습니다. 이는 속도, 메모리 효율성 및 전반적인 정확도가 크게 향상되었으며 현재 가장 잘 알려진 사이드체인 구조 예측 알고리즘입니다. 단백질 측쇄 예측과 서열 설계를 동시에 수행할 수 있는 AI 알고리즘입니다.
이 논문은 PNAS(Proceedings of the National Academy of Sciences)에 게재되었으며 사전 훈련된 모델, 소스 코드 및 추론 스크립트는 Github에서 오픈 소스로 공개되었습니다.

- 논문 링크: https://www.pnas.org/doi/10.1073/pnas.2216438120#supplementary-materials
- 오픈 소스 링크 : https:/ / github.com/MattMcPartlon/AttnPacker
Background
단백질은 여러 아미노산으로 접혀져 있으며 그 구조는 주쇄와 측쇄로 나누어져 있습니다. 측쇄의 차이는 단백질 구조와 기능, 특히 생물학적 활성에 큰 영향을 미칩니다. 과학자들은 측쇄 구조에 대한 명확한 이해를 바탕으로 단백질의 3차원 구조를 보다 정확하게 파악하고 단백질 간 상호 작용을 분석하며 합리적인 단백질 설계를 수행할 수 있습니다. 약물 설계 분야에 적용하면 과학자들은 약물과 수용체에 적합한 결합 부위를 빠르고 정확하게 찾을 수 있으며, 효소 최적화 분야에서는 필요에 따라 결합 부위를 최적화하거나 설계할 수도 있습니다. 변환을 통해 다중 측쇄가 허용됩니다. 보다 효율적이고 구체적인 촉매 효과를 달성하기 위해 촉매 반응에 참여합니다.
현재 대부분의 단백질 구조 예측 알고리즘은 주로 주쇄의 구조 분석에 중점을 두고 있지만, 단백질 측쇄 구조 예측은 여전히 완전히 극복되지 않은 문제입니다. AlphaFold2와 같은 대중적인 단백질 구조 예측 알고리즘이든, DLPacker 및 RosettaPacker와 같은 측쇄 구조 예측에 중점을 둔 알고리즘이든 정확도나 속도는 만족스럽지 않습니다. 이는 또한 단백질 설계에 제한을 가합니다.
RosettaPacker와 같은 전통적인 방법은 에너지 최적화 방법을 주로 사용하는데, 먼저 곁사슬 원자의 분포를 그룹화한 다음 특정 아미노산에 대한 곁사슬 그룹을 검색하여 가장 작은 에너지와 결합을 찾습니다. 이러한 방법은 주로 연구원이 선택한 로타머 라이브러리, 에너지 기능 및 에너지 최소화 절차와 다르며 정확도는 검색 휴리스틱 및 이산 샘플링 절차를 사용하여 제한됩니다. 업계에는 PSCP를 이미지 대 이미지 변환 문제로 공식화하고 U-net 모델 구조를 채택하는 DLPacker와 같은 딥러닝 기반의 사이드 체인 예측 방법도 있습니다. 그러나 예측 정확도와 속도는 여전히 이상적이지 않습니다.
Method
AttnPacker는 단백질 측쇄 좌표를 예측하기 위한 엔드투엔드 딥러닝 방법입니다. 원자 충돌이 적고 결합 길이와 각도가 더 이상적이며 물리적으로 더 실현 가능한 직접 예측된 측쇄 구조를 사용하여 측쇄 상호 작용을 공동으로 시뮬레이션합니다.
특히 AttnPacker는 PSCP의 기하학적 및 관계형 측면을 활용하는 깊이 맵 변환기 아키텍처를 도입합니다. AlphaFold2에서 영감을 받은 Molecular Heart는 삼각형 주의 및 곱셈 업데이트를 계산하는 그래프 기반 프레임워크를 사용하여 쌍별 특징을 최적화하는 위치 인식 삼각형 업데이트를 제안합니다. 이 접근 방식을 사용하면 AttnPacker는 훨씬 적은 메모리와 더 높은 용량 모델을 갖게 됩니다. 또한 Molecular Heart는 여러 SE(3) 등변 주의 메커니즘을 탐색하고 3D 점에서 학습하기 위한 등변 변환기 아키텍처를 제안합니다.
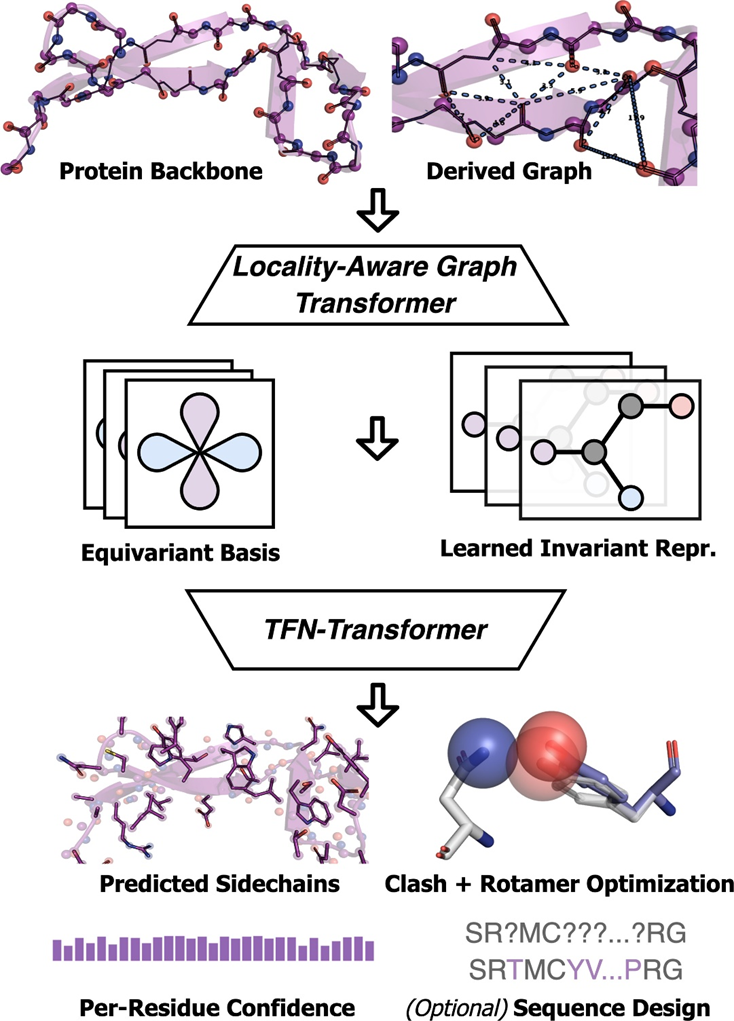
AttnPacker가 프로세스를 실행합니다. 단백질 백본 좌표와 서열을 입력으로 사용하고, 좌표 정보를 바탕으로 공간 특징 맵과 등변 기저를 도출합니다. 특징 맵은 불변 그래프 변환기 모듈에 의해 처리된 후 예측된 측쇄 좌표, 각 잔기에 대한 신뢰 점수 및 선택적 설계 시퀀스를 출력하는 등변 TFN 변환기로 전달됩니다. 예측된 좌표는 모든 공간 충돌을 제거하고 이상적인 형상을 보장하기 위해 사후 처리됩니다.
Effect
AttnPacker는 예측 성능 측면에서 자연적 및 비자연적 백본 구조 모두에 대한 정확성과 효율성 향상을 보여줍니다. 동시에 물리적 타당성이 보장되고 이상적인 결합 길이 및 각도의 편차가 무시할 수 있으며 최소한의 원자 입체 장애가 생성됩니다.
Molecular Heart는 CASP13 및 CASP14 천연 및 비원래 단백질 백본 데이터 세트에 대해 AttnPacker와 현재 최첨단 방법인 SCWRL4, FASPR, RosettaPacker 및 DLPacker에 대한 비교 테스트를 수행합니다. 결과에 따르면 AttnPacker는 CASP13 및 CASP14 기본 백본에 대한 기존 단백질 측쇄 예측 방법보다 훨씬 뛰어난 성능을 보여주며, 평균 재구성 RMSD는 각 테스트 세트의 차선책 방법보다 18% 이상 낮습니다. AttnPacker는 또한 딥 러닝 방법인 DLPacker보다 성능이 뛰어나 평균 RMSD를 11% 이상 줄이는 동시에 사이드체인 2면체 정확도를 크게 향상시킵니다. 정확성 외에도 AttnPacker는 다른 방법보다 원자 충돌이 훨씬 적습니다.
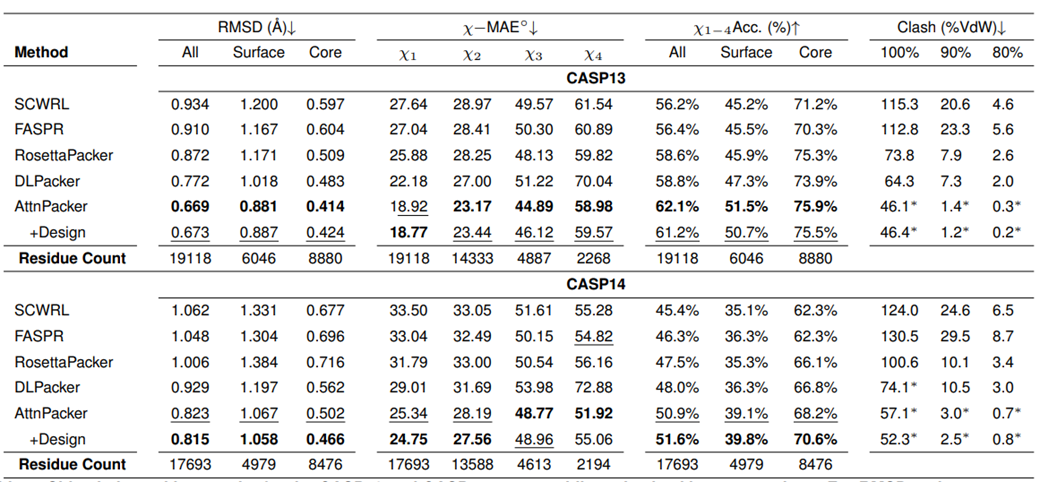
천연 주쇄 구조를 부여했을 때 CASP13 및 CASP14 표적 단백질에 대한 각 알고리즘의 측쇄 구조 예측 결과입니다. 별표는 평균 충돌 값이 기본 구조(CASP13의 경우 56.0, 5.9, 0.4, CASP14의 경우 80.4, 7.9, 2.5)보다 낮다는 것을 나타냅니다.
CASP13 및 CASP14 비기본 백본에서 AttnPacker는 다른 방법보다 훨씬 우수하며 원자 충돌도 다른 방법보다 훨씬 적습니다.
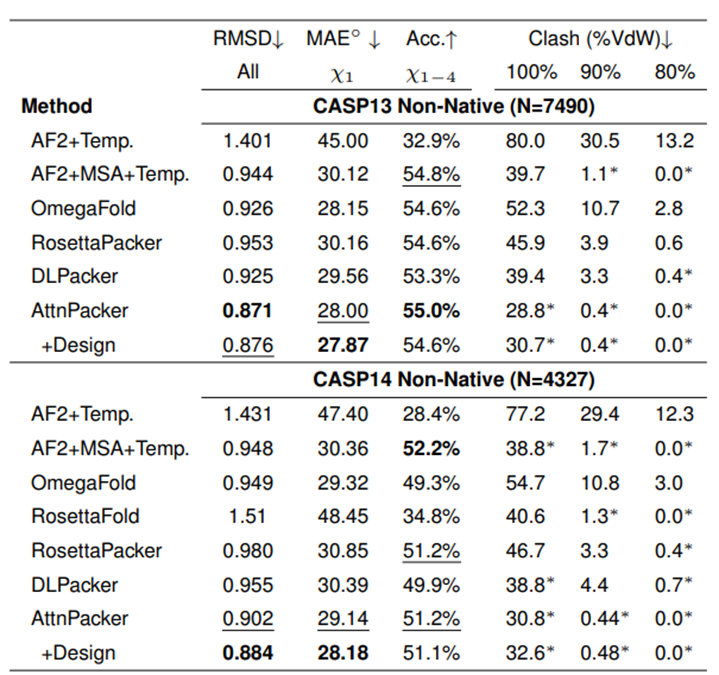
비천연 백본 구조를 부여했을 때 CASP13 및 CASP14 타겟 단백질에 대한 각 알고리즘의 측쇄 구조 예측 결과입니다. 별표는 평균 충돌 값이 해당 기본 구조(CASP13의 경우 34.6, 2.2, 0.5, CASP14의 경우 40.0, 2.7, 0.7)보다 낮음을 나타냅니다.
개별 로타머 라이브러리와 계산 비용이 많이 드는 구조 검색 및 샘플링 단계를 혁신적으로 버리고 메인 체인 3D 기하학을 직접 결합하여 모든 사이드 체인 좌표를 병렬로 계산합니다. AttnPacker는 딥러닝 기반 방식인 DLPacker와 기존 컴퓨팅 방식 기반인 RosettaPacker에 비해 컴퓨팅 효율성을 대폭 향상시키고 추론 시간을 100배 이상 단축했습니다.
다양한 PSCP 방법의 시간 비교. 모든 83개 CASP13 표적 단백질에 대한 측쇄 원자의 상대적 시간을 재구성합니다.
AttnPacker는 단백질 디자인에서도 똑같이 뛰어난 성능을 발휘합니다. Molecular Heart는 현재의 최첨단 방법에 필적하는 기본 시퀀스 복구 속도를 달성하는 동시에 매우 정확한 어셈블리를 생산하는 공동 설계를 위해 AttnPacker 변형을 교육했습니다. Rosetta 시뮬레이션 검증에서는 AttnPacker가 설계한 구조가 일반적으로 하위(낮은) Rosetta 에너지를 생성한다는 것을 보여줍니다.
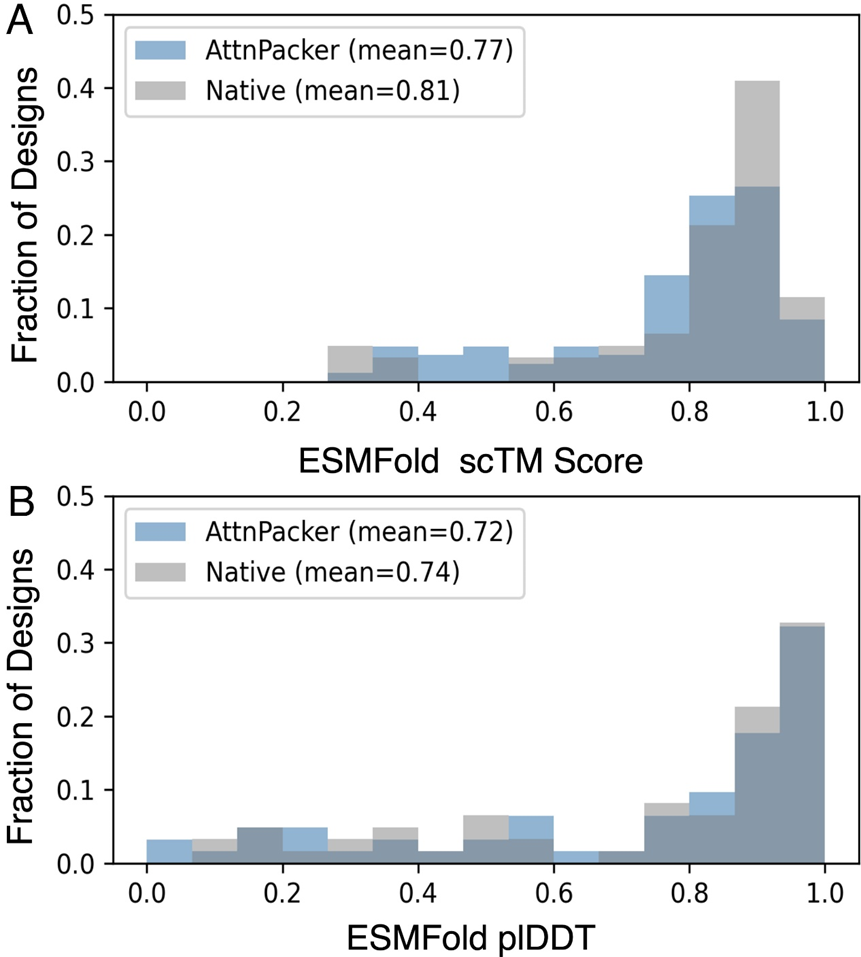
AttnPacker 생성 품질을 평가하기 위해 ESMFold scTM 및 plDDT 측정항목을 사용하여 AttnPacker에서 생성된 시퀀스와 네이티브 단백질 시퀀스를 비교한 결과 강한 상관관계가 나타났습니다.
AttnPaker는 놀라운 효과와 효율성 외에도 매우 실용적인 가치도 가지고 있습니다. 사용이 매우 쉽습니다. AttnPaker를 실행하려면 단백질 구조 파일만 필요합니다. 대조적으로, OPUS-Rota4(28)는 DLPacker의 원자 환경에 대한 복셀 표현, trRosetta100의 논리, 2차 구조, OPUS-CM 출력의 제약 파일이 필요합니다. 또한 AttnPacker는 측쇄 좌표를 직접 예측하기 때문에 출력이 완전히 미분 가능하여 최적화 또는 단백질-단백질 상호 작용과 같은 다운스트림 예측 작업을 용이하게 합니다. Xu Jinbo 교수는 "우수한 예측 효과, 고효율 및 사용 용이성 등의 장점은 AttnPacker를 연구 및 산업 분야에서 널리 사용하는 데 도움이 됩니다."라고 말했습니다.
Summary
1. AttnPacker는 서열 및 측쇄 좌표를 직접 예측하는 데 사용되는 선구적인 작업입니다. 일하다.
2. AttnPacker의 정확도는 다른 방법보다 우수하고 효율성이 크게 향상되었으며 사용이 매우 쉽습니다.
위 내용은 세계 최초: 단백질 측쇄 예측 및 서열 설계 문제를 극복하기 위한 Molecular Heart의 오픈 소스 새로운 AI 알고리즘의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!

핫 AI 도구

Undresser.AI Undress
사실적인 누드 사진을 만들기 위한 AI 기반 앱

AI Clothes Remover
사진에서 옷을 제거하는 온라인 AI 도구입니다.

Undress AI Tool
무료로 이미지를 벗다

Clothoff.io
AI 옷 제거제

AI Hentai Generator
AI Hentai를 무료로 생성하십시오.

인기 기사

뜨거운 도구

메모장++7.3.1
사용하기 쉬운 무료 코드 편집기

SublimeText3 중국어 버전
중국어 버전, 사용하기 매우 쉽습니다.

스튜디오 13.0.1 보내기
강력한 PHP 통합 개발 환경

드림위버 CS6
시각적 웹 개발 도구

SublimeText3 Mac 버전
신 수준의 코드 편집 소프트웨어(SublimeText3)

뜨거운 주제
 7561
7561
 15
15
 1384
1384
 52
52
 84
84
 11
11
 60
60
 19
19
 28
28
 98
98
 Centos Shutdown 명령 줄
Apr 14, 2025 pm 09:12 PM
Centos Shutdown 명령 줄
Apr 14, 2025 pm 09:12 PM
CentOS 종료 명령은 종료이며 구문은 종료 [옵션] 시간 [정보]입니다. 옵션은 다음과 같습니다. -H 시스템 중지 즉시 옵션; -P 종료 후 전원을 끕니다. -R 다시 시작; -대기 시간. 시간은 즉시 (현재), 분 (분) 또는 특정 시간 (HH : MM)으로 지정할 수 있습니다. 추가 정보는 시스템 메시지에 표시 될 수 있습니다.
 Centos HDFS 구성을 확인하는 방법
Apr 14, 2025 pm 07:21 PM
Centos HDFS 구성을 확인하는 방법
Apr 14, 2025 pm 07:21 PM
CentOS 시스템에서 HDFS 구성 확인에 대한 완전한 안내서이 기사에서는 CentOS 시스템에서 HDF의 구성 및 실행 상태를 효과적으로 확인하는 방법을 안내합니다. 다음 단계는 HDF의 설정 및 작동을 완전히 이해하는 데 도움이됩니다. Hadoop 환경 변수 확인 : 먼저 Hadoop 환경 변수가 올바르게 설정되어 있는지 확인하십시오. 터미널에서 다음 명령을 실행하여 Hadoop이 올바르게 설치되고 구성되었는지 확인하십시오. Hadoopversion Check HDFS 구성 파일 : HDFS의 Core 구성 파일은/etc/hadoop/conf/directory에 있으며 Core-Site.xml 및 HDFS-Site.xml이 중요합니다. 사용
 Centos에서 Gitlab의 백업 방법은 무엇입니까?
Apr 14, 2025 pm 05:33 PM
Centos에서 Gitlab의 백업 방법은 무엇입니까?
Apr 14, 2025 pm 05:33 PM
CentOS 시스템 하에서 Gitlab의 백업 및 복구 정책 데이터 보안 및 복구 가능성을 보장하기 위해 CentOS의 Gitlab은 다양한 백업 방법을 제공합니다. 이 기사는 완전한 GITLAB 백업 및 복구 전략을 설정하는 데 도움이되는 몇 가지 일반적인 백업 방법, 구성 매개 변수 및 복구 프로세스를 자세히 소개합니다. 1. 수동 백업 gitlab-rakegitlab : 백업 : 명령을 작성하여 수동 백업을 실행하십시오. 이 명령은 gitlab 저장소, 데이터베이스, 사용자, 사용자 그룹, 키 및 권한과 같은 주요 정보를 백업합니다. 기본 백업 파일은/var/opt/gitlab/backups 디렉토리에 저장됩니다. /etc /gitlab을 수정할 수 있습니다
 Centos에서 Pytorch에 대한 GPU 지원은 어떻습니까?
Apr 14, 2025 pm 06:48 PM
Centos에서 Pytorch에 대한 GPU 지원은 어떻습니까?
Apr 14, 2025 pm 06:48 PM
CentOS 시스템에서 Pytorch GPU 가속도를 활성화하려면 Cuda, Cudnn 및 GPU 버전의 Pytorch를 설치해야합니다. 다음 단계는 프로세스를 안내합니다. CUDA 및 CUDNN 설치 CUDA 버전 호환성 결정 : NVIDIA-SMI 명령을 사용하여 NVIDIA 그래픽 카드에서 지원하는 CUDA 버전을보십시오. 예를 들어, MX450 그래픽 카드는 CUDA11.1 이상을 지원할 수 있습니다. Cudatoolkit 다운로드 및 설치 : NVIDIACUDATOOLKIT의 공식 웹 사이트를 방문하여 그래픽 카드에서 지원하는 가장 높은 CUDA 버전에 따라 해당 버전을 다운로드하여 설치하십시오. CUDNN 라이브러리 설치 :
 Docker 원리에 대한 자세한 설명
Apr 14, 2025 pm 11:57 PM
Docker 원리에 대한 자세한 설명
Apr 14, 2025 pm 11:57 PM
Docker는 Linux 커널 기능을 사용하여 효율적이고 고립 된 응용 프로그램 실행 환경을 제공합니다. 작동 원리는 다음과 같습니다. 1. 거울은 읽기 전용 템플릿으로 사용되며, 여기에는 응용 프로그램을 실행하는 데 필요한 모든 것을 포함합니다. 2. Union 파일 시스템 (Unionfs)은 여러 파일 시스템을 스택하고 차이점 만 저장하고 공간을 절약하고 속도를 높입니다. 3. 데몬은 거울과 컨테이너를 관리하고 클라이언트는 상호 작용을 위해 사용합니다. 4. 네임 스페이스 및 CGroup은 컨테이너 격리 및 자원 제한을 구현합니다. 5. 다중 네트워크 모드는 컨테이너 상호 연결을 지원합니다. 이러한 핵심 개념을 이해 함으로써만 Docker를 더 잘 활용할 수 있습니다.
 Centos 설치 MySQL
Apr 14, 2025 pm 08:09 PM
Centos 설치 MySQL
Apr 14, 2025 pm 08:09 PM
Centos에 MySQL을 설치하려면 다음 단계가 필요합니다. 적절한 MySQL Yum 소스 추가. mysql 서버를 설치하려면 yum install mysql-server 명령을 실행하십시오. mysql_secure_installation 명령을 사용하여 루트 사용자 비밀번호 설정과 같은 보안 설정을 작성하십시오. 필요에 따라 MySQL 구성 파일을 사용자 정의하십시오. MySQL 매개 변수를 조정하고 성능을 위해 데이터베이스를 최적화하십시오.
 Centos에서 Gitlab 로그를 보는 방법
Apr 14, 2025 pm 06:18 PM
Centos에서 Gitlab 로그를 보는 방법
Apr 14, 2025 pm 06:18 PM
Centos 시스템에서 Gitlab 로그를보기위한 완전한 안내서이 기사에서는 메인 로그, 예외 로그 및 기타 관련 로그를 포함한 CentOS 시스템에서 다양한 Gitlab 로그를 보는 방법을 안내합니다. 로그 파일 경로는 Gitlab 버전 및 설치 방법에 따라 다를 수 있습니다. 다음 경로가 존재하지 않으면 GitLab 설치 디렉토리 및 구성 파일을 확인하십시오. 1. 기본 gitlab 로그보기 다음 명령을 사용하여 Gitlabrails 응용 프로그램의 기본 로그 파일을 보려면 다음 명령 : 명령 : sudocat/var/log/gitlab/gitlab-rails/production.log이 명령은 제품을 표시합니다.
 Centos에서 Pytorch의 분산 교육을 운영하는 방법
Apr 14, 2025 pm 06:36 PM
Centos에서 Pytorch의 분산 교육을 운영하는 방법
Apr 14, 2025 pm 06:36 PM
CentOS 시스템에 대한 Pytorch 분산 교육에는 다음 단계가 필요합니다. Pytorch 설치 : 전제는 Python과 PIP가 CentOS 시스템에 설치된다는 것입니다. CUDA 버전에 따라 Pytorch 공식 웹 사이트에서 적절한 설치 명령을 받으십시오. CPU 전용 교육의 경우 다음 명령을 사용할 수 있습니다. PipinStalltorchtorchvisiontorchaudio GPU 지원이 필요한 경우 CUDA 및 CUDNN의 해당 버전이 설치되어 있는지 확인하고 해당 PyTorch 버전을 설치하려면 설치하십시오. 분산 환경 구성 : 분산 교육에는 일반적으로 여러 기계 또는 단일 기계 다중 GPU가 필요합니다. 장소




