 Peranti teknologi
Peranti teknologi
 AI
AI
 Sumber terbuka! Cina Hong Kong, MIT, dan Fudan mencadangkan model asas RNA yang pertama
Sumber terbuka! Cina Hong Kong, MIT, dan Fudan mencadangkan model asas RNA yang pertama
Sumber terbuka! Cina Hong Kong, MIT, dan Fudan mencadangkan model asas RNA yang pertama
Berbeza dengan bidang protein, penyelidikan dalam bidang RNA selalunya kekurangan data anotasi yang mencukupi Contohnya, data 3D hanya mempunyai lebih daripada 1,000 RNA. Ini sangat mengehadkan pembangunan kaedah pembelajaran mesin dalam tugas ramalan fungsi struktur RNA.
Untuk mengimbangi kekurangan data beranotasi, Artikel ini menunjukkan model asas yang boleh memberikan pengetahuan struktur dan fungsi yang kaya untuk pelbagai kajian RNA - RNA model asas ( RNA-FM). Sebagai model asas RNA pertama di dunia dilatih dengan cara tanpa pengawasan berdasarkan 23 juta jujukan RNA tidak berlabel, RNA-FM melombong corak evolusi dan struktur yang terkandung dalam jujukan RNA.
Perlu diperhatikan bahawa RNA-FM hanya perlu memadankan model hiliran yang mudah atau hanya menyediakan pembenaman, dan ia boleh mencapai prestasi yang jauh melebihi SOTA dalam banyak tugas hiliran, seperti Ia boleh dipertingkatkan sebanyak 20% dalam ramalan struktur sekunder dan 30% dalam ramalan peta jarak. Eksperimen berskala besar telah membuktikan bahawa model ini sangat boleh digeneralisasikan malah boleh digunakan untuk COVID-19 dan serpihan pengawalseliaan mRNA.

- Pracetak kertas: https://arxiv org /abs/2204.00300
- Kod dan model: https://github.com/ml4bio/RNA-FM
- Pelayan: https://proj.cse.cuhk.edu.hk/rnafm
Pengenalan
Dalam beberapa tahun kebelakangan ini, kaedah pengkomputeran biologi berdasarkan pembelajaran mendalam telah mencapai kemajuan yang cemerlang dalam bidang protein Pencapaian yang paling terkenal ialah rangka kerja ramalan struktur 3D protein hujung ke hujung AlphaFold2 yang dibangunkan oleh pasukan Google DeepMind. Walau bagaimanapun, protein hanyalah satu jenis daripada banyak molekul biologi (DNA/RNA), sebagai sumber penghasilan protein, mengandungi lebih banyak maklumat asas daripada yang terakhir dan mempunyai nilai penyelidikan yang lebih penting.
Secara umumnya, protein ialah produk terjemahan daripada RNA yang digunakan untuk pengekodan, iaitu mRNA tetap boleh diterjemahkan ke dalam urutan protein tetap. Malah, bahagian pengekodan RNA ini hanya menyumbang 2% daripada semua urutan RNA, dan baki 98% ialah RNA bukan pengekodan (ncRNA). Walaupun ncRNA tidak "diterjemahkan" secara langsung kepada protein, ia terlipat menjadi struktur tertier dengan fungsi tertentu dan memainkan peranan pengawalseliaan dalam proses terjemahan mRNA atau fungsi biologi yang lain. Oleh itu, menganalisis struktur dan fungsi ncRNA adalah penyelidikan yang lebih asas dan kompleks daripada analisis protein.
Walau bagaimanapun, berbanding medan protein di mana kaedah pengiraan lebih matang, ramalan struktur dan fungsi berasaskan RNA masih dalam peringkat awal, dan kaedah pengiraan pada asalnya digunakan untuk medan protein adalah juga sukar untuk dipindahkan secara langsung ke medan RNA. Had utama kaedah pengiraan ini ialah anotasi data RNA biasanya sukar diperoleh, dan ia memerlukan banyak sumber percubaan dan masa untuk melengkapkan anotasi sejumlah kecil data Kebanyakan kaedah pengiraan memerlukan sejumlah besar data beranotasi untuk penyeliaan untuk mencapai prestasi tinggi. Walaupun tidak banyak data beranotasi, medan RNA sebenarnya telah mengumpul banyak data jujukan yang tidak bernotasi. Kaedah artikel ini adalah menggunakan data tidak berlabel ini untuk menyediakan maklumat tambahan yang berkesan untuk pelbagai tugas hiliran.
Berdasarkan pertimbangan ini, Pasukan Makmal Kecerdasan Buatan Hong Kong, MIT, Fudan dan Shanghai mencadangkan kaedah tanpa pengawasan kepada Yayasan RNA model (RNA-FM) dilatih pada 23 juta jujukan RNA tulen tanpa label. Walaupun data tidak memberikan maklumat anotasi semasa proses latihan, RNA-FM masih menambang corak evolusi dan struktur yang terkandung dalam jujukan RNA ini dengan cara yang tidak diawasi.
Jika RNA-FM boleh digunakan dengan berkesan pada struktur RNA hiliran dan tugas ramalan fungsi, kaedah pengiraan ini pasti akan mendapat manfaat daripada pengetahuan yang didorong oleh RNA-FM dan mencapai peningkatan prestasi prestasi yang lebih baik. Rangka kerja pra-latihan huluan dan migrasi hiliran dan aplikasi RNA-FM ditunjukkan dalam rajah di bawah.
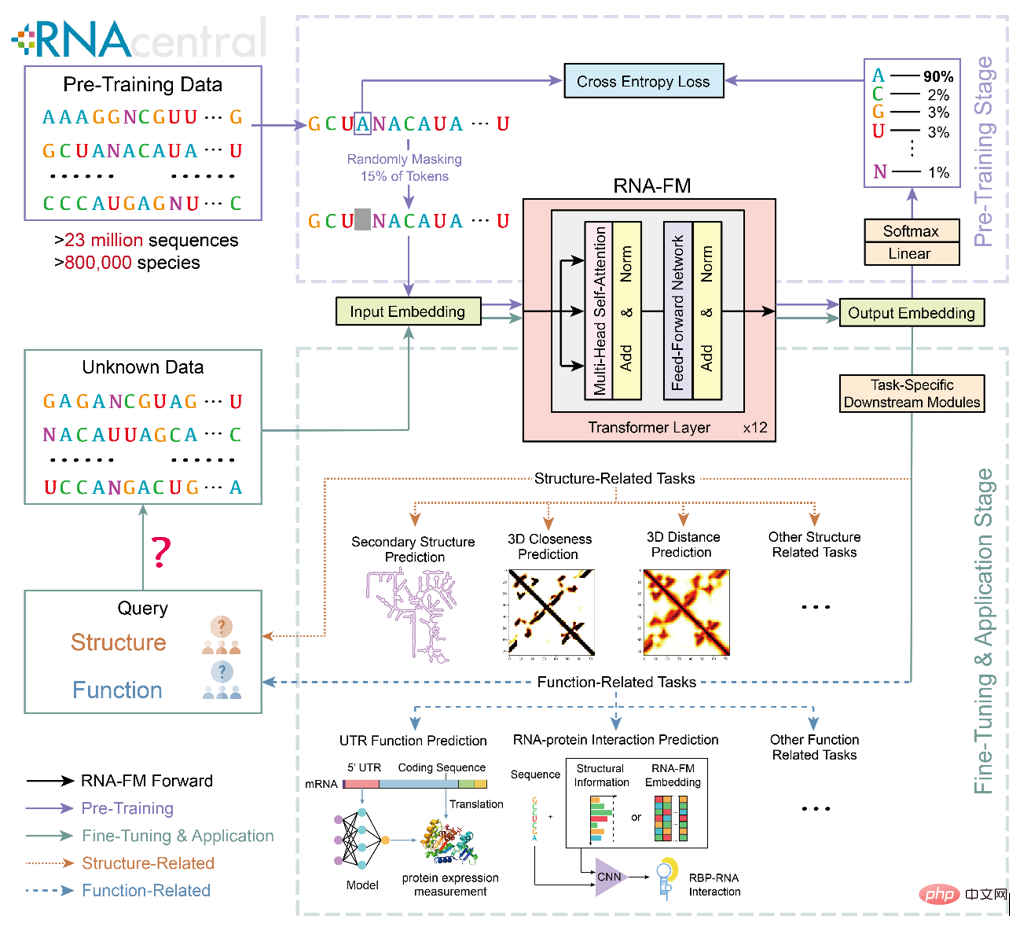
Gambaran Keseluruhan Penyelidikan
Untuk mengesahkan sama ada RNA-FM pra-latihan telah mempelajari "pengetahuan" daripada sejumlah besar data tidak berlabel dan Apakah jenis "pengetahuan" yang telah dipelajari? Artikel menjalankan satu siri analisis mengenai pembenaman.
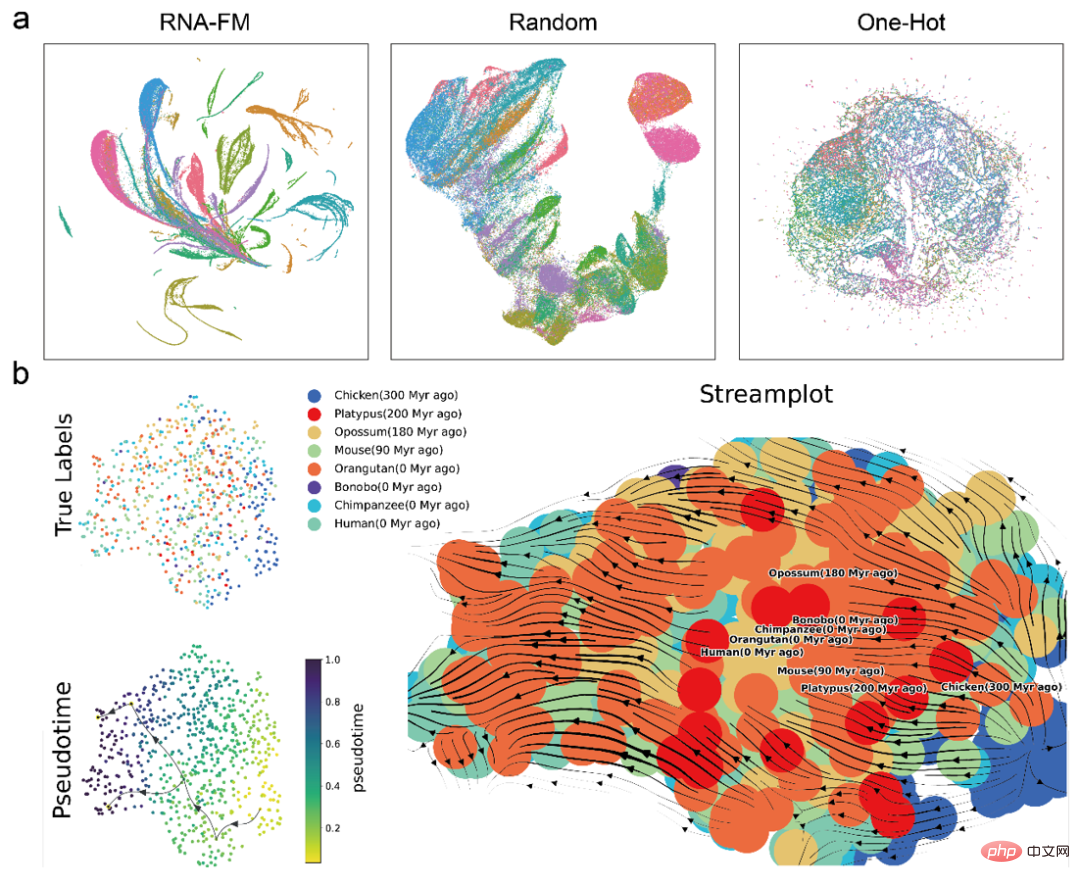
Pertama ialah perbandingan pengelompokan mudah pelbagai ciri secara terus melalui UMAP , dan didapati bahawa pembenaman daripada RNA pra-latihan -FM lebih baik daripada yang lain. Membenamkan membentuk kelompok dengan spesies RNA yang lebih berbeza. Ini bermakna pembenaman RNA-FM memang mengandungi maklumat struktur atau berfungsi untuk membezakan spesies RNA.
Kemudian, Artikel itu juga menggunakan inferens trajektori (inferens Trajektori) untuk meramalkan evolusi lncRNA daripada spesies yang berbeza melalui pembenaman RNA-FM . Daripada streamplot di bawah, ramalan masa pseudo evolusi antara spesies adalah kira-kira konsisten dengan maklumat evolusi spesies sebenar, menunjukkan bahawa pembenaman RNA-FM juga mengandungi sebahagian daripada maklumat evolusi.
Perlu diambil perhatian bahawa sama ada maklumat komuniti spesies RNA atau maklumat evolusi lncRNA, RNA-FM tidak terdedah secara langsung kepada label ini semasa latihan. RNA-FM menemui corak yang berkaitan dengan struktur, fungsi dan evolusi daripada jujukan tulen dengan cara yang diselia sendiri sepenuhnya .
Lebih banyak hasil eksperimen
Selain menganalisis secara langsung pembenaman RNA-FM , artikel itu juga cuba memperkenalkan RNA-FM kepada pelbagai tugas ramalan struktur RNA hiliran, termasuk struktur sekunder, ramalan kenalan, ramalan jarak dan ramalan struktur tertier, dan telah mencapai hasil yang jelas .
Terutama dalam ramalan struktur sekunder, artikel itu menggunakan RNA-FM sebagai tulang belakang dan hanya rangkaian ResNet yang ringkas sebagai model hiliran, mengatasi dua set data awam yang lain kaedah -of-the-art adalah lebih baik daripada UFold terbaik dengan 3-5 mata peratusan dalam F1score Dalam perbandingan head-to-head dengan UFold, RNA-FM berprestasi lebih baik dalam kebanyakan kategori RNA. Jika RNA-FM digabungkan dengan E2Efold, peningkatan prestasi 5% lagi boleh dicapai.

Untuk mengesahkan nilai aplikasi praktikal model, artikel Menggunakan RNA-FM untuk menjalankan analisis lengkap data COVID-19, termasuk menggunakan RNA-FM untuk meramal dengan tepat elemen pengawalseliaan utama dalam genom rujukan COVID-19 (29870 nt), dan menggunakan pembenaman RNA-FM untuk meramalkan secara kasar arah aliran evolusi COVID-19 utama -19 varian. 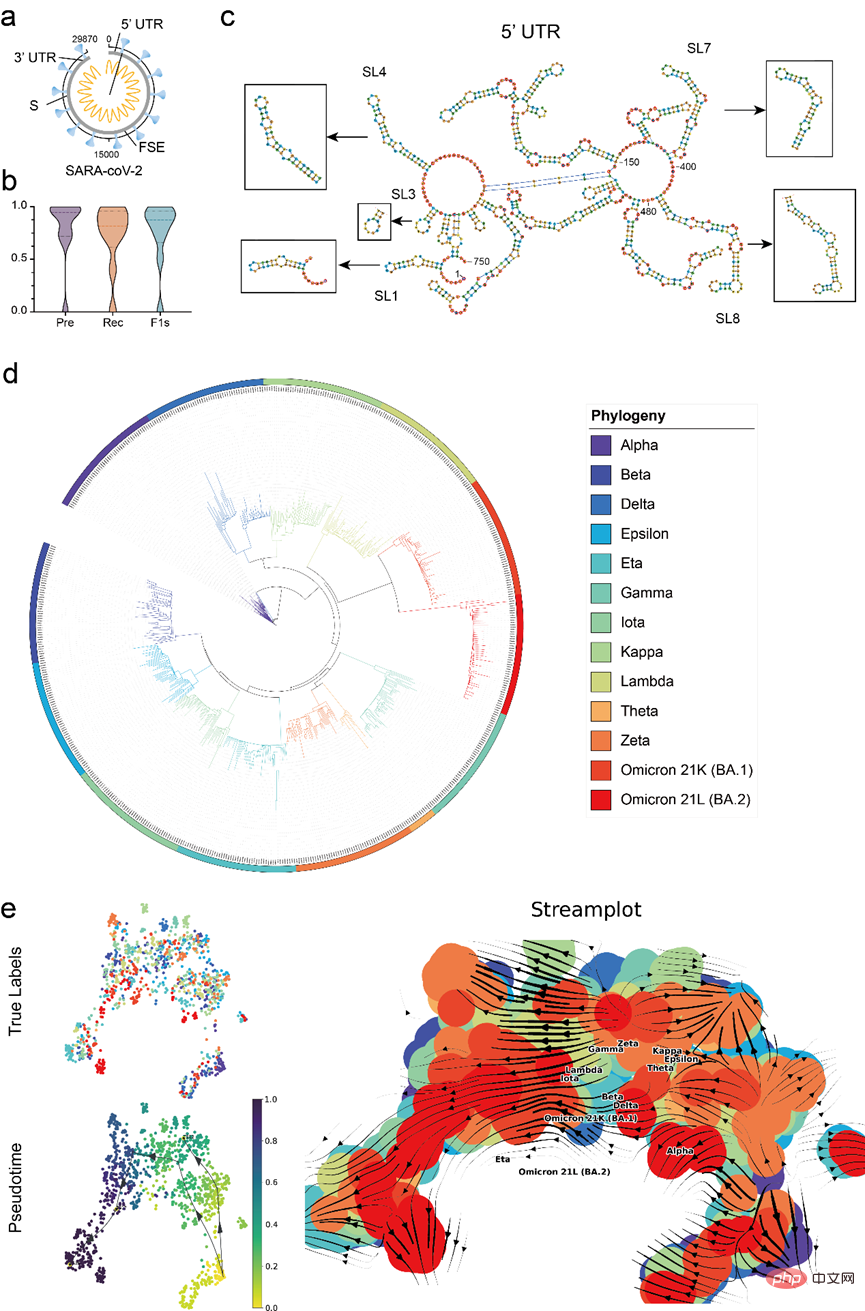
Secara umumnya, struktur molekul menentukan fungsinya Memandangkan RNA-FM boleh menyelesaikan tugas ramalan struktur RNA dengan cemerlang, adakah mungkin untuk menggunakan RNA-FM untuk juga meningkatkan ramalan fungsi?
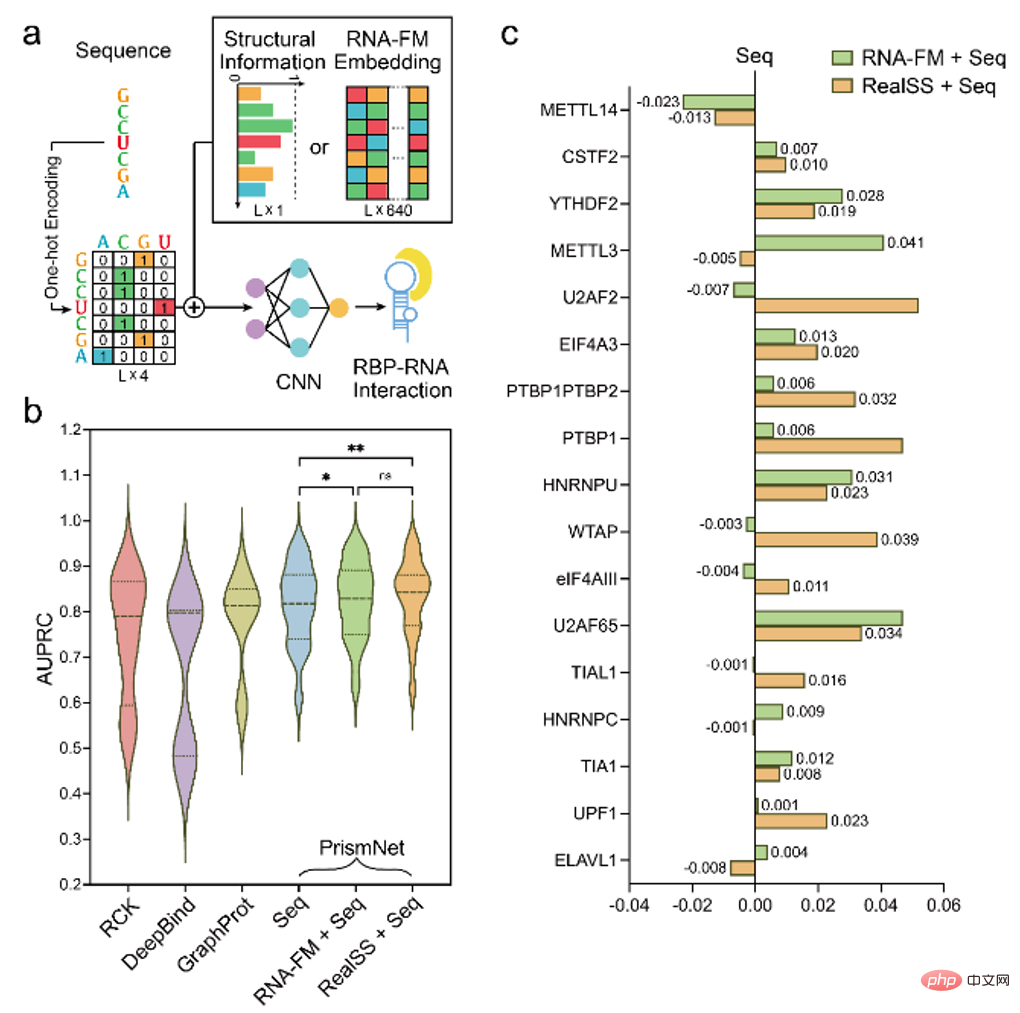
Oleh itu, artikel seterusnya cuba memperkenalkan RNA-FM ke dalam tugas ramalan fungsi RNA hiliran , seperti menggunakan pembenaman RNA-FM. Ramalan peranan RNA-protein.
Percubaan telah membuktikan bahawa pengenalan pembenaman RNA-FM telah meningkatkan prestasi model, dan dalam beberapa kes ia telah mencapai hasil ramalan yang sepadan dengan maklumat struktur sekunder sebenar sebagai input.
Untuk meneroka sama ada RNA-FM berdasarkan latihan ncRNA boleh digeneralisasikan kepada RNA lain, artikel akhirnya mencuba untuk menggunakan RNA -FM melaksanakan ramalan fungsi ekspresi protein berdasarkan 5'UTR pada mRNA. Walaupun mRNA bukan milik ncRNA, 5'UTR di atasnya adalah rantau yang tidak diterjemahkan tetapi mempunyai fungsi pengawalseliaan, yang konsisten dengan ciri-ciri ncRNA dan tidak muncul dalam data latihan.
Seperti yang anda lihat daripada rajah di bawah, model yang menyertakan pembenaman RNA-FM sentiasa lebih baik daripada model yang tidak. Walaupun peningkatan dalam prestasi agak terhad, ia sebahagiannya menunjukkan bahawa RNA-FM juga mempunyai keupayaan generalisasi tertentu pada data bukan ncRNA.

Kesimpulan
Secara amnya, artikel ini menggunakan data jujukan RNA yang tidak berlabel untuk pra-melatih model bahasa RNA-FM, dan melalui kaedah langsung atau tidak langsung, satu siri pengesahan Komprehensif struktur atau berfungsi pada tugas yang berbeza membuktikan bahawa RNA-FM sememangnya boleh meningkatkan prestasi kaedah pengkomputeran dalam tugas hiliran dengan berkesan.
Kemunculan RNA-FM telah mengurangkan keadaan semasa data berlabel RNA pada tahap tertentu, dan menyediakan penyelidik lain antara muka yang mudah untuk mengakses kumpulan besar data tidak berlabel, yang akan Sebagai model asas dalam bidang RNA, ia memberikan sokongan dan bantuan yang kuat untuk pelbagai penyelidikan dalam bidang ini.
Mengenai pengarang
Artikel ini mempunyai dua pengarang bersama pertama. Chen Jiayang ialah pembantu penyelidik di Universiti Cina Hong Kong. Hu Zhihang ialah calon kedoktoran di Universiti Cina Hong Kong.
Terdapat dua pengarang yang sepadan untuk artikel ini. Sun Siqi, penyelidik muda di Makmal Sistem Kompleks Pintar Universiti Fudan dan Makmal Kepintaran Buatan Shanghai, halaman utama https://intersun.github.io.
Li Yu, Penolong Profesor di Universiti China Hong Kong, Penolong Profesor Pelawat di MIT James Collins Lab, Saintis Penyelidikan di Institut Broad MIT dan Harvard, Sarjana Pelawat di Institut Wyss di Universiti Harvard, senarai Forbes 30 Bawah 30 Asia–Kelas 2022, Penjagaan Kesihatan & Sains. Laman utama: https://liyu95.com.
Atas ialah kandungan terperinci Sumber terbuka! Cina Hong Kong, MIT, dan Fudan mencadangkan model asas RNA yang pertama. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!

Alat AI Hot

Undresser.AI Undress
Apl berkuasa AI untuk mencipta foto bogel yang realistik

AI Clothes Remover
Alat AI dalam talian untuk mengeluarkan pakaian daripada foto.

Undress AI Tool
Gambar buka pakaian secara percuma

Clothoff.io
Penyingkiran pakaian AI

AI Hentai Generator
Menjana ai hentai secara percuma.

Artikel Panas

Alat panas

Notepad++7.3.1
Editor kod yang mudah digunakan dan percuma

SublimeText3 versi Cina
Versi Cina, sangat mudah digunakan

Hantar Studio 13.0.1
Persekitaran pembangunan bersepadu PHP yang berkuasa

Dreamweaver CS6
Alat pembangunan web visual

SublimeText3 versi Mac
Perisian penyuntingan kod peringkat Tuhan (SublimeText3)

Topik panas
 1379
1379
 52
52
 Model MoE sumber terbuka paling berkuasa di dunia ada di sini, dengan keupayaan bahasa Cina setanding dengan GPT-4, dan harganya hanya hampir satu peratus daripada GPT-4-Turbo
May 07, 2024 pm 04:13 PM
Model MoE sumber terbuka paling berkuasa di dunia ada di sini, dengan keupayaan bahasa Cina setanding dengan GPT-4, dan harganya hanya hampir satu peratus daripada GPT-4-Turbo
May 07, 2024 pm 04:13 PM
Bayangkan model kecerdasan buatan yang bukan sahaja mempunyai keupayaan untuk mengatasi pengkomputeran tradisional, tetapi juga mencapai prestasi yang lebih cekap pada kos yang lebih rendah. Ini bukan fiksyen sains, DeepSeek-V2[1], model MoE sumber terbuka paling berkuasa di dunia ada di sini. DeepSeek-V2 ialah gabungan model bahasa pakar (MoE) yang berkuasa dengan ciri-ciri latihan ekonomi dan inferens yang cekap. Ia terdiri daripada 236B parameter, 21B daripadanya digunakan untuk mengaktifkan setiap penanda. Berbanding dengan DeepSeek67B, DeepSeek-V2 mempunyai prestasi yang lebih kukuh, sambil menjimatkan 42.5% kos latihan, mengurangkan cache KV sebanyak 93.3% dan meningkatkan daya pemprosesan penjanaan maksimum kepada 5.76 kali. DeepSeek ialah sebuah syarikat yang meneroka kecerdasan buatan am
 Apr 09, 2024 am 11:52 AM
Apr 09, 2024 am 11:52 AM
AI memang mengubah matematik. Baru-baru ini, Tao Zhexuan, yang telah mengambil perhatian terhadap isu ini, telah memajukan keluaran terbaru "Buletin Persatuan Matematik Amerika" (Buletin Persatuan Matematik Amerika). Memfokuskan pada topik "Adakah mesin akan mengubah matematik?", ramai ahli matematik menyatakan pendapat mereka Seluruh proses itu penuh dengan percikan api, tegar dan menarik. Penulis mempunyai barisan yang kuat, termasuk pemenang Fields Medal Akshay Venkatesh, ahli matematik China Zheng Lejun, saintis komputer NYU Ernest Davis dan ramai lagi sarjana terkenal dalam industri. Dunia AI telah berubah secara mendadak Anda tahu, banyak artikel ini telah dihantar setahun yang lalu.
 Google gembira: prestasi JAX mengatasi Pytorch dan TensorFlow! Ia mungkin menjadi pilihan terpantas untuk latihan inferens GPU
Apr 01, 2024 pm 07:46 PM
Google gembira: prestasi JAX mengatasi Pytorch dan TensorFlow! Ia mungkin menjadi pilihan terpantas untuk latihan inferens GPU
Apr 01, 2024 pm 07:46 PM
Prestasi JAX, yang dipromosikan oleh Google, telah mengatasi Pytorch dan TensorFlow dalam ujian penanda aras baru-baru ini, menduduki tempat pertama dalam 7 penunjuk. Dan ujian tidak dilakukan pada TPU dengan prestasi JAX terbaik. Walaupun dalam kalangan pembangun, Pytorch masih lebih popular daripada Tensorflow. Tetapi pada masa hadapan, mungkin lebih banyak model besar akan dilatih dan dijalankan berdasarkan platform JAX. Model Baru-baru ini, pasukan Keras menanda aras tiga hujung belakang (TensorFlow, JAX, PyTorch) dengan pelaksanaan PyTorch asli dan Keras2 dengan TensorFlow. Pertama, mereka memilih satu set arus perdana
 Hello, Atlas elektrik! Robot Boston Dynamics hidup semula, gerakan pelik 180 darjah menakutkan Musk
Apr 18, 2024 pm 07:58 PM
Hello, Atlas elektrik! Robot Boston Dynamics hidup semula, gerakan pelik 180 darjah menakutkan Musk
Apr 18, 2024 pm 07:58 PM
Boston Dynamics Atlas secara rasmi memasuki era robot elektrik! Semalam, Atlas hidraulik hanya "menangis" menarik diri daripada peringkat sejarah Hari ini, Boston Dynamics mengumumkan bahawa Atlas elektrik sedang berfungsi. Nampaknya dalam bidang robot humanoid komersial, Boston Dynamics berazam untuk bersaing dengan Tesla. Selepas video baharu itu dikeluarkan, ia telah pun ditonton oleh lebih sejuta orang dalam masa sepuluh jam sahaja. Orang lama pergi dan peranan baru muncul. Ini adalah keperluan sejarah. Tidak dinafikan bahawa tahun ini adalah tahun letupan robot humanoid. Netizen mengulas: Kemajuan robot telah menjadikan majlis pembukaan tahun ini kelihatan seperti manusia, dan tahap kebebasan adalah jauh lebih besar daripada manusia Tetapi adakah ini benar-benar bukan filem seram? Pada permulaan video, Atlas berbaring dengan tenang di atas tanah, seolah-olah terlentang. Apa yang berikut adalah rahang-jatuh
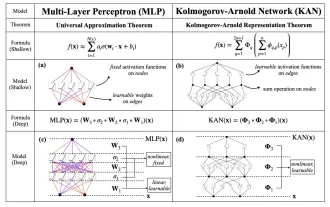 KAN, yang menggantikan MLP, telah diperluaskan kepada konvolusi oleh projek sumber terbuka
Jun 01, 2024 pm 10:03 PM
KAN, yang menggantikan MLP, telah diperluaskan kepada konvolusi oleh projek sumber terbuka
Jun 01, 2024 pm 10:03 PM
Awal bulan ini, penyelidik dari MIT dan institusi lain mencadangkan alternatif yang sangat menjanjikan kepada MLP - KAN. KAN mengatasi MLP dari segi ketepatan dan kebolehtafsiran. Dan ia boleh mengatasi prestasi MLP berjalan dengan bilangan parameter yang lebih besar dengan bilangan parameter yang sangat kecil. Sebagai contoh, penulis menyatakan bahawa mereka menggunakan KAN untuk menghasilkan semula keputusan DeepMind dengan rangkaian yang lebih kecil dan tahap automasi yang lebih tinggi. Khususnya, MLP DeepMind mempunyai kira-kira 300,000 parameter, manakala KAN hanya mempunyai kira-kira 200 parameter. KAN mempunyai asas matematik yang kukuh seperti MLP berdasarkan teorem penghampiran universal, manakala KAN berdasarkan teorem perwakilan Kolmogorov-Arnold. Seperti yang ditunjukkan dalam rajah di bawah, KAN telah
 Disyorkan: Projek pengesanan dan pengecaman muka sumber terbuka JS yang sangat baik
Apr 03, 2024 am 11:55 AM
Disyorkan: Projek pengesanan dan pengecaman muka sumber terbuka JS yang sangat baik
Apr 03, 2024 am 11:55 AM
Teknologi pengesanan dan pengecaman muka adalah teknologi yang agak matang dan digunakan secara meluas. Pada masa ini, bahasa aplikasi Internet yang paling banyak digunakan ialah JS Melaksanakan pengesanan muka dan pengecaman pada bahagian hadapan Web mempunyai kelebihan dan kekurangan berbanding dengan pengecaman muka bahagian belakang. Kelebihan termasuk mengurangkan interaksi rangkaian dan pengecaman masa nyata, yang sangat memendekkan masa menunggu pengguna dan meningkatkan pengalaman pengguna termasuk: terhad oleh saiz model, ketepatannya juga terhad. Bagaimana untuk menggunakan js untuk melaksanakan pengesanan muka di web? Untuk melaksanakan pengecaman muka di Web, anda perlu biasa dengan bahasa dan teknologi pengaturcaraan yang berkaitan, seperti JavaScript, HTML, CSS, WebRTC, dll. Pada masa yang sama, anda juga perlu menguasai visi komputer yang berkaitan dan teknologi kecerdasan buatan. Perlu diingat bahawa kerana reka bentuk bahagian Web
 Robot Tesla bekerja di kilang, Musk: Tahap kebebasan tangan akan mencapai 22 tahun ini!
May 06, 2024 pm 04:13 PM
Robot Tesla bekerja di kilang, Musk: Tahap kebebasan tangan akan mencapai 22 tahun ini!
May 06, 2024 pm 04:13 PM
Video terbaru robot Tesla Optimus dikeluarkan, dan ia sudah boleh berfungsi di kilang. Pada kelajuan biasa, ia mengisih bateri (bateri 4680 Tesla) seperti ini: Pegawai itu juga mengeluarkan rupanya pada kelajuan 20x - pada "stesen kerja" kecil, memilih dan memilih dan memilih: Kali ini ia dikeluarkan Salah satu sorotan video itu ialah Optimus menyelesaikan kerja ini di kilang, sepenuhnya secara autonomi, tanpa campur tangan manusia sepanjang proses. Dan dari perspektif Optimus, ia juga boleh mengambil dan meletakkan bateri yang bengkok, memfokuskan pada pembetulan ralat automatik: Berkenaan tangan Optimus, saintis NVIDIA Jim Fan memberikan penilaian yang tinggi: Tangan Optimus adalah robot lima jari di dunia paling cerdik. Tangannya bukan sahaja boleh disentuh
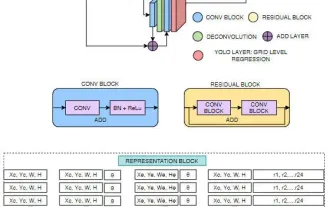 FisheyeDetNet: algoritma pengesanan sasaran pertama berdasarkan kamera fisheye
Apr 26, 2024 am 11:37 AM
FisheyeDetNet: algoritma pengesanan sasaran pertama berdasarkan kamera fisheye
Apr 26, 2024 am 11:37 AM
Pengesanan objek ialah masalah yang agak matang dalam sistem pemanduan autonomi, antaranya pengesanan pejalan kaki adalah salah satu algoritma terawal untuk digunakan. Penyelidikan yang sangat komprehensif telah dijalankan dalam kebanyakan kertas kerja. Walau bagaimanapun, persepsi jarak menggunakan kamera fisheye untuk pandangan sekeliling agak kurang dikaji. Disebabkan herotan jejari yang besar, perwakilan kotak sempadan standard sukar dilaksanakan dalam kamera fisheye. Untuk mengurangkan perihalan di atas, kami meneroka kotak sempadan lanjutan, elips dan reka bentuk poligon am ke dalam perwakilan kutub/sudut dan mentakrifkan metrik mIOU pembahagian contoh untuk menganalisis perwakilan ini. Model fisheyeDetNet yang dicadangkan dengan bentuk poligon mengatasi model lain dan pada masa yang sama mencapai 49.5% mAP pada set data kamera fisheye Valeo untuk pemanduan autonomi



