 Peranti teknologi
Peranti teknologi
 AI
AI
 Meta AI membuka 600 juta+ peta struktur protein metagenomik, dan 15 bilion model bahasa telah disiapkan dalam masa dua minggu
Meta AI membuka 600 juta+ peta struktur protein metagenomik, dan 15 bilion model bahasa telah disiapkan dalam masa dua minggu
Meta AI membuka 600 juta+ peta struktur protein metagenomik, dan 15 bilion model bahasa telah disiapkan dalam masa dua minggu
Tahun ini, DeepMind menerbitkan struktur ramalan kira-kira 220 juta protein, yang meliputi hampir semua protein organisma yang diketahui dalam pangkalan data DNA. Kini satu lagi gergasi teknologi, Meta, sedang mengisi kekosongan lain, iaitu mikrob.
Ringkasnya, Meta menggunakan teknologi AI untuk meramalkan struktur kira-kira 600 juta protein daripada bakteria dan mikroorganisma lain yang belum dicirikan. Ketua pasukan Alexander Rives berkata: "Protein ini adalah struktur yang paling kurang kita ketahui, dan ia adalah protein yang sangat misteri. Saya rasa penemuan ini memberikan potensi untuk pemahaman yang lebih mendalam tentang biologi."
Lazimnya, model bahasa dilatih pada jumlah teks yang banyak. Meta Untuk menggunakan model bahasa pada protein, Rives dan rakan sekerja mengambil urutan protein yang diketahui sebagai input, yang terdiri daripada 20 asid amino yang diwakili oleh huruf yang berbeza. Rangkaian kemudian belajar untuk melengkapkan protein secara automatik sambil menutup bahagian tertentu asid amino.
Meta menamakan rangkaian ini ESMFold. Walaupun ketepatan ramalan ESMFold tidak sebaik AlphaFold, ia adalah kira-kira 60 kali lebih pantas daripada AlphaFold dalam meramalkan struktur. Kelajuan ini bermakna ramalan struktur protein boleh ditingkatkan kepada pangkalan data yang lebih besar.

- Alamat kertas: https://www.biorxiv.org/content/10.1101/2022.07.20.500902v2
- Alamat projek: https://github.com/facebookresearch/esm
Sekarang, Sebagai ujian, Meta memutuskan untuk menggunakan model mereka pada pangkalan data DNA metagenomik, semuanya diperoleh daripada alam sekitar, termasuk tanah, air laut, usus manusia, kulit dan habitat mikrob lain. Meta AI mengumumkan pelancaran ESM Metagenomic Atlas yang mengandungi lebih daripada 600 juta protein, yang merupakan pandangan komprehensif pertama tentang "jirim gelap" alam semesta protein . Ia juga merupakan pangkalan data terbesar bagi struktur ramalan resolusi tinggi, 3 kali lebih besar daripada mana-mana pangkalan data struktur protein sedia ada, dan yang pertama menyediakan liputan komprehensif, berskala besar bagi protein metagenomik.
Secara keseluruhan, pasukan Meta meramalkan lebih daripada 617 juta struktur protein dalam masa dua minggu sahaja. Rives berkata ramalan itu percuma dan tersedia kepada sesiapa sahaja, sama seperti kod asas model itu.
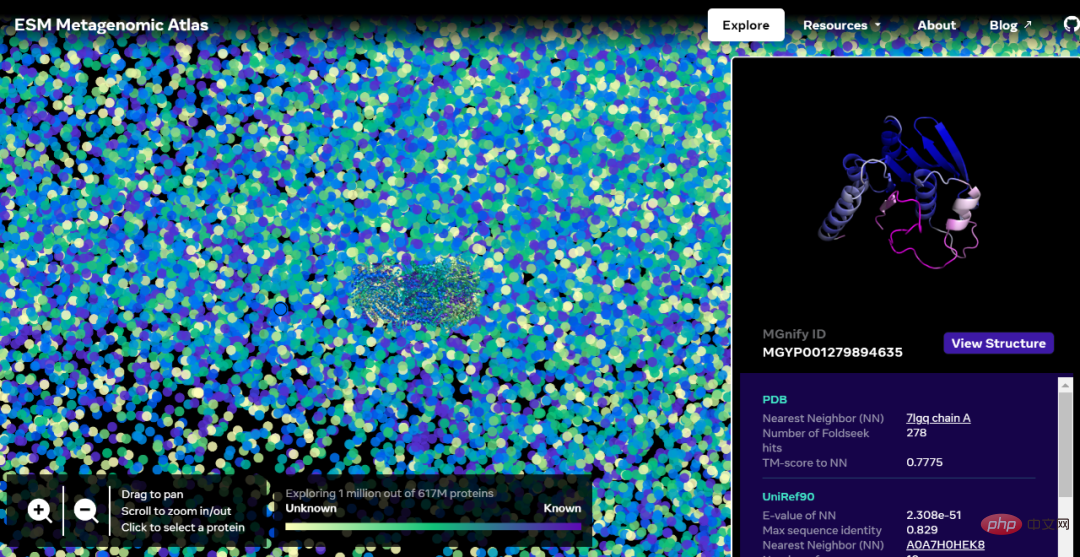
Alamat versi interaktif: https://esmatlas.com/explore?at=1%2C1%2C21.999999344348925
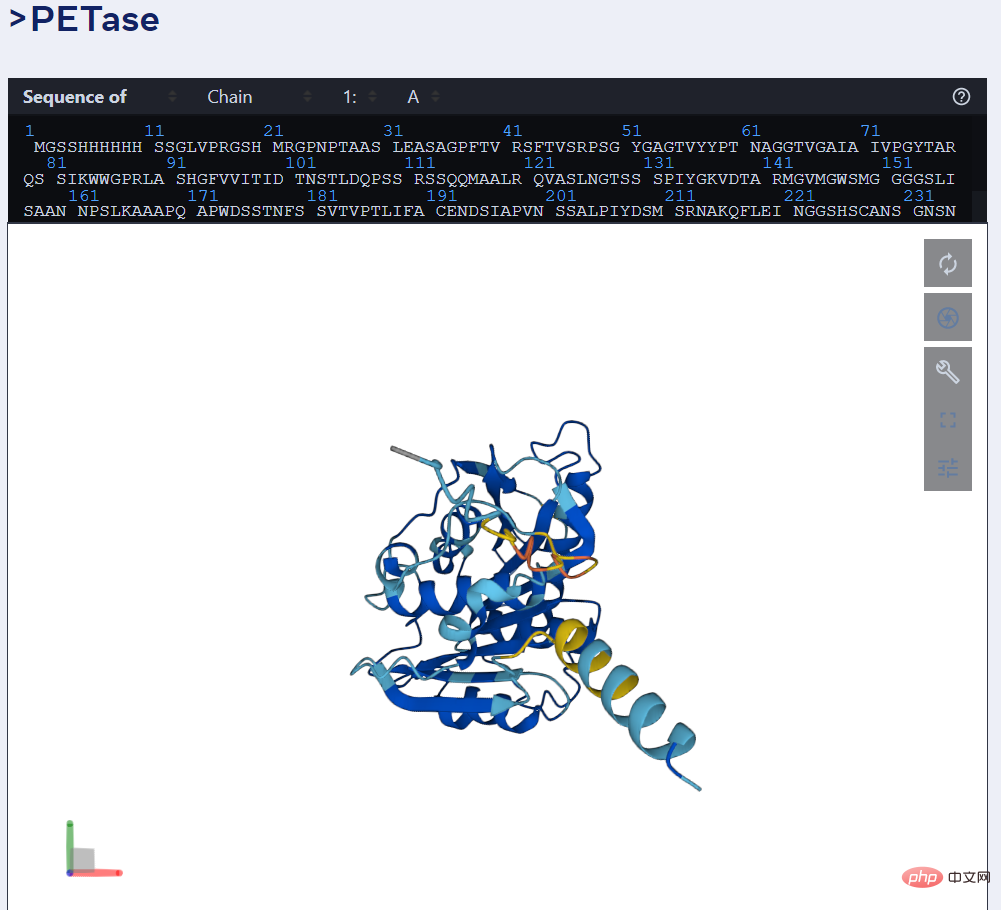
Sebagai contoh, gambar di bawah menunjukkan ramalan enzim PET oleh ESMFold.
Pengenalan
Seperti yang kita sedia maklum, protein adalah molekul kompleks dan dinamik yang dikodkan oleh gen dan bertanggungjawab terutamanya untuk proses asas kehidupan. Protein mempunyai peranan yang menakjubkan dalam biologi. Sebagai contoh, batang dan kon dalam mata manusia boleh merasakan cahaya, jadi kita boleh melihat dunia luar yang membentuk asas pendengaran dan sentuhan dalam tumbuhan yang menukar tenaga cahaya kepada molekul; yang mendorong mikroorganisma dan "motor" yang membuat otot manusia bergerak;
Pada tahun 1998, Jo Handelsman dari Jabatan Patologi Tumbuhan di Universiti Wisconsin mula-mula mencadangkan konsep metagenomik pada tahap tertentu, ia boleh dianggap sebagai idea penyelidikan dan analisis genom tunggal, dan nama Inggeris makro ialah meta-, yang juga diterjemahkan sebagai meta.
Metagenomics mendedahkan berbilion-bilion jujukan protein yang baru kepada sains dan dikatalogkan buat pertama kali oleh NCBI, Institut Bioinformatik Eropah dan dalam pangkalan data besar yang disusun oleh projek awam seperti Institut Genom Bersama .
Kaedah lipatan protein baharu yang dibangunkan oleh Meta AI yang memanfaatkan model bahasa yang besar untuk mencipta pandangan komprehensif pertama struktur protein dalam pangkalan data metagenomik (dengan ratusan juta protein). Meta mendapati bahawa model bahasa boleh meramalkan struktur tiga dimensi tahap atom protein 60 kali lebih cepat daripada kaedah ramalan struktur protein SOTA sedia ada. Kemajuan ini akan membantu mempercepatkan era baharu pemahaman struktur protein, yang membolehkan buat kali pertama memahami struktur berbilion-bilion protein yang dikatalogkan oleh teknologi penjujukan genetik.
Membuka dunia tersembunyi alam semula jadi: pandangan komprehensif pertama ruang struktur metagenomik
Kami tahu bahawa kemajuan dalam penjujukan genetik telah membolehkan analisis berbilion-bilion jujukan protein metagenomik Pengkatalogan menjadi mungkin. Tetapi secara eksperimen menentukan struktur 3D berbilion-bilion protein melangkaui skop teknik makmal intensif masa seperti kristalografi sinar-X, yang boleh mengambil masa beberapa minggu atau bahkan bertahun-tahun untuk mengesan satu protein. Pendekatan pengiraan boleh memberikan pandangan tentang protein metagenomik yang tidak mungkin menggunakan teknik eksperimen.
Pemetaan metagenomik ESM akan membolehkan saintis mencari dan menganalisis struktur protein metagenomik pada skala ratusan juta protein. Ini boleh membantu mengenal pasti struktur yang tidak dicirikan sebelum ini, mencari hubungan evolusi yang jauh dan menemui protein baharu yang boleh digunakan dalam perubatan dan aplikasi lain.
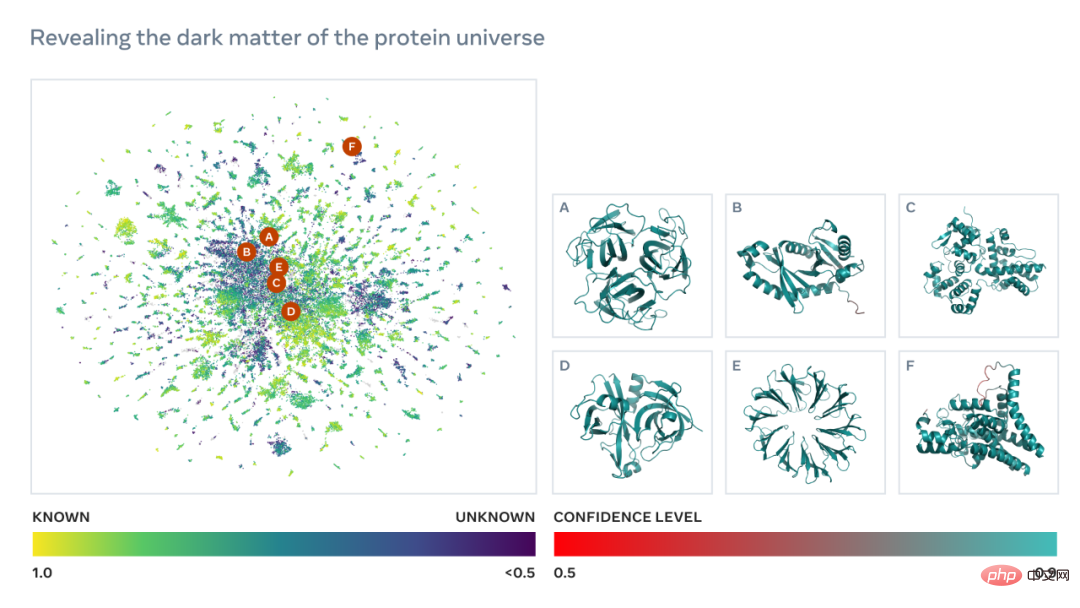
Berikut ialah peta yang mengandungi puluhan ribu ramalan berkeyakinan tinggi, menunjukkan persamaan dengan protein dengan struktur yang diketahui pada masa ini. Dan, buat pertama kalinya, imej itu menunjukkan kawasan ruang struktur protein yang lebih besar yang tidak diketahui sepenuhnya.
Belajar membaca bahasa biologi
Seperti yang ditunjukkan dalam rajah di bawah, model bahasa ESM-2 telah dilatih untuk meramal proses evolusi Asid amino bertopeng mengikut urutan. Meta AI mendapati bahawa, sebagai hasil latihan, maklumat tentang struktur protein muncul dalam keadaan dalaman model. Ini mengejutkan kerana model itu hanya dilatih mengikut urutan.
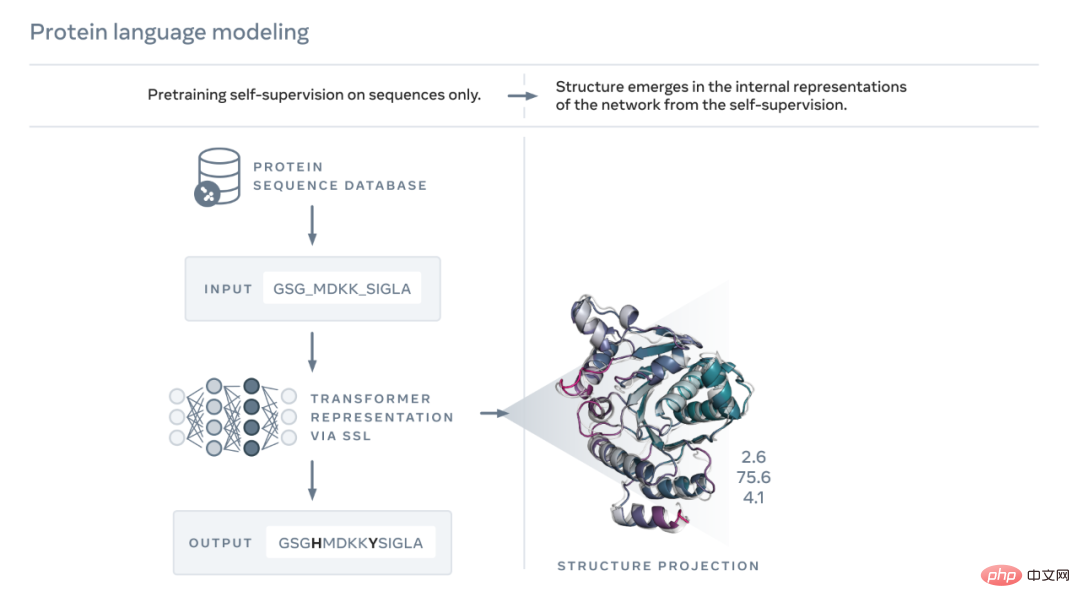
Sama seperti teks kertas atau surat, protein boleh ditulis sebagai jujukan aksara. Setiap watak sepadan dengan satu daripada 20 unsur kimia piawai (asid amino), masing-masing mempunyai sifat yang berbeza dan yang merupakan blok binaan protein. Blok binaan ini boleh disatukan dengan cara yang berbeza secara astronomi, contohnya untuk protein yang terdiri daripada 200 asid amino, terdapat 20^200 urutan yang mungkin, iaitu lebih daripada bilangan atom dalam alam semesta yang boleh dilihat. Setiap jujukan dilipat menjadi bentuk 3D (tetapi tidak semua jujukan dilipat menjadi struktur yang koheren, banyak yang dilipat menjadi bentuk tidak teratur), dan bentuk inilah yang menentukan sebahagian besar fungsi biologi protein.
Belajar membaca bahasa biologi memberikan cabaran yang hebat. Walaupun kedua-dua urutan protein dan petikan teks boleh ditulis sebagai aksara, terdapat perbezaan yang mendalam dan asas di antara mereka. Urutan protein menerangkan struktur kimia molekul yang dilipat menjadi bentuk 3D yang kompleks mengikut undang-undang fizik.
Jujukan protein mengandungi corak statistik yang menyampaikan maklumat tentang struktur lipatan protein. Sebagai contoh, jika dua kedudukan dalam protein berkembang bersama, atau dalam erti kata lain, jika asid amino tertentu berlaku pada satu kedudukan yang biasanya berpasangan dengan asid amino tertentu pada kedudukan yang lain, ini mungkin bermakna kedua-dua kedudukan berada dalam interaksi struktur terlipat. Ini serupa dengan dua keping teka-teki jigsaw, di mana evolusi mesti memilih asid amino yang sesuai bersama dalam struktur terlipat. Ini pula bermakna bahawa kita sering boleh membuat kesimpulan struktur protein dengan memerhati corak dalam urutannya.
ESM menggunakan AI untuk belajar membaca corak ini. Pada 2019, Meta AI memberikan bukti bahawa model bahasa mempelajari sifat protein, seperti struktur dan fungsinya. Melalui satu bentuk pembelajaran penyeliaan sendiri yang dipanggil pemodelan bahasa bertopeng, Meta AI melatih model bahasa mengenai jujukan berjuta-juta protein semula jadi. Menggunakan kaedah ini, model mesti mengisi celah dalam perenggan teks dengan betul, seperti "Ke _ atau tidak kepada , itu adalah _____".
Selepas itu, Meta AI melatih model bahasa untuk mengisi jurang dalam jujukan protein. Mereka mendapati bahawa maklumat tentang struktur dan fungsi protein muncul semasa latihan ini. Pada tahun 2020, Meta mengeluarkan model bahasa protein SOTA, ESM1b, untuk pelbagai aplikasi, termasuk membantu saintis meramalkan evolusi COVID-19 dan menemui punca genetik penyakit itu.
Kini, Meta AI telah memperluaskan pendekatan ini untuk mencipta model bahasa protein generasi seterusnya ESM-2, yang pada 15 bilion parameter merupakan model bahasa protein terbesar setakat ini. Mereka mendapati bahawa apabila parameter model ditingkatkan daripada 8 juta kepada 15 bilion, maklumat muncul dalam perwakilan dalaman, membolehkan ramalan struktur 3D pada resolusi atom.
Mencapai tertib pecutan magnitud dalam lipatan protein
Dalam rajah di bawah, apabila model diperbesarkan, struktur protein resolusi tinggi muncul. Pada masa yang sama, apabila model berskala, butiran baharu muncul dalam imej resolusi atom struktur protein.

Menggunakan alat pengiraan SOTA semasa, meramalkan struktur ratusan juta jujukan protein dalam rangka masa yang realistik akan mengambil masa bertahun-tahun, walaupun menggunakan penyelidikan utama institusi Begitu juga dengan sumber. Oleh itu, untuk membuat ramalan pada skala metagenomik, satu kejayaan dalam kelajuan ramalan adalah penting.
Meta AI mendapati bahawa menggunakan model bahasa bagi jujukan protein dengan ketara mempercepatkan ramalan struktur, sehingga 60 kali ganda. Ini mencukupi untuk membuat ramalan pada keseluruhan pangkalan data metagenomik dalam beberapa minggu sahaja dan boleh diskalakan kepada pangkalan data yang lebih besar daripada pangkalan data kami yang diterbitkan sekarang. Malah, keupayaan ramalan struktur baharu ini dapat meramalkan jujukan lebih daripada 600 juta protein metagenom dalam masa dua minggu sahaja pada kelompok kira-kira 2,000 GPU.
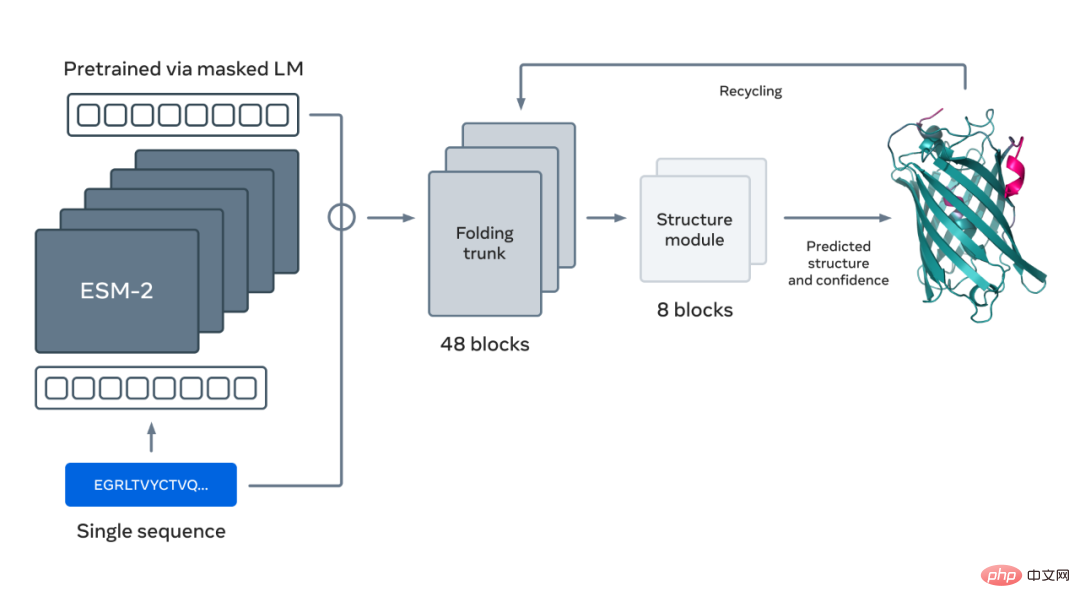
Selain itu, kaedah ramalan struktur SOTA semasa memerlukan pencarian pangkalan data protein yang besar untuk mengenal pasti jujukan yang berkaitan. Kaedah ini sebenarnya memerlukan keseluruhan set jujukan berkaitan evolusi sebagai input supaya ia boleh mengekstrak corak berkaitan struktur. Model bahasa ESM-2 Meta AI mempelajari corak evolusi ini semasa latihannya tentang jujukan protein, membolehkan ramalan resolusi tinggi struktur 3D terus daripada jujukan protein.
Rajah di bawah menunjukkan lipatan protein menggunakan model bahasa ESM-2. Anak panah dari kiri ke kanan menunjukkan aliran maklumat dalam rangkaian daripada model bahasa ke batang lipat ke modul struktur, dan akhirnya mengeluarkan koordinat dan keyakinan 3D.
Sila rujuk artikel asal untuk butiran lanjut.
Pautan blog: https://ai.facebook.com/blog/protein-folding-esmfold-metagenomics/
Atas ialah kandungan terperinci Meta AI membuka 600 juta+ peta struktur protein metagenomik, dan 15 bilion model bahasa telah disiapkan dalam masa dua minggu. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!

Alat AI Hot

Undresser.AI Undress
Apl berkuasa AI untuk mencipta foto bogel yang realistik

AI Clothes Remover
Alat AI dalam talian untuk mengeluarkan pakaian daripada foto.

Undress AI Tool
Gambar buka pakaian secara percuma

Clothoff.io
Penyingkiran pakaian AI

AI Hentai Generator
Menjana ai hentai secara percuma.

Artikel Panas

Alat panas

Notepad++7.3.1
Editor kod yang mudah digunakan dan percuma

SublimeText3 versi Cina
Versi Cina, sangat mudah digunakan

Hantar Studio 13.0.1
Persekitaran pembangunan bersepadu PHP yang berkuasa

Dreamweaver CS6
Alat pembangunan web visual

SublimeText3 versi Mac
Perisian penyuntingan kod peringkat Tuhan (SublimeText3)

Topik panas
 1377
1377
 52
52
 Cara Membuat Pangkalan Data Oracle Cara Membuat Pangkalan Data Oracle
Apr 11, 2025 pm 02:33 PM
Cara Membuat Pangkalan Data Oracle Cara Membuat Pangkalan Data Oracle
Apr 11, 2025 pm 02:33 PM
Mewujudkan pangkalan data Oracle tidak mudah, anda perlu memahami mekanisme asas. 1. Anda perlu memahami konsep pangkalan data dan Oracle DBMS; 2. Menguasai konsep teras seperti SID, CDB (pangkalan data kontena), PDB (pangkalan data pluggable); 3. Gunakan SQL*Plus untuk membuat CDB, dan kemudian buat PDB, anda perlu menentukan parameter seperti saiz, bilangan fail data, dan laluan; 4. Aplikasi lanjutan perlu menyesuaikan set aksara, memori dan parameter lain, dan melakukan penalaan prestasi; 5. Beri perhatian kepada ruang cakera, keizinan dan parameter, dan terus memantau dan mengoptimumkan prestasi pangkalan data. Hanya dengan menguasai ia dengan mahir memerlukan amalan yang berterusan, anda boleh benar -benar memahami penciptaan dan pengurusan pangkalan data Oracle.
 Cara Membuat Pangkalan Data Oracle Cara Membuat Pangkalan Data Oracle
Apr 11, 2025 pm 02:36 PM
Cara Membuat Pangkalan Data Oracle Cara Membuat Pangkalan Data Oracle
Apr 11, 2025 pm 02:36 PM
Untuk membuat pangkalan data Oracle, kaedah biasa adalah menggunakan alat grafik DBCA. Langkah -langkah adalah seperti berikut: 1. Gunakan alat DBCA untuk menetapkan DBName untuk menentukan nama pangkalan data; 2. Tetapkan SYSPASSWORD dan SYSTEMPASSWORD kepada kata laluan yang kuat; 3. Tetapkan aksara dan NationalCharacterset ke Al32utf8; 4. Tetapkan MemorySize dan Tablespacesize untuk menyesuaikan mengikut keperluan sebenar; 5. Tentukan laluan logfile. Kaedah lanjutan dibuat secara manual menggunakan arahan SQL, tetapi lebih kompleks dan terdedah kepada kesilapan. Perhatikan kekuatan kata laluan, pemilihan set aksara, saiz dan memori meja makan
 Cara Menulis Penyataan Pangkalan Data Oracle
Apr 11, 2025 pm 02:42 PM
Cara Menulis Penyataan Pangkalan Data Oracle
Apr 11, 2025 pm 02:42 PM
Inti dari pernyataan Oracle SQL adalah pilih, masukkan, mengemas kini dan memadam, serta aplikasi fleksibel dari pelbagai klausa. Adalah penting untuk memahami mekanisme pelaksanaan di sebalik pernyataan, seperti pengoptimuman indeks. Penggunaan lanjutan termasuk subqueries, pertanyaan sambungan, fungsi analisis, dan PL/SQL. Kesilapan umum termasuk kesilapan sintaks, isu prestasi, dan isu konsistensi data. Amalan terbaik pengoptimuman prestasi melibatkan menggunakan indeks yang sesuai, mengelakkan pilih *, mengoptimumkan di mana klausa, dan menggunakan pembolehubah terikat. Menguasai Oracle SQL memerlukan amalan, termasuk penulisan kod, debugging, berfikir dan memahami mekanisme asas.
 Cara Menambah, Ubah Suai dan Padam Panduan Operasi Lapangan Jadual MySQL Data
Apr 11, 2025 pm 05:42 PM
Cara Menambah, Ubah Suai dan Padam Panduan Operasi Lapangan Jadual MySQL Data
Apr 11, 2025 pm 05:42 PM
Panduan Operasi Lapangan di MySQL: Tambah, mengubah suai, dan memadam medan. Tambahkan medan: alter table table_name tambah column_name data_type [not null] [default default_value] [primary kekunci] [AUTO_INCREMENT] Modify Field: Alter Table Table_Name Ubah suai column_name data_type [not null] [default default_value] [Kunci Utama]
 Apakah kekangan integriti jadual pangkalan data Oracle?
Apr 11, 2025 pm 03:42 PM
Apakah kekangan integriti jadual pangkalan data Oracle?
Apr 11, 2025 pm 03:42 PM
Kekangan integriti pangkalan data Oracle dapat memastikan ketepatan data, termasuk: tidak null: nilai null dilarang; Unik: Keunikan menjamin, membolehkan nilai null tunggal; Kunci utama: kekangan utama utama, menguatkan unik, dan melarang nilai null; Kunci asing: Mengekalkan hubungan antara jadual, kunci asing merujuk kepada kunci utama jadual utama; Semak: Hadkan nilai lajur mengikut syarat.
 Penjelasan terperinci mengenai contoh pertanyaan bersarang dalam pangkalan data MySQL
Apr 11, 2025 pm 05:48 PM
Penjelasan terperinci mengenai contoh pertanyaan bersarang dalam pangkalan data MySQL
Apr 11, 2025 pm 05:48 PM
Pertanyaan bersarang adalah cara untuk memasukkan pertanyaan lain dalam satu pertanyaan. Mereka digunakan terutamanya untuk mendapatkan data yang memenuhi syarat kompleks, mengaitkan pelbagai jadual, dan mengira nilai ringkasan atau maklumat statistik. Contohnya termasuk mencari pekerja di atas gaji purata, mencari pesanan untuk kategori tertentu, dan mengira jumlah jumlah pesanan bagi setiap produk. Apabila menulis pertanyaan bersarang, anda perlu mengikuti: Tulis subqueries, tulis hasilnya kepada pertanyaan luar (dirujuk dengan alias atau sebagai klausa), dan mengoptimumkan prestasi pertanyaan (menggunakan indeks).
 Cara Mengkonfigurasi Format Log Debian Apache
Apr 12, 2025 pm 11:30 PM
Cara Mengkonfigurasi Format Log Debian Apache
Apr 12, 2025 pm 11:30 PM
Artikel ini menerangkan cara menyesuaikan format log Apache pada sistem Debian. Langkah -langkah berikut akan membimbing anda melalui proses konfigurasi: Langkah 1: Akses fail konfigurasi Apache Fail konfigurasi Apache utama sistem Debian biasanya terletak di /etc/apache2/apache2.conf atau /etc/apache2/httpd.conf. Buka fail konfigurasi dengan kebenaran root menggunakan arahan berikut: Sudonano/etc/Apache2/Apache2.conf atau Sudonano/etc/Apache2/httpd.conf Langkah 2: Tentukan format log tersuai untuk mencari atau
 Apa yang dilakukan Oracle
Apr 11, 2025 pm 06:06 PM
Apa yang dilakukan Oracle
Apr 11, 2025 pm 06:06 PM
Oracle adalah syarikat perisian Sistem Pengurusan Pangkalan Data (DBMS) terbesar di dunia. Produk utamanya termasuk fungsi berikut: Sistem Pengurusan Pengurusan Pangkalan Data Relasi (Oracle Database) Alat Pembangunan (Oracle Apex, Oracle Visual Builder) Middleware (Oracle Weblogic Server, Oracle SOA Suite) Analisis Awan (Oracle Cloud Infrastructure)



