 Peranti teknologi
Peranti teknologi
 AI
AI
 Pasukan China berjaya membangunkan AI untuk meramalkan ubat yang sesuai untuk pesakit kanser, dan hasilnya diterbitkan dalam sub-jurnal Nature
Pasukan China berjaya membangunkan AI untuk meramalkan ubat yang sesuai untuk pesakit kanser, dan hasilnya diterbitkan dalam sub-jurnal Nature
Pasukan China berjaya membangunkan AI untuk meramalkan ubat yang sesuai untuk pesakit kanser, dan hasilnya diterbitkan dalam sub-jurnal Nature
Dengan hanya satu AI, tindak balas klinikal 9,808 pesakit kanser terhadap ubat boleh diramalkan sepenuhnya.
Dan hasilnya konsisten dengan pemerhatian klinikal.
Ini adalah hasil terbaharu CODE-AE (pengekod auto penyahkeliruan konteks) yang dibawa oleh pasukan Lei Xie di City University of New York.

Ia mencadangkan model autopengekodan kontekstual novel yang boleh meramalkan tindak balas khusus pesakit yang berbeza terhadap ubat.
Ini akan memberi impak yang besar terhadap pembangunan ubat baharu dan ujian klinikal.
Anda tahu, di bawah model tradisional, ia mengambil masa hampir 10 tahun untuk membangunkan, menguji dan memasarkan sepenuhnya ubat baharu, dan dana yang digunakan adalah sangat besar, dengan mudah mencecah 1 bilion dolar A.S..
Kitaran ini begitu lama kerana tindak balas ubat baru dalam tubuh manusia sukar untuk diramalkan, dan ujian berulang sering diperlukan untuk ujian.
Jika AI boleh menggunakan data untuk membuat ramalan, ia akan memendekkan masa dengan ketara untuk memasarkan ubat baharu dan mengurangkan kos.
Pada masa ini, penyelidikan ini telah diterbitkan dalam sub-jurnal Alam "Nature Machine Intelligence".
Ringkasnya, CODE-AE menggunakan data daripada pengesahan sel in vitro ubat baharu untuk meramalkan tindak balas ubat dalam tubuh manusia.
Ini mengelakkan pergantungan latihan model AI pada data klinikal pesakit.
Sebab terbesar mengapa AI tidak begitu berkesan dalam ramalan tindak balas klinikal pada masa lalu ialah terlalu sukar untuk mengumpul data tindak balas klinikal yang besar dan berterusan.
Dari perspektif mekanisme, penyelidik membahagikan biomarker ubat kepada domain sumber dan domain sasaran.
Domain sumber mewakili domain yang berbeza daripada sampel ujian, tetapi mempunyai maklumat penyeliaan yang kaya, yang boleh difahami sebagai data pengesahan sel in vitro.
Domain sasaran ialah domain di mana sampel ujian berada Ia tidak mempunyai label atau hanya beberapa label, iaitu data pesakit.
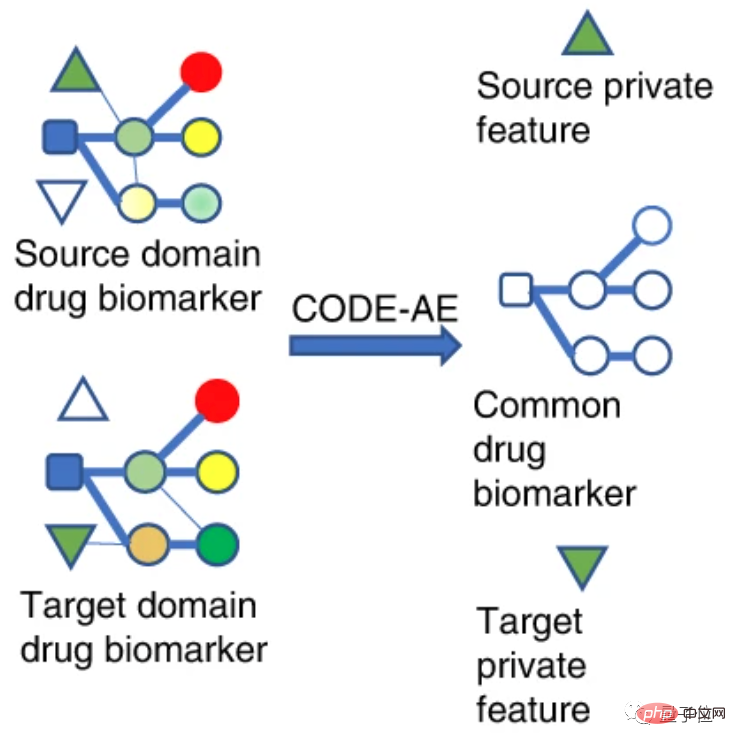
Petakan ciri data dalam medan berbeza ke dalam ruang ciri yang sama supaya jaraknya dalam ruang ini sedekat mungkin.
Jadi fungsi objektif yang dilatih pada domain sumber dalam ruang ciri boleh dipindahkan ke domain sasaran untuk meningkatkan ketepatan dalam domain sasaran.
Dalam konteks penyelidikan ini, kedua-dua domain sumber dan domain sasaran adalah ciri data biomarker ubat, iaitu ciri data sasaran dadah.
Melihat secara khusus rangka kerja model, ia terbahagi terutamanya kepada tiga bahagian: pra-latihan, penalaan halus dan inferens.
Pralatihan terutamanya menggunakan pembelajaran penyeliaan kendiri untuk membina modul pengekodan ciri untuk memetakan profil ekspresi gen yang tidak berlabel data sel in vitro dan data pesakit ke dalam ruang benam. Dengan cara ini, beberapa faktor yang mengelirukan boleh dihapuskan dan pengedaran terpendam kedua-dua data boleh konsisten untuk menghapuskan bias sistematik.
Peringkat penalaan halus adalah untuk menambah model diselia berdasarkan pra-latihan dan penggunaan data sel in vitro yang dilabelkan untuk latihan.
Akhir sekali, dalam peringkat inferens, pesakit yang diperolehi daripada pra-latihan mula-mula disambiguasi dan dibenamkan, dan kemudian model yang ditala digunakan untuk meramalkan tindak balas pesakit terhadap ubat tersebut.

Dalam mod ini, CODE-AE mempunyai dua ciri.
Pertama, ia boleh mengekstrak isyarat biologi biasa dan perwakilan peribadi dalam sampel yang tidak koheren, dengan itu menghapuskan gangguan yang disebabkan oleh corak data yang berbeza.
Kedua, selepas memisahkan isyarat tindak balas dadah dan faktor yang mengelirukan, penjajaran tempatan juga boleh dicapai.
Untuk meringkaskan, CODE-AE boleh difahami sebagai proses memilih ciri unik dalam ruang pembenaman corak data yang tidak koheren bagi data berlabel dan tidak berlabel.
Untuk menunjukkan keberkesanan model tersebut, para penyelidik meramalkan kesesuaian ubat bagi 9,808 pesakit kanser.
Sekiranya keputusan tapak yang diramalkan oleh model untuk keadaan pesakit berkaitan dengan sasaran ubat yang digunakannya, ia membuktikan ramalan itu betul.
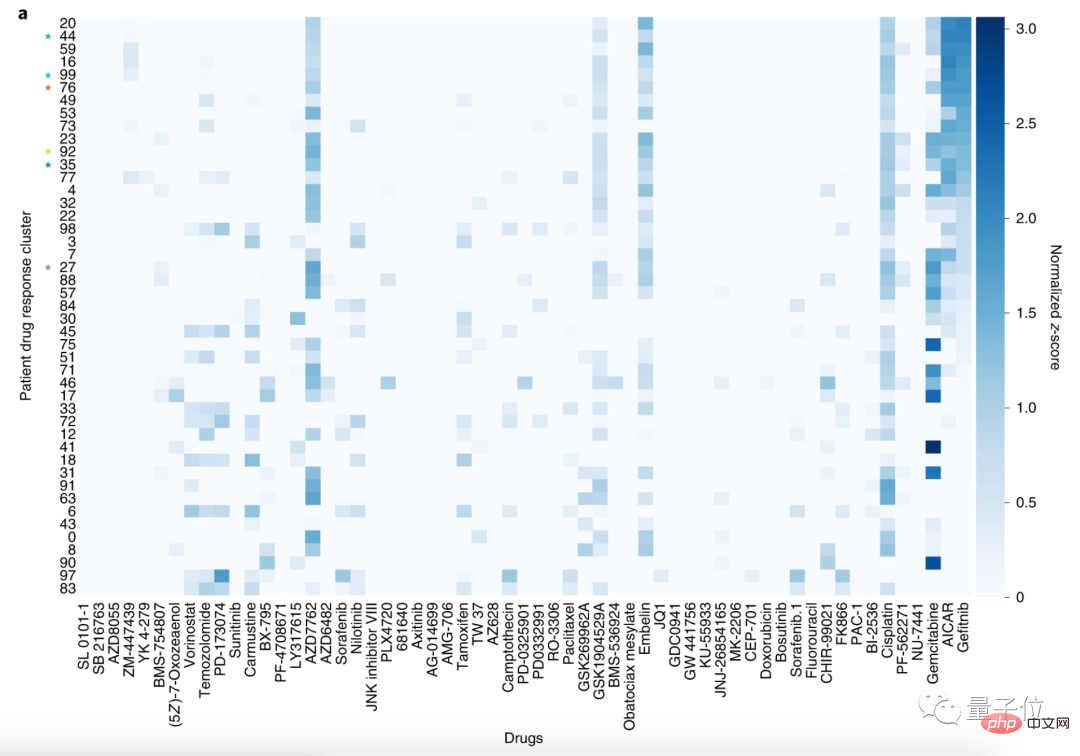
Para penyelidik kemudian membahagikan pesakit kepada 100 kelompok dan 59 ubat kepada 30 kelompok.
Dengan kaedah analisis ini, pesakit dengan profil tindak balas ubat yang serupa boleh dikumpulkan bersama.
Di sini, kami mengambil kluster pesakit dengan karsinoma sel skuamosa paru-paru (LSCC) dan kanser paru-paru bukan sel kecil (NSCLC) sebagai contoh.
Antara 59 ubat, ubat yang paling sensitif untuk LSCC ialah gefitinib, AICAR dan gemcitabine.
Gefitinib dan AICAR kedua-duanya menyasarkan reseptor faktor pertumbuhan epidermis (EGFR), dan gemcitabine sering digunakan untuk merawat kanser paru-paru bukan sel kecil tanpa mutasi EGFR.
Makalah itu menyatakan bahawa, selaras dengan mod tindakan ubat-ubatan ini, CODE-AE mendapati bahawa pesakit yang menggunakan gefitinib dan AICAR mempunyai profil tindak balas ubat yang serupa.
Dalam erti kata lain, CODE-AE telah menemui sasaran yang betul untuk rawatan pesakit, iaitu, ia boleh meramalkan ubat yang berkenaan.
Pasukan penyelidik di atas adalah dari City University of New York.
Pengarang yang sepadan ialah Lei Xie, yang lulus dari Universiti Sains dan Teknologi China dalam fizik polimer.
Berlulus dengan ijazah sarjana dalam sains komputer dari Universiti Rutgers;

Difahamkan bahawa langkah seterusnya pasukan penyelidik adalah untuk membangunkan fungsi ramalan CODE-AE untuk kepekatan dan metabolisme tindak balas klinikal ubat baharu.
Para penyelidik berkata bahawa model AI juga boleh disesuaikan untuk meramalkan kesan sampingan dadah pada tubuh manusia.
Perlu dinyatakan bahawa sub-jurnal Nature "Nature Machine Intelligence" memfokuskan secara khusus pada penyelidikan gunaan antara disiplin dalam kecerdasan buatan dan sains hayat, dengan purata bilangan kertas yang dikumpul setiap tahun kira-kira 60.
Alamat kertas: https://www.nature.com/articles/s42256-022-00541-0
Pautan rujukan: https://phys.org/news/2022-10 -ai-ubat-tindak balas-manusia dengan tepat.html
Atas ialah kandungan terperinci Pasukan China berjaya membangunkan AI untuk meramalkan ubat yang sesuai untuk pesakit kanser, dan hasilnya diterbitkan dalam sub-jurnal Nature. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!

Alat AI Hot

Undresser.AI Undress
Apl berkuasa AI untuk mencipta foto bogel yang realistik

AI Clothes Remover
Alat AI dalam talian untuk mengeluarkan pakaian daripada foto.

Undress AI Tool
Gambar buka pakaian secara percuma

Clothoff.io
Penyingkiran pakaian AI

AI Hentai Generator
Menjana ai hentai secara percuma.

Artikel Panas

Alat panas

Notepad++7.3.1
Editor kod yang mudah digunakan dan percuma

SublimeText3 versi Cina
Versi Cina, sangat mudah digunakan

Hantar Studio 13.0.1
Persekitaran pembangunan bersepadu PHP yang berkuasa

Dreamweaver CS6
Alat pembangunan web visual

SublimeText3 versi Mac
Perisian penyuntingan kod peringkat Tuhan (SublimeText3)

Topik panas
 1378
1378
 52
52
 Petua Konfigurasi Firewall Pelayan Mel Debian
Apr 13, 2025 am 11:42 AM
Petua Konfigurasi Firewall Pelayan Mel Debian
Apr 13, 2025 am 11:42 AM
Mengkonfigurasi firewall pelayan Mail Debian adalah langkah penting dalam memastikan keselamatan pelayan. Berikut adalah beberapa kaedah konfigurasi firewall yang biasa digunakan, termasuk penggunaan iptables dan firewalld. Gunakan iptables untuk mengkonfigurasi firewall untuk memasang iptables (jika belum dipasang): sudoapt-getupdateudoapt-getinstalliplesview peraturan iptables semasa: konfigurasi sudoiptable-l
 Cara Menetapkan Tahap Log Debian Apache
Apr 13, 2025 am 08:33 AM
Cara Menetapkan Tahap Log Debian Apache
Apr 13, 2025 am 08:33 AM
Artikel ini menerangkan cara menyesuaikan tahap pembalakan pelayan Apacheweb dalam sistem Debian. Dengan mengubah suai fail konfigurasi, anda boleh mengawal tahap maklumat log yang direkodkan oleh Apache. Kaedah 1: Ubah suai fail konfigurasi utama untuk mencari fail konfigurasi: Fail konfigurasi apache2.x biasanya terletak di direktori/etc/apache2/direktori. Nama fail mungkin apache2.conf atau httpd.conf, bergantung pada kaedah pemasangan anda. Edit Fail Konfigurasi: Buka Fail Konfigurasi dengan Kebenaran Root Menggunakan Editor Teks (seperti Nano): Sudonano/ETC/APACHE2/APACHE2.CONF
 Cara Mengoptimumkan Prestasi Debian Readdir
Apr 13, 2025 am 08:48 AM
Cara Mengoptimumkan Prestasi Debian Readdir
Apr 13, 2025 am 08:48 AM
Dalam sistem Debian, panggilan sistem Readdir digunakan untuk membaca kandungan direktori. Jika prestasinya tidak baik, cuba strategi pengoptimuman berikut: Memudahkan bilangan fail direktori: Split direktori besar ke dalam pelbagai direktori kecil sebanyak mungkin, mengurangkan bilangan item yang diproses setiap panggilan readdir. Dayakan Caching Kandungan Direktori: Bina mekanisme cache, kemas kini cache secara teratur atau apabila kandungan direktori berubah, dan mengurangkan panggilan kerap ke Readdir. Cafh memori (seperti memcached atau redis) atau cache tempatan (seperti fail atau pangkalan data) boleh dipertimbangkan. Mengamalkan struktur data yang cekap: Sekiranya anda melaksanakan traversal direktori sendiri, pilih struktur data yang lebih cekap (seperti jadual hash dan bukannya carian linear) untuk menyimpan dan mengakses maklumat direktori
 Cara Melaksanakan Penyortiran Fail oleh Debian Readdir
Apr 13, 2025 am 09:06 AM
Cara Melaksanakan Penyortiran Fail oleh Debian Readdir
Apr 13, 2025 am 09:06 AM
Dalam sistem Debian, fungsi Readdir digunakan untuk membaca kandungan direktori, tetapi urutan yang dikembalikannya tidak ditentukan sebelumnya. Untuk menyusun fail dalam direktori, anda perlu membaca semua fail terlebih dahulu, dan kemudian menyusunnya menggunakan fungsi QSORT. Kod berikut menunjukkan cara menyusun fail direktori menggunakan ReadDir dan QSORT dalam sistem Debian:#termasuk#termasuk#termasuk#termasuk // fungsi perbandingan adat, yang digunakan untuk qSortintCompare (Constvoid*A, Constvoid*b) {Returnstrcmp (*(*(*(*(*(*(*(*(*(*(*(*(*(*(*(*(*(
 Bagaimana Debian Readdir Bersepadu Dengan Alat Lain
Apr 13, 2025 am 09:42 AM
Bagaimana Debian Readdir Bersepadu Dengan Alat Lain
Apr 13, 2025 am 09:42 AM
Fungsi Readdir dalam sistem Debian adalah panggilan sistem yang digunakan untuk membaca kandungan direktori dan sering digunakan dalam pengaturcaraan C. Artikel ini akan menerangkan cara mengintegrasikan Readdir dengan alat lain untuk meningkatkan fungsinya. Kaedah 1: Menggabungkan Program Bahasa C dan Pipeline Pertama, tulis program C untuk memanggil fungsi Readdir dan output hasilnya:#termasuk#termasuk#includeintMain (intargc, char*argv []) {dir*dir; structdirent*entry; if (argc! = 2) {
 Kaedah pemasangan sijil SSL Server Server Debian
Apr 13, 2025 am 11:39 AM
Kaedah pemasangan sijil SSL Server Server Debian
Apr 13, 2025 am 11:39 AM
Langkah -langkah untuk memasang sijil SSL pada pelayan mel Debian adalah seperti berikut: 1. Pasang OpenSSL Toolkit terlebih dahulu, pastikan bahawa OpenSSL Toolkit telah dipasang pada sistem anda. Jika tidak dipasang, anda boleh menggunakan arahan berikut untuk memasang: sudoapt-getupdateudoapt-getinstallopenssl2. Menjana permintaan kunci dan sijil peribadi seterusnya, gunakan OpenSSL untuk menjana kunci peribadi RSA 2048-bit dan permintaan sijil (CSR): Membuka
 Bagaimana Debian OpenSSL Menghalang Serangan Man-dalam-Middle
Apr 13, 2025 am 10:30 AM
Bagaimana Debian OpenSSL Menghalang Serangan Man-dalam-Middle
Apr 13, 2025 am 10:30 AM
Dalam sistem Debian, OpenSSL adalah perpustakaan penting untuk pengurusan penyulitan, penyahsulitan dan sijil. Untuk mengelakkan serangan lelaki-dalam-pertengahan (MITM), langkah-langkah berikut boleh diambil: Gunakan HTTPS: Pastikan semua permintaan rangkaian menggunakan protokol HTTPS dan bukannya HTTP. HTTPS menggunakan TLS (Protokol Keselamatan Lapisan Pengangkutan) untuk menyulitkan data komunikasi untuk memastikan data tidak dicuri atau diganggu semasa penghantaran. Sahkan Sijil Pelayan: Sahkan secara manual Sijil Pelayan pada klien untuk memastikan ia boleh dipercayai. Pelayan boleh disahkan secara manual melalui kaedah perwakilan urlSession
 Cara Melakukan Pengurusan Log Debian Hadoop
Apr 13, 2025 am 10:45 AM
Cara Melakukan Pengurusan Log Debian Hadoop
Apr 13, 2025 am 10:45 AM
Menguruskan Log Hadoop pada Debian, anda boleh mengikuti langkah-langkah berikut dan amalan terbaik: Agregasi log membolehkan pengagregatan log: tetapkan benang.log-agregasi-enable untuk benar dalam fail benang-site.xml untuk membolehkan pengagregatan log. Konfigurasikan dasar pengekalan log: tetapkan yarn.log-aggregasi.Retain-seconds Untuk menentukan masa pengekalan log, seperti 172800 saat (2 hari). Nyatakan Laluan Penyimpanan Log: Melalui Benang



