 Peranti teknologi
Peranti teknologi
 AI
AI
 Pertama di dunia: Algoritma AI baharu sumber terbuka Molecular Heart untuk mengatasi masalah ramalan rantai sisi protein dan reka bentuk jujukan
Pertama di dunia: Algoritma AI baharu sumber terbuka Molecular Heart untuk mengatasi masalah ramalan rantai sisi protein dan reka bentuk jujukan
Pertama di dunia: Algoritma AI baharu sumber terbuka Molecular Heart untuk mengatasi masalah ramalan rantai sisi protein dan reka bentuk jujukan
Pembentukan struktur dan fungsi protein sebahagian besarnya bergantung kepada interaksi antara atom rantai sisi Oleh itu, ramalan rantai sisi protein (PSCP) yang tepat adalah kunci untuk menyelesaikan masalah ramalan struktur protein dan reka bentuk protein. Walau bagaimanapun, ramalan struktur protein sebelumnya kebanyakannya tertumpu pada struktur rantai utama, dan ramalan struktur rantai sisi sentiasa menjadi masalah sukar yang belum diselesaikan sepenuhnya.
Baru-baru ini, pasukan Xu Jinbo di Molecular Heart melancarkan AttnPacker seni bina PSCP baharu, yang telah mencapai peningkatan ketara dalam kelajuan, kecekapan memori dan ketepatan keseluruhannya pada masa ini. Algoritma ramalan struktur rantai juga merupakan algoritma AI pertama di dunia yang boleh meramal rantai sisi protein dan reka bentuk jujukan secara serentak.
Kertas kerja itu diterbitkan dalam Prosiding Akademi Sains Kebangsaan (PNAS), dan model pra-latihan, kod sumber dan skrip inferensnya telah bersumberkan terbuka di Github.

- Pautan kertas: https://www.pnas. org/doi/10.1073/pnas.2216438120#bahan-tambahan
- Pautan sumber terbuka: https://github.com/MattMcPartlon/AttnPacker Latar Belakang
Kebanyakan algoritma ramalan struktur protein semasa tertumpu terutamanya pada analisis struktur rantai utama, tetapi ramalan struktur rantai sisi protein masih merupakan masalah sukar yang belum dapat diatasi sepenuhnya. Sama ada algoritma ramalan struktur protein yang popular seperti AlphaFold2 atau algoritma yang memfokuskan pada ramalan struktur rantai sisi seperti DLPacker dan RosettaPacker, ketepatan atau kelajuannya tidak memuaskan. Ini juga mengenakan batasan pada reka bentuk protein.
Kaedah tradisional, seperti RosettaPacker, terutamanya menggunakan kaedah pengoptimuman tenaga, mula-mula mengumpulkan pengedaran atom rantai sisi, dan kemudian mencari pengelompokan rantai sampingan untuk asid amino tertentu untuk dicari gabungan tenaga minimum. Kaedah ini berbeza terutamanya daripada pilihan penyelidik pustaka pemutar, fungsi tenaga dan prosedur pengecilan tenaga, dengan ketepatan dihadkan oleh penggunaan heuristik carian dan prosedur pensampelan diskret. Terdapat juga kaedah ramalan rantaian sisi berdasarkan pembelajaran mendalam dalam industri, seperti DLPacker, yang merumuskan PSCP sebagai masalah penukaran imej-ke-imej dan menggunakan struktur model U-net. Walau bagaimanapun, ketepatan dan kelajuan ramalan masih tidak ideal.
Kaedah
AttnPacker ialah kaedah pembelajaran mendalam hujung ke hujung untuk meramal koordinat rantai sisi protein. Ia bersama-sama mensimulasikan interaksi rantai sisi, dengan struktur rantai sisi yang diramalkan secara langsung yang lebih boleh dilaksanakan secara fizikal, dengan perlanggaran atom yang lebih sedikit dan panjang dan sudut ikatan yang lebih ideal.Secara khusus, AttnPacker memperkenalkan seni bina penukar peta mendalam yang memanfaatkan aspek geometri dan perhubungan PSCP. Diilhamkan oleh AlphaFold2, Molecular Heart mencadangkan kemas kini segi tiga sedar kedudukan untuk mengoptimumkan ciri berpasangan menggunakan rangka kerja berasaskan graf untuk mengira perhatian segi tiga dan kemas kini berganda. Dengan pendekatan ini, AttnPacker mempunyai lebih sedikit memori dan model kapasiti yang lebih tinggi. Tambahan pula, Molecular Heart meneroka beberapa mekanisme perhatian setara SE (3) dan mencadangkan seni bina pengubah setara untuk belajar daripada titik 3D.
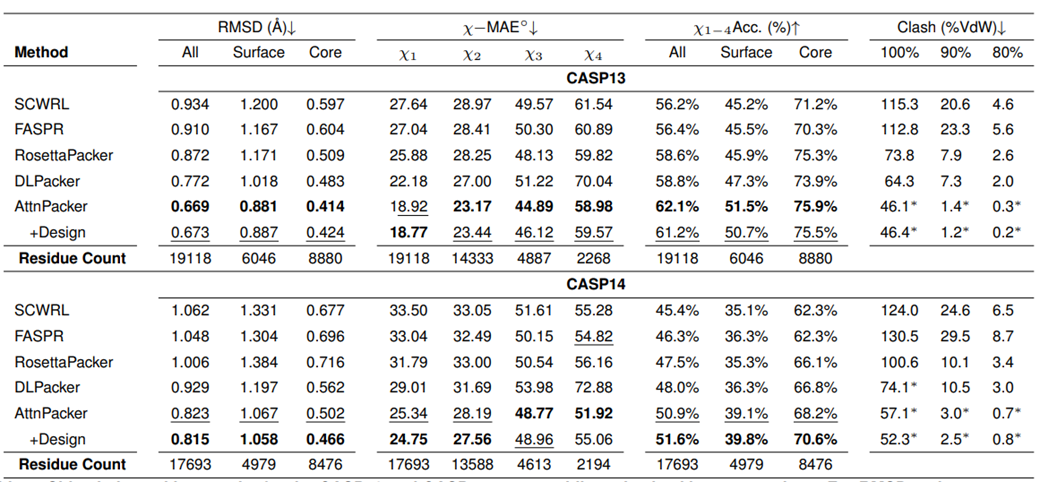
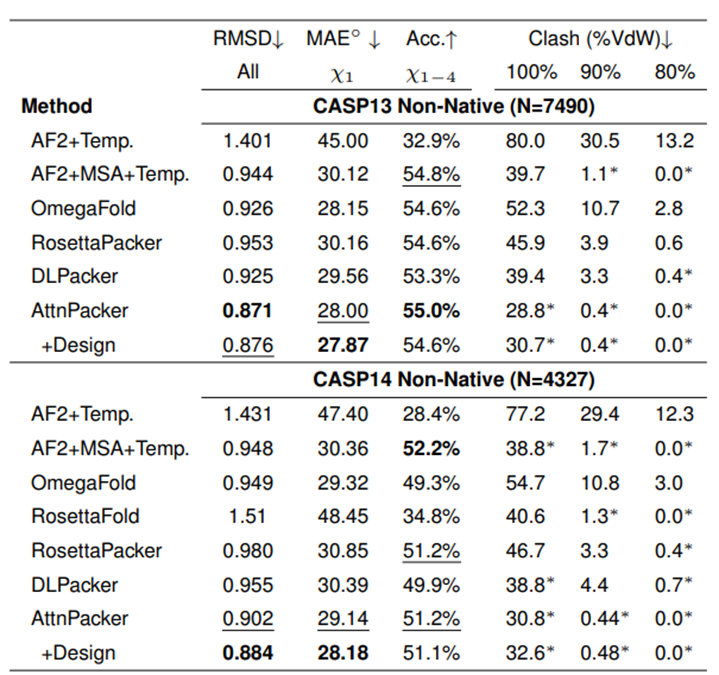
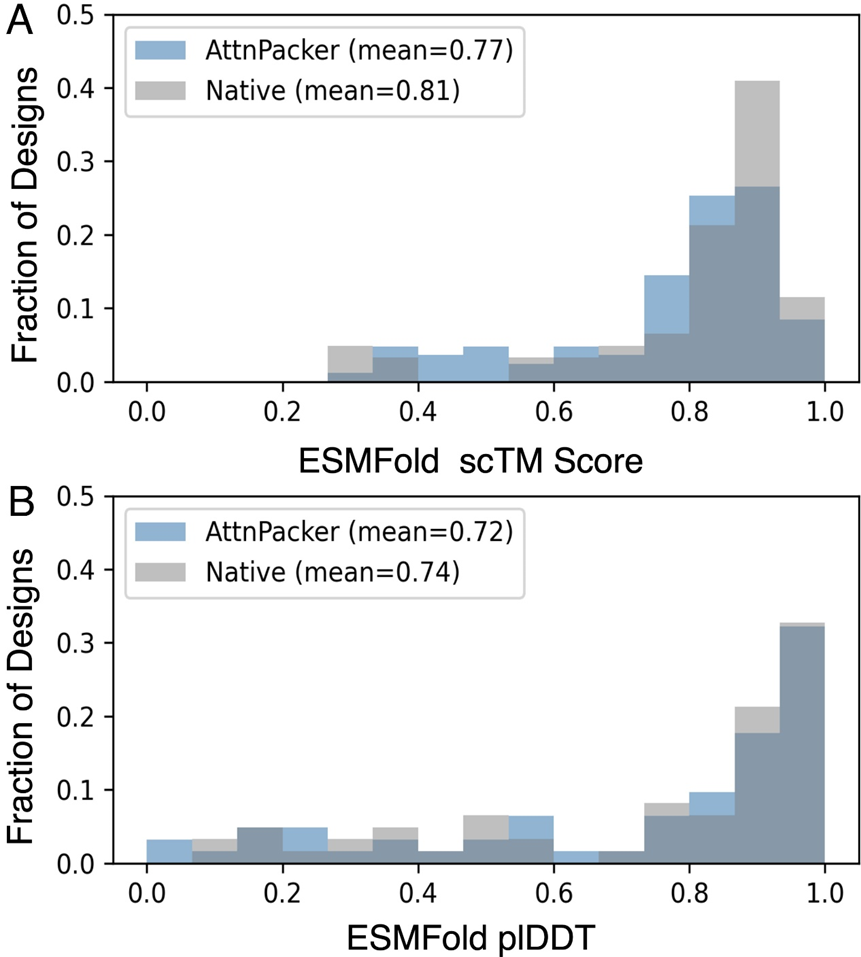
AttnPacker menjalankan proses tersebut. Koordinat dan jujukan tulang belakang protein digunakan sebagai input, dan peta ciri spatial dan asas persamaan diperoleh berdasarkan maklumat koordinat. Peta ciri diproses oleh modul pengubah graf invarian dan kemudian diserahkan kepada TFN-Transformer yang setara yang menghasilkan koordinat rantai sisi yang diramalkan, skor keyakinan untuk setiap sisa dan urutan reka bentuk pilihan. Koordinat yang diramalkan diproses pasca untuk membuang semua konflik spatial dan memastikan geometri ideal. Dari segi kesan ramalan, AttnPacker menunjukkan peningkatan dalam ketepatan dan kecekapan untuk kedua-dua struktur tulang belakang semula jadi dan bukan semula jadi. Pada masa yang sama, kebolehlaksanaan fizikal dipastikan, sisihan daripada panjang dan sudut ikatan yang ideal boleh diabaikan, dan halangan sterik atom yang minimum dihasilkan. Jantung Molekul menguji AttnPacker terhadap kaedah terkini semasa - SCWRL4, FASPR, RosettaPacker dan DLPacker pada set data tulang belakang protein semulajadi dan bukan asli CASP13 dan CASP14. Keputusan menunjukkan bahawa AttnPacker dengan ketara mengatasi kaedah ramalan rantai sisi protein tradisional pada tulang belakang asli CASP13 dan CASP14, dengan purata RMSD pembinaan semula lebih 18% lebih rendah daripada kaedah suboptimum pada setiap set ujian. AttnPacker juga mengatasi kaedah pembelajaran mendalam DLPacker, mengurangkan purata RMSD sebanyak lebih daripada 11% sambil juga meningkatkan ketepatan dihedral rantai sisi dengan ketara. Selain ketepatan, AttnPacker mempunyai perlanggaran atom yang jauh lebih sedikit daripada kaedah lain. Apabila struktur rantai utama semulajadi diberikan, setiap algoritma berfungsi dalam CASP13 dan CASP14 keputusan ramalan struktur rantai sisi pada protein sasaran. Asterisk menunjukkan bahawa nilai konflik purata adalah lebih rendah daripada struktur asli—56.0, 5.9, dan 0.4 untuk CASP13 dan 80.4, 7.9, dan 2.5 untuk CASP14. Pada CASP13 dan CASP14 tulang belakang bukan asli, AttnPacker juga jauh lebih baik daripada kaedah lain dan mempunyai perlanggaran atom yang jauh lebih sedikit daripada kaedah lain. Apabila struktur rantai utama bukan semula jadi diberikan, setiap algoritma berfungsi baik dalam CASP13 dan keputusan ramalan struktur rantai sisi pada protein sasaran CASP14. Asterisk menunjukkan bahawa nilai konflik purata adalah lebih rendah daripada struktur asli yang sepadan—34.6, 2.2, 0.5 untuk CASP13 dan 40.0, 2.7, 0.7 untuk CASP14. Secara inovatif meninggalkan perpustakaan pemutar diskret dan langkah carian dan pensampelan konformasi yang mahal secara pengiraan, dan secara langsung menggabungkan geometri 3D rantaian utama untuk mengira semua sisi dalam koordinat rantai selari. Berbanding dengan kaedah berasaskan pembelajaran mendalam DLPacker dan RosettaPacker berasaskan kaedah pengkomputeran tradisional, AttnPacker telah meningkatkan kecekapan pengkomputeran dengan ketara dan mengurangkan masa inferens sebanyak lebih daripada 100 kali. Perbandingan masa kaedah PSCP yang berbeza. Membina semula masa relatif atom rantai sisi untuk semua 83 protein sasaran CASP13. AttnPacker menunjukkan prestasi yang sama baik dalam reka bentuk protein. Molecular Heart melatih varian AttnPacker untuk reka bentuk bersama yang mencapai kadar pemulihan jujukan asli yang setanding dengan kaedah terkini semasa sambil juga menghasilkan pemasangan yang sangat tepat. Pengesahan simulasi Rosetta menunjukkan bahawa struktur rekaan AttnPacker umumnya menghasilkan tenaga Rosetta subnatif (rendah). Gunakan penunjuk ESMFold scTM dan plDDT untuk membandingkan jujukan protein asli dan Urutan yang dijana oleh AttnPacker digunakan untuk menilai kualiti penjanaan AttnPacker, dan keputusan menunjukkan korelasi yang kukuh. Selain keberkesanan dan kecekapannya yang menakjubkan, AttnPaker juga mempunyai nilai yang sangat praktikal - ia sangat mudah digunakan. AttnPaker hanya memerlukan fail struktur protein untuk dijalankan. Sebaliknya, OPUS-Rota4 (28) memerlukan perwakilan voxel bagi persekitaran atom daripada DLPacker, logik, struktur sekunder daripada trRosetta100, dan fail kekangan daripada output OPUS-CM. Selain itu, memandangkan AttnPacker meramalkan secara langsung koordinat rantaian sisi, output boleh dibezakan sepenuhnya, yang memudahkan tugas ramalan hiliran seperti pengoptimuman atau interaksi protein-protein. "Kelebihan kesan ramalan yang baik, kecekapan tinggi dan kemudahan penggunaan adalah kondusif kepada penggunaan meluas AttnPacker dalam bidang penyelidikan dan perindustrian," kata Profesor Xu Jinbo. 1. AttnPacker ialah model setara SE (3) yang digunakan untuk meramalkan secara langsung jujukan dan koordinat rantai sisi, dan boleh digunakan untuk ramalan struktur rantai sisi protein , yang juga boleh digunakan untuk reka bentuk jujukan protein dan merupakan kerja perintis. 2. Ketepatan AttnPacker adalah lebih baik daripada kaedah lain, kecekapannya bertambah baik, dan ia amat mudah digunakan. 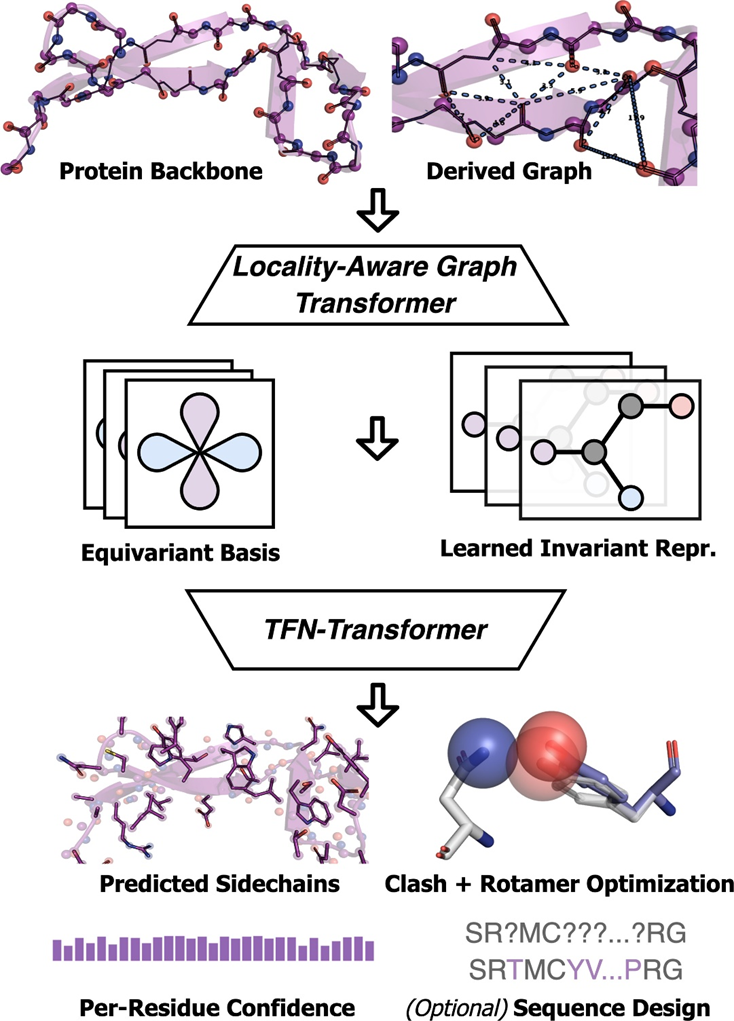
Kesan
Ringkasan
Atas ialah kandungan terperinci Pertama di dunia: Algoritma AI baharu sumber terbuka Molecular Heart untuk mengatasi masalah ramalan rantai sisi protein dan reka bentuk jujukan. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!

Alat AI Hot

Undresser.AI Undress
Apl berkuasa AI untuk mencipta foto bogel yang realistik

AI Clothes Remover
Alat AI dalam talian untuk mengeluarkan pakaian daripada foto.

Undress AI Tool
Gambar buka pakaian secara percuma

Clothoff.io
Penyingkiran pakaian AI

AI Hentai Generator
Menjana ai hentai secara percuma.

Artikel Panas

Alat panas

Notepad++7.3.1
Editor kod yang mudah digunakan dan percuma

SublimeText3 versi Cina
Versi Cina, sangat mudah digunakan

Hantar Studio 13.0.1
Persekitaran pembangunan bersepadu PHP yang berkuasa

Dreamweaver CS6
Alat pembangunan web visual

SublimeText3 versi Mac
Perisian penyuntingan kod peringkat Tuhan (SublimeText3)

Topik panas
 1384
1384
 52
52
 Baris arahan shutdown centos
Apr 14, 2025 pm 09:12 PM
Baris arahan shutdown centos
Apr 14, 2025 pm 09:12 PM
Perintah shutdown CentOS adalah penutupan, dan sintaks adalah tutup [pilihan] [maklumat]. Pilihan termasuk: -h menghentikan sistem dengan segera; -P mematikan kuasa selepas penutupan; -r mulakan semula; -T Waktu Menunggu. Masa boleh ditentukan sebagai segera (sekarang), minit (minit), atau masa tertentu (HH: mm). Maklumat tambahan boleh dipaparkan dalam mesej sistem.
 Cara Memeriksa Konfigurasi HDFS CentOS
Apr 14, 2025 pm 07:21 PM
Cara Memeriksa Konfigurasi HDFS CentOS
Apr 14, 2025 pm 07:21 PM
Panduan Lengkap untuk Memeriksa Konfigurasi HDFS Dalam Sistem CentOS Artikel ini akan membimbing anda bagaimana untuk memeriksa konfigurasi dan menjalankan status HDFS secara berkesan pada sistem CentOS. Langkah -langkah berikut akan membantu anda memahami sepenuhnya persediaan dan operasi HDFS. Sahkan Pembolehubah Alam Sekitar Hadoop: Pertama, pastikan pembolehubah persekitaran Hadoop ditetapkan dengan betul. Di terminal, laksanakan arahan berikut untuk mengesahkan bahawa Hadoop dipasang dan dikonfigurasi dengan betul: Hadoopversion Semak fail konfigurasi HDFS: Fail konfigurasi teras HDFS terletak di/etc/hadoop/conf/direktori, di mana core-site.xml dan hdfs-site.xml adalah kritikal. gunakan
 Apakah kaedah sandaran untuk gitlab di centos
Apr 14, 2025 pm 05:33 PM
Apakah kaedah sandaran untuk gitlab di centos
Apr 14, 2025 pm 05:33 PM
Dasar sandaran dan pemulihan Gitlab di bawah sistem CentOS untuk memastikan keselamatan data dan pemulihan, Gitlab pada CentOS menyediakan pelbagai kaedah sandaran. Artikel ini akan memperkenalkan beberapa kaedah sandaran biasa, parameter konfigurasi dan proses pemulihan secara terperinci untuk membantu anda menubuhkan strategi sandaran dan pemulihan GitLab lengkap. 1. Backup Manual Gunakan Gitlab-Rakegitlab: Backup: Buat Perintah untuk Melaksanakan Backup Manual. Perintah ini menyokong maklumat utama seperti repositori Gitlab, pangkalan data, pengguna, kumpulan pengguna, kunci, dan kebenaran. Fail sandaran lalai disimpan dalam direktori/var/opt/gitlab/sandaran. Anda boleh mengubah suai /etc /gitlab
 Bagaimana sokongan GPU untuk Pytorch di CentOS
Apr 14, 2025 pm 06:48 PM
Bagaimana sokongan GPU untuk Pytorch di CentOS
Apr 14, 2025 pm 06:48 PM
Membolehkan pecutan GPU pytorch pada sistem CentOS memerlukan pemasangan cuda, cudnn dan GPU versi pytorch. Langkah-langkah berikut akan membimbing anda melalui proses: Pemasangan CUDA dan CUDNN Tentukan keserasian versi CUDA: Gunakan perintah NVIDIA-SMI untuk melihat versi CUDA yang disokong oleh kad grafik NVIDIA anda. Sebagai contoh, kad grafik MX450 anda boleh menyokong CUDA11.1 atau lebih tinggi. Muat turun dan pasang Cudatoolkit: Lawati laman web rasmi Nvidiacudatoolkit dan muat turun dan pasang versi yang sepadan mengikut versi CUDA tertinggi yang disokong oleh kad grafik anda. Pasang Perpustakaan Cudnn:
 Penjelasan terperinci mengenai Prinsip Docker
Apr 14, 2025 pm 11:57 PM
Penjelasan terperinci mengenai Prinsip Docker
Apr 14, 2025 pm 11:57 PM
Docker menggunakan ciri -ciri kernel Linux untuk menyediakan persekitaran berjalan yang cekap dan terpencil. Prinsip kerjanya adalah seperti berikut: 1. Cermin digunakan sebagai templat baca sahaja, yang mengandungi semua yang anda perlukan untuk menjalankan aplikasi; 2. Sistem Fail Kesatuan (Unionfs) menyusun pelbagai sistem fail, hanya menyimpan perbezaan, menjimatkan ruang dan mempercepatkan; 3. Daemon menguruskan cermin dan bekas, dan pelanggan menggunakannya untuk interaksi; 4. Ruang nama dan cgroups melaksanakan pengasingan kontena dan batasan sumber; 5. Pelbagai mod rangkaian menyokong interkoneksi kontena. Hanya dengan memahami konsep -konsep teras ini, anda boleh menggunakan Docker dengan lebih baik.
 CentOS memasang MySQL
Apr 14, 2025 pm 08:09 PM
CentOS memasang MySQL
Apr 14, 2025 pm 08:09 PM
Memasang MySQL pada CentOS melibatkan langkah -langkah berikut: Menambah sumber MySQL YUM yang sesuai. Jalankan YUM Pasang Perintah MySQL-Server untuk memasang pelayan MySQL. Gunakan perintah mysql_secure_installation untuk membuat tetapan keselamatan, seperti menetapkan kata laluan pengguna root. Sesuaikan fail konfigurasi MySQL seperti yang diperlukan. Tune parameter MySQL dan mengoptimumkan pangkalan data untuk prestasi.
 Cara Melihat Log Gitlab Di Bawah Centos
Apr 14, 2025 pm 06:18 PM
Cara Melihat Log Gitlab Di Bawah Centos
Apr 14, 2025 pm 06:18 PM
Panduan Lengkap untuk Melihat Log Gitlab Di bawah Sistem CentOS Artikel ini akan membimbing anda bagaimana untuk melihat pelbagai log Gitlab dalam sistem CentOS, termasuk log utama, log pengecualian, dan log lain yang berkaitan. Sila ambil perhatian bahawa laluan fail log mungkin berbeza -beza bergantung pada versi GitLab dan kaedah pemasangan. Jika laluan berikut tidak wujud, sila semak fail Direktori Pemasangan dan Konfigurasi GitLab. 1. Lihat log Gitlab utama Gunakan arahan berikut untuk melihat fail log utama aplikasi GitLabRails: Perintah: Sudocat/var/Log/Gitlab/Gitlab-Rails/Production.log Perintah ini akan memaparkan produk
 Cara Mengendalikan Latihan Pittorch Diagihkan di Centos
Apr 14, 2025 pm 06:36 PM
Cara Mengendalikan Latihan Pittorch Diagihkan di Centos
Apr 14, 2025 pm 06:36 PM
Latihan yang diedarkan Pytorch pada sistem CentOS memerlukan langkah -langkah berikut: Pemasangan Pytorch: Premisnya ialah Python dan PIP dipasang dalam sistem CentOS. Bergantung pada versi CUDA anda, dapatkan arahan pemasangan yang sesuai dari laman web rasmi Pytorch. Untuk latihan CPU sahaja, anda boleh menggunakan arahan berikut: PipinstallToRchTorchVisionTorchaudio Jika anda memerlukan sokongan GPU, pastikan versi CUDA dan CUDNN yang sama dipasang dan gunakan versi pytorch yang sepadan untuk pemasangan. Konfigurasi Alam Sekitar Teragih: Latihan yang diedarkan biasanya memerlukan pelbagai mesin atau mesin berbilang mesin tunggal. Tempat



