SOTA性能,多尺度學習,中山大學提出蛋白質-藥物交互作用AI框架
編輯 | 紫羅蘭
蛋白質、藥物和其他生物分子之間的相互作用,在各種生物過程中發揮至關重要的作用。了解這些交互作用對於破解生物學過程背後的分子機制和開發新的治療策略至關重要。 蛋白質是細胞中最重要的分子之一,它們在細胞內執行各種功能。藥物通常透過與特定的蛋白質相互作用來調節生理過程。這些交互作用可以促進或抑制特定的分子訊號傳遞路徑
目前的多尺度計算方法,常常過於依賴單一尺度,而對其他尺度的擬合不足。這可能與多尺度學習的不平多尺度向性和固有的貪婪性有關。
為了緩解優化不平衡,中山大學和上海交通大學的研究人員提出了一種基於變量期望最大化的多尺度表示學習框架 MUSE,它可以有效地整合多尺度資訊進行學習。此策略透過相互監督和迭代優化,有效融合原子結構和分子網路尺度之間的多尺度資訊。這種方法可以提供更大程度的資訊傳遞和學習。此策略透過相互監督和迭代優化,有效融合原子結構和分子網路尺度之間的多尺度資訊。
MUSE+不僅在分子交互作用(蛋白質-蛋白質、藥物-蛋白質和藥物)任務方面優於目前最先進的模型,而且在原子結構尺度的蛋白質界面預測方面也優於目前最先進的模型。更重要的是,多尺度學習框架可擴展到其他尺度的計算藥物發現。
研究以「A variational expectation-maximization framework for balanced multi-scale learning of protein and drug interactions」為題,於5 月25 日發佈在《Nature Communications 》上。

#生物分子間相互作用
蛋白質功能的特徵是與蛋白質、藥物和其他生物分子的相互作用。了解這些相互作用對於破解生物過程的分子機制和開發新的治療策略至關重要。然而,與實驗相互作用相關的需求和成本的大幅增長,需要計算工具來自動預測和理解生物分子之間的相互作用。 為了滿足這些需求和成本的成長,需要計算工具來自動預測和理解生物分子之間的相互作用。
純粹從結構預測這些交互作用是結構生物學中最重要的挑戰之一。目前的計算方法大多基於分子網路或結構資訊來預測相互作用,並沒有將它們整合到統一的多尺度框架中。
雖然有些多視圖學習方法致力於融合多尺度訊息,學習多尺度表示的直覺方法是將分子圖與互動網路結合並共同優化它們。然而,由於多尺度學習的不平衡性和固有的貪婪性,這些模型通常集中依賴單一尺度。無法有效地利用所有尺度相關的信息,並且泛化差。
此外,有效的多尺度框架不僅需要捕捉不同尺度內的豐富訊息,而且還需要很好地保留它們之間的潛在關係。
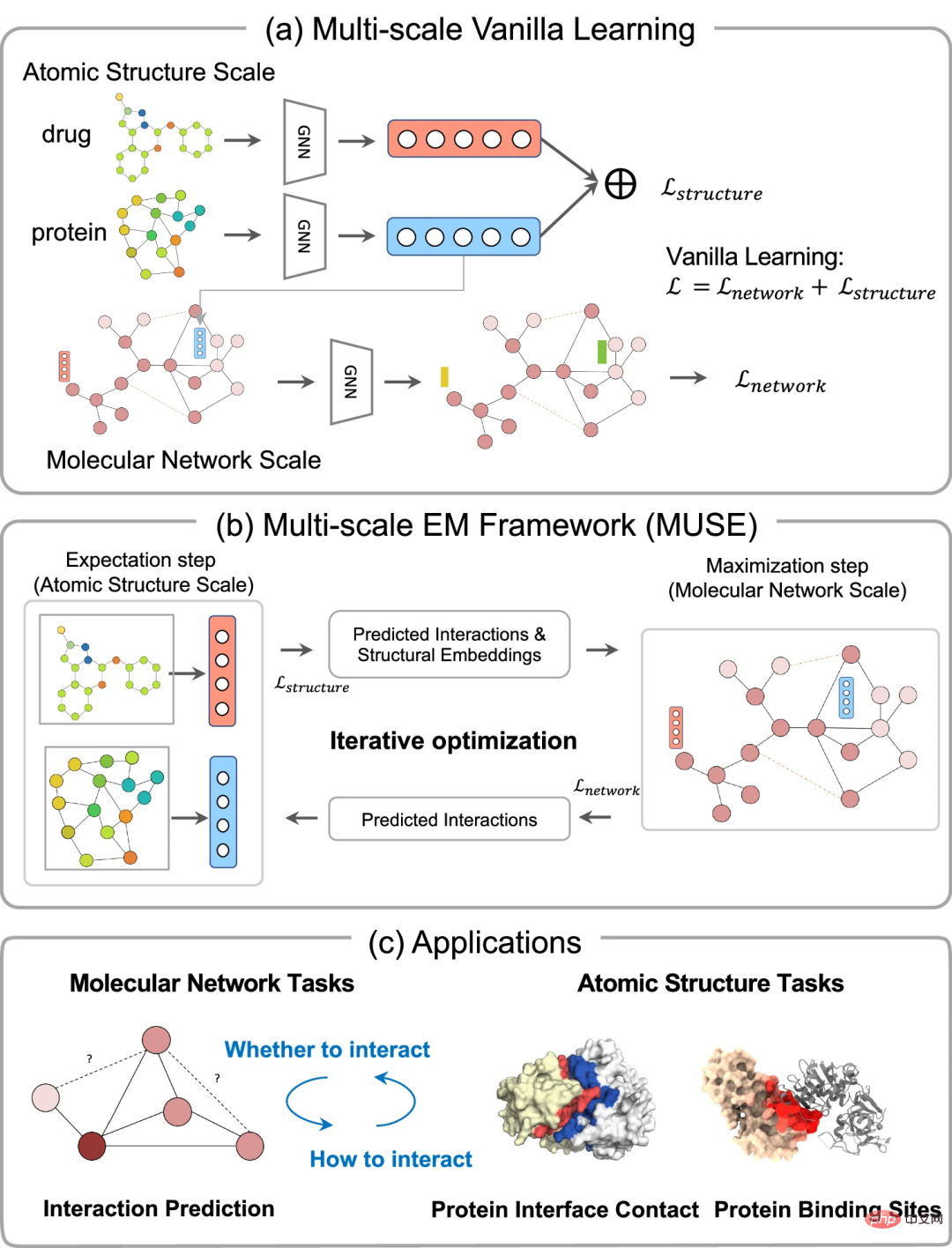
MUSE 用於學習蛋白質和藥物多尺度資訊
在此,中山大學研究團隊提出了MUSE,一種基於變量期望最大化(Expectation Maximization)的多尺度表示學習框架,它可以在多次迭代的交替過程中最佳化不同尺度。與嚴重依賴單尺度資訊的現有方法相比,MUSE 透過相互監督和迭代優化,有效解決了多尺度學習中的最佳化不平衡問題。
MUSE 是一種多尺度學習方法,透過變分期望最大化(EM)框架,將分子結構建模和蛋白質與藥物交互作用網絡學習相結合。 EM 框架在多次迭代的交替過程中優化兩個模組,即期望步驟(E-step)和最大化步驟(M-step)。
在 E-step 期間,MUSE 利用每個生物分子的結構資訊來學習有效的結構表示,以便在 M-step 中使用已知的相互作用和增強樣本進行訓練。它將蛋白質和藥物對及其原子級結構資訊作為輸入,並透過 M-step 預測的相互作用進行增強。 M-step 將分子級相互作用網絡、結構嵌入和 E-step 的預測相互作用作為輸入,並輸出預測的相互作用。 E-step 和 M-step 之間的迭代優化確保了分子結構和網路資訊的交互捕獲,並在兩個尺度上具有不同的學習率。
相互監督確保每個尺度模型以適當的方式學習,從而能夠利用不同尺度的有效資訊。該框架將在蛋白質和藥物之間相互作用的多個多尺度中得到證明。分析了 MUSE 減輕了多尺度學習中的不平衡特徵,並有效地整合了來自不同尺度的分層和互補資訊。
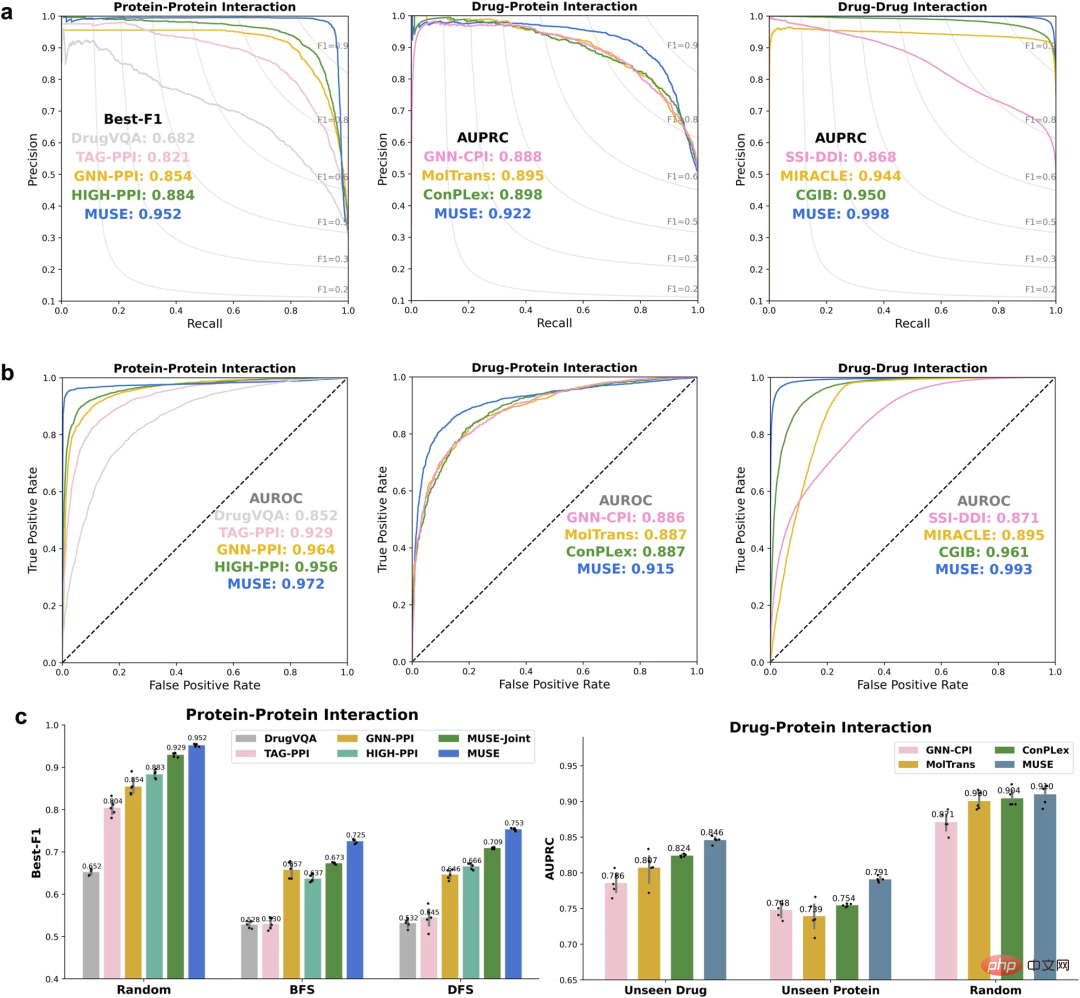
在多尺度任務上優於目前最先進的模型
利用原子結構資訊來改進分子網路尺度的預測
為了評估其方法,首先,研究人員利用MUSE 整合原子結構資訊來改進分子網路尺度(scale)預測。 MUSE 在蛋白質-蛋白質交互作用(PPI)、藥物-蛋白質交互作用(DPI)和藥物-藥物交互作用(DDI)這三個多尺度交互作用預測任務中取得了最先進的表現。
從分子網路尺度改進原子結構尺度的預測
除了利用原子結構資訊改進分子網路尺度預測之外,研究人員也進一步研究了MUSE 在原子結構尺度上學習和預測結構特性的能力,包括預測與PPI 相關的界面接觸和結合位點。
為了評估蛋白質鏈間接觸的預測,將 MUSE 與 DIPS-Plus 基準測試中最先進的方法進行了比較。 MUSE 始終優於所有其他方法,驗證了其在原子結構預測中的有效性和適應性。
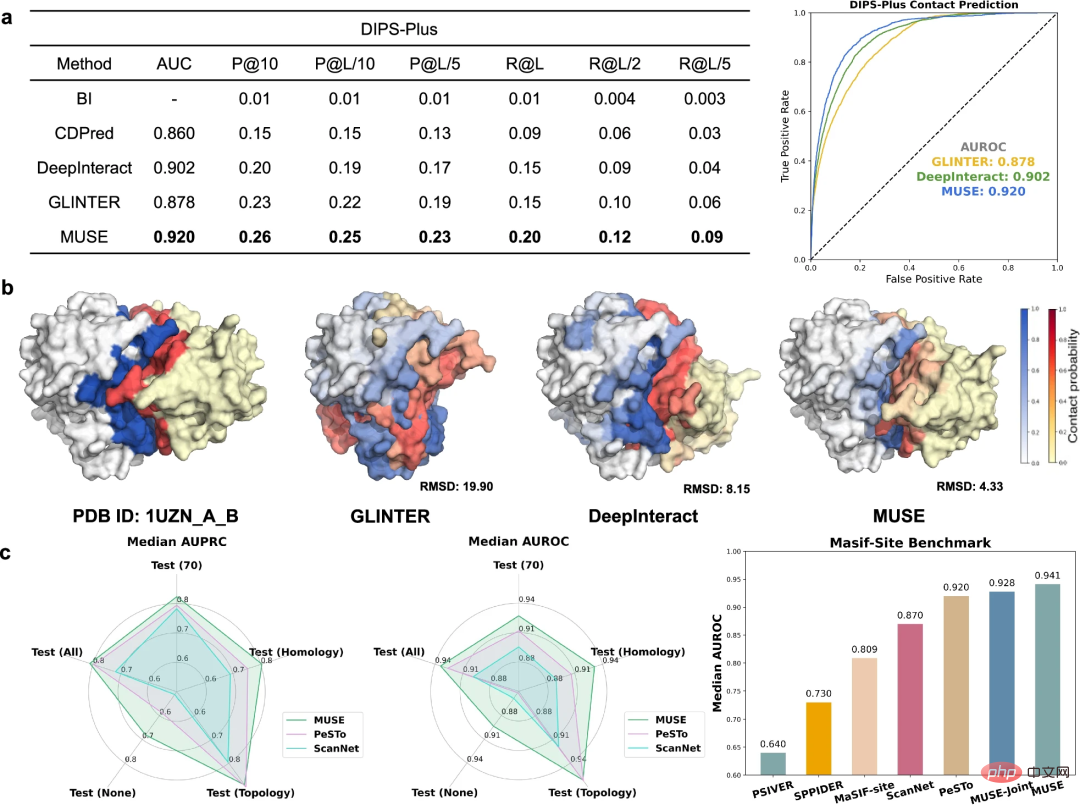
進一步評估 MUSE 以預測殘基是否直接參與蛋白質-蛋白質交互作用。結果表明,MUSE 中分子網路規模的學習可以為原子結構尺度的預測提供有價值的見解。
透過迭代優化來緩解多尺度學習的不平衡特性
為了探究為什麼MUSE 能夠取得多尺度表示的優越性能,研究人員針對多尺度學習的不平衡特性分析了MUSE 的學習能力。
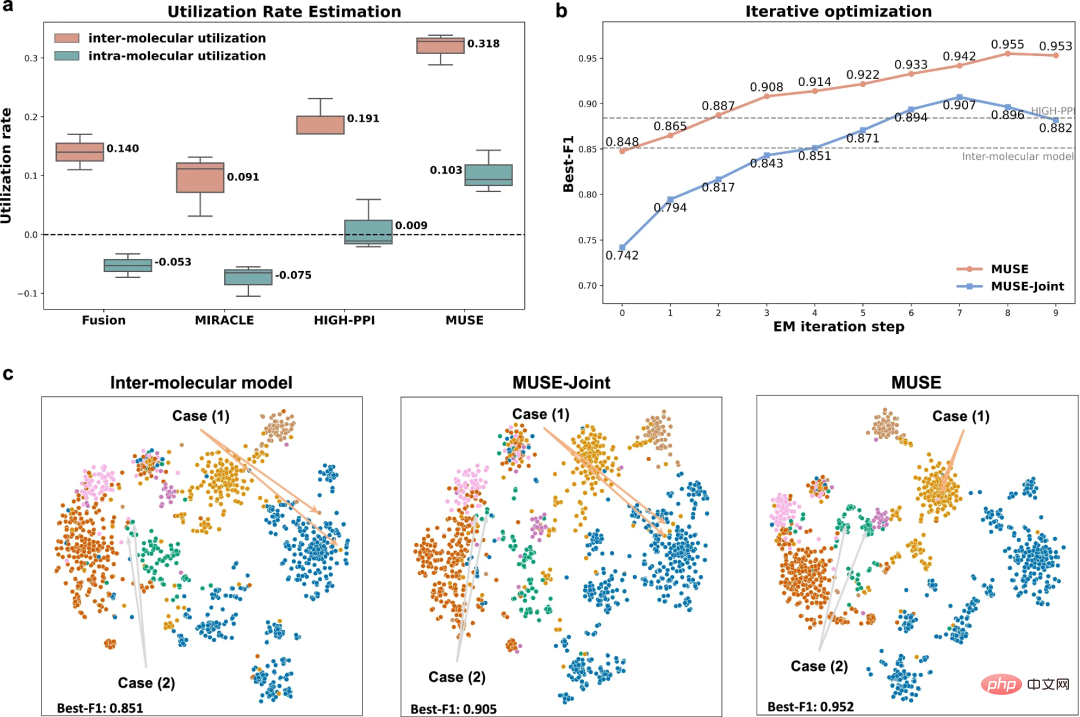
結果表明,MUSE有效緩解了多尺度學習中的不平衡特性和貪婪學習,保證了訓練過程中不同尺度資訊的綜合利用。此外,利用率(utilization rate)分析的實驗使研究人員能夠具體了解模型學到了什麼,並證明使用 MUSE 來平衡模型在不同尺度上的學習可以增強泛化能力。
所學習的多尺度表徵的視覺化和解釋
為了更好地理解學習到的多尺度表示,研究人員從不同的角度研究了MUSE 學習到的多尺度表示,包括(1)MUSE 捕獲PPI 中涉及的原子結構資訊(即結構基序和嵌入)的能力,以及(2)學習到的原子結構和分子網路表示之間的相互監督。
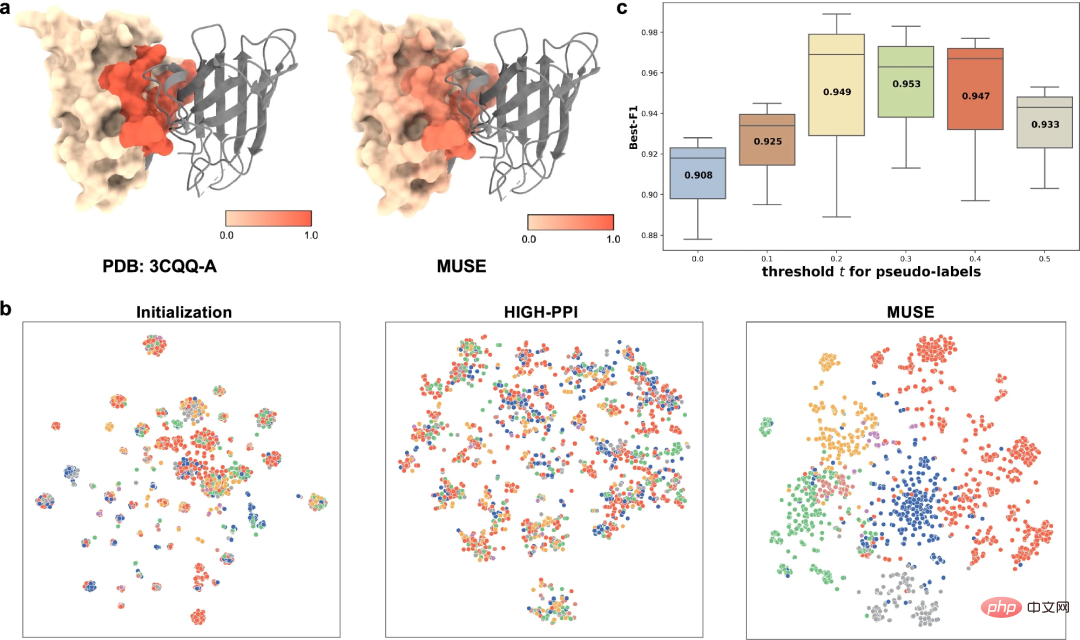
作為結合位點預測的範例(PDB id:3CQQ-A),MUSE 可以準確識別屬於結合位點的殘基,準確率為 97.7%。這表明 MUSE 中的相互監督有助於原子結構尺度模型學習與相互作用相關的關鍵子結構。
最後,研究人員也進行了消融研究,來研究原子結構尺度預測的偽標籤對分子網路尺度的影響。
雖然 MUSE 在基準測試中展示了最先進的性能,但仍有可能提高其處理噪音和不完整的多尺度下游任務的能力。這可以透過知識圖和可解釋的 AI 技術將先驗知識結合起來。另一方面,此概念性多尺度架構也可擴及其他尺度的計算藥物發現。
以上是SOTA性能,多尺度學習,中山大學提出蛋白質-藥物交互作用AI框架的詳細內容。更多資訊請關注PHP中文網其他相關文章!

熱AI工具

Undresser.AI Undress
人工智慧驅動的應用程序,用於創建逼真的裸體照片

AI Clothes Remover
用於從照片中去除衣服的線上人工智慧工具。

Undress AI Tool
免費脫衣圖片

Clothoff.io
AI脫衣器

AI Hentai Generator
免費產生 AI 無盡。

熱門文章

熱工具

記事本++7.3.1
好用且免費的程式碼編輯器

SublimeText3漢化版
中文版,非常好用

禪工作室 13.0.1
強大的PHP整合開發環境

Dreamweaver CS6
視覺化網頁開發工具

SublimeText3 Mac版
神級程式碼編輯軟體(SublimeText3)
 突破傳統缺陷檢測的界限,\'Defect Spectrum\'首次實現超高精度豐富語意的工業缺陷檢測。
Jul 26, 2024 pm 05:38 PM
突破傳統缺陷檢測的界限,\'Defect Spectrum\'首次實現超高精度豐富語意的工業缺陷檢測。
Jul 26, 2024 pm 05:38 PM
突破傳統缺陷檢測的界限,\'Defect Spectrum\'首次實現超高精度豐富語意的工業缺陷檢測。
 GoogleAI拿下IMO奧數銀牌,數學推理模型AlphaProof面世,強化學習 is so back
Jul 26, 2024 pm 02:40 PM
GoogleAI拿下IMO奧數銀牌,數學推理模型AlphaProof面世,強化學習 is so back
Jul 26, 2024 pm 02:40 PM
GoogleAI拿下IMO奧數銀牌,數學推理模型AlphaProof面世,強化學習 is so back
 為大模型提供全新科學複雜問答基準與評估體系,UNSW、阿貢、芝加哥大學等多家機構共同推出SciQAG框架
Jul 25, 2024 am 06:42 AM
為大模型提供全新科學複雜問答基準與評估體系,UNSW、阿貢、芝加哥大學等多家機構共同推出SciQAG框架
Jul 25, 2024 am 06:42 AM
為大模型提供全新科學複雜問答基準與評估體系,UNSW、阿貢、芝加哥大學等多家機構共同推出SciQAG框架
 數百萬晶體資料訓練,解決晶體學相位問題,深度學習方法PhAI登Science
Aug 08, 2024 pm 09:22 PM
數百萬晶體資料訓練,解決晶體學相位問題,深度學習方法PhAI登Science
Aug 08, 2024 pm 09:22 PM
數百萬晶體資料訓練,解決晶體學相位問題,深度學習方法PhAI登Science
 SOTA性能,廈大多模態蛋白質-配體親和力預測AI方法,首次結合分子表面訊息
Jul 17, 2024 pm 06:37 PM
SOTA性能,廈大多模態蛋白質-配體親和力預測AI方法,首次結合分子表面訊息
Jul 17, 2024 pm 06:37 PM
SOTA性能,廈大多模態蛋白質-配體親和力預測AI方法,首次結合分子表面訊息
 涵蓋文本、定位和分割任務,智源、港中文聯合提出首個多功能3D醫學多模態大模型
Jun 22, 2024 am 07:16 AM
涵蓋文本、定位和分割任務,智源、港中文聯合提出首個多功能3D醫學多模態大模型
Jun 22, 2024 am 07:16 AM
涵蓋文本、定位和分割任務,智源、港中文聯合提出首個多功能3D醫學多模態大模型








