清華AIR等提出ESM-AA,首個從胺基酸到原子尺度的蛋白質語言模型

來自清華大學AIR、北京大學、南京大學的研究團隊提出了 ESM-AA 模型。該模型在蛋白質語言建模領域取得了重要進展,提供了一套整合多尺度資訊的統一建模方案。
它是第一個能同時處理胺基酸資訊和原子資訊的蛋白質預訓練語言模型。模型的出色性能展示了多尺度統一建模在克服現有限制和解鎖新能力方面的巨大潛力。
作為基座模型,ESM-AA 獲得了多位學者的關注與廣泛討論(截圖見下方),被認為有潛力基於ESM-AA 開發出可與AlphaFold3、RoseTTAFold All-Atom 相競爭的模型,為研究不同生物結構間的相互作用開闢了新的道路。目前論文已被 ICML 2024 錄⽤。
研究背景
蛋白質是各種生命活動的關鍵執行者。深入理解蛋白質及其與其他生物結構的相互作用是生物科學中的核心議題,這對標靶藥物篩選、酵素工程等領域具有顯著的實際意義。
因此,如何更好地理解與建模蛋白質也成為了目前 AI4Science 領域的一個研究熱點。
近日來,包括 Deepmind、華盛頓大學Baker 組在內的各大前沿研究機構也針對蛋白質全原子建模問題展開了深入研究,提出了包括AlphaFold 3、RoseTTAFold All-Atom 等針對蛋白質以及其他生命活動相關分子的全原子尺度建模模型,可以在很高的精度下實現對蛋白質結構、分子結構以及受體-配體結構等全原子尺度的精確預測。
雖然這些模型對於全原子尺度的結構建模取得了重大進展,但目前主流的蛋白質語言模型仍無法實現全原子尺度的蛋白質理解與表示學習。
多尺度,下一代蛋白質模型的「必經之路」
以ESM-2為代表的蛋白質表示學習模型,它們以氨基酸作為構建模型的唯一尺度,這對於專注於處理蛋白質的情境而言是一種合理的方法。
然而,要全面理解蛋白質的本質,關鍵在於闡述它們與其他生物結構(如小分子、DNA、RNA 等)之間的相互作用。
面對這種需求,需要描述不同結構間複雜的相互作用,單一尺度的建模策略難以提供有效的全面覆蓋。
為了克服這個缺陷,蛋白質模型正在經歷一場轉變為多尺度模型的深刻革新。例如,5月初發表在《Science》雜誌上的 RoseTTAFold All-Atom 模型,作為 RoseTTAFold 的後續產品,引入了多尺度概念。
這個模型不僅限於蛋白質結構預測,也拓展到了蛋白質與分子/核酸的對接、蛋白質翻譯後修飾等更廣泛的研究領域。
同時,DeepMind 最新發布的 AlphaFold3 也採用了多尺度建模策略,支持預測多種蛋白複合物的結構,其表現令人矚目,無疑將對人工智慧和生物學領域產生重大影響。
ESM All-Atom,多尺度的蛋白質語言模型基座
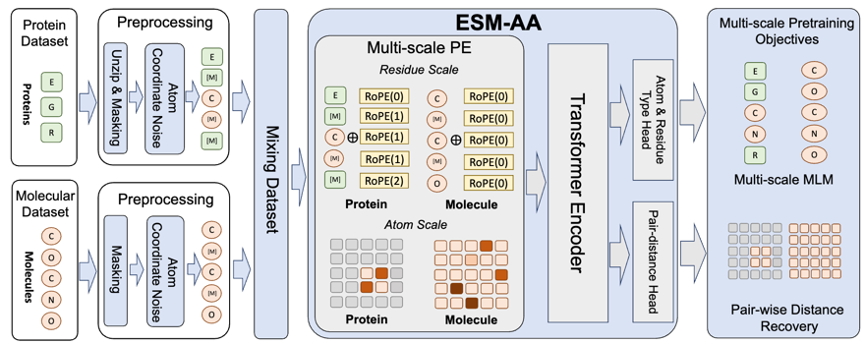
RoseTTAFold All-Atom 和AlphaFold3 對多尺度概念的成功應用啟發了一個尺度概念的成功應用啟發了一個尺度概念的成功應用啟發了一個尺度概念的成功應用啟發重要思考,即:作為蛋白質基座模型的蛋白質語言模型應如何採納多尺度技術。基於此,團隊提出了多尺度蛋白質語言模型 ESM All-Atom(ESM-AA)。
簡要來說,ESM-AA 透過將部分胺基酸「展開」(Unzip)為對應的原子組成引入了多尺度概念。隨後,透過混合蛋白質資料與分子資料進行預訓練,這使得模型具備了同時處理不同尺度生物結構的能力。
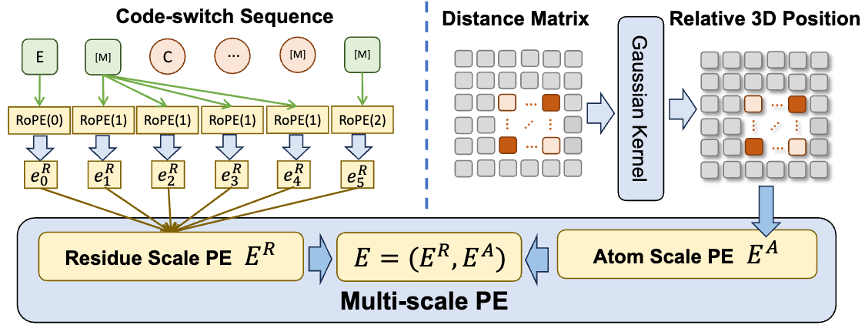
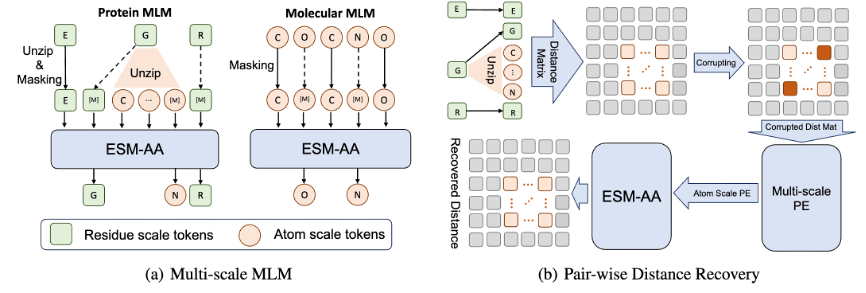
此外,為了幫助模型更好地學習優質的原子尺度信息,ESM-AA 還會利用原子尺度的分子結構數據進行訓練。而且透過引入圖 2 所示的多尺度位置編碼機制,ESM-AA 模型可以很好地對不同尺度的資訊進行區分,確保模型能夠精確理解殘基層面與原子層面的位置與結構資訊。
多尺度預訓練目標
為了幫助模型學習多尺度訊息,該團隊為 ESM-AA 模型設計了多種預訓練目標。 ESM-AA 的多尺度預訓練目標包括遮罩語言建模(MLM)和成對距離恢復(PDR)。如圖 3(a) 所示,MLM 透過遮蓋胺基酸和原子,要求模型根據周圍的上下文進行預測,這項訓練任務可以在胺基酸和原子兩個尺度上進行。而 PDR 則要求模型準確預測不同原子之間的歐幾里德距離,以訓練模型理解原子級的結構資訊(如圖 3(b) 所示)。
實驗驗證
性能評估
ESM-AA 模型在多種蛋白-小分子基準上進行微調和小分子基準評估物親和力回歸任務(結果顯示於圖4)、酵素-底物對分類任務(結果顯示於圖4)和藥物-標靶親和力回歸任務(結果顯示於圖5)。
結果顯示,ESM-AA 在這些任務中優於先前的模型,顯示其在胺基酸和原子尺度上充分發揮了蛋白質預訓練語言模型的潛力。
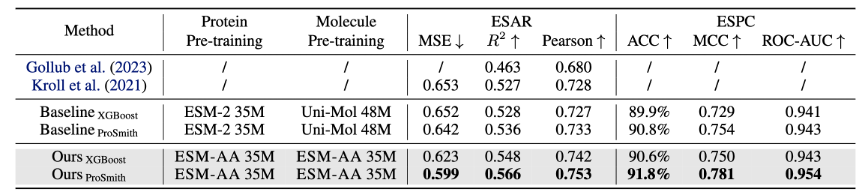
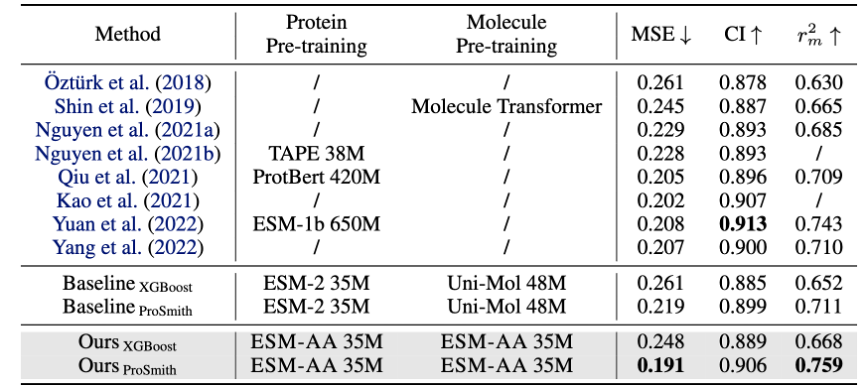
圖5:藥物-靶標親和力回歸任務性能比較在蛋白質接觸預測、蛋白質功能分類以及分子性質預測等任務上測試了表現。
結果顯示,在處理僅涉及蛋白質的任務時,ESM-AA 的表現與ESM-2 相當;在分子任務上,ESM-AA 模型的性能優於大多數基準模型,與Uni-Mol 的表現相近。
這表明,ESM-AA 在獲取強大分子知識的過程中並未犧牲對蛋白質的理解能力,也進一步說明了ESM-AA 模型成功復用了ESM-2 模型的知識,而無需從頭開始重新開發,顯著降低了模型訓練成本。
可視化分析為了進一步分析ESM-AA 在蛋白-小分子基準任務上表現優異的原因,該論文展示了ESM-AA 模型以及ESM-2+Uni-Mol 模型組合在該任務中抽取的樣本表徵分佈的可視化結果。
如圖 6 所示,ESM-AA 模型所學習的蛋白和小分子表示之間更為緊湊,這預示著兩者處於同一表示空間,這是ESM-AA 模型優於ESM-2+Uni- Mol 模型的原因,進一步說明了多尺度統一分子建模的優勢。
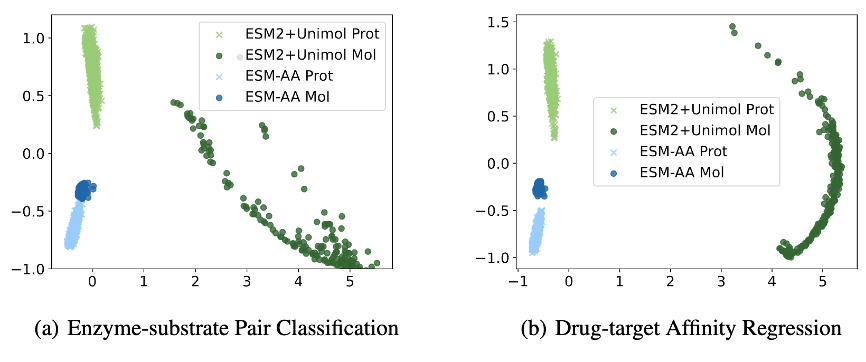 圖 6:蛋白/分子表示的可視化分析
圖 6:蛋白/分子表示的可視化分析
清華 AIR 團隊開發的 ESM-AA 是首個融合氨基酸與原子訊息處理的蛋白質預訓練模型。模型透過整合多尺度訊息,展現出穩健且卓越的性能,為解決生物結構間相互作用難題提供了新途徑。
ESM-AA不僅促進了對蛋白質更深層次的理解,還在多項生物分子任務中表現出色,證明了其在保持蛋白質理解能力的同時可以有效融合分子層面知識,降低了模型訓練的成本,為AI 輔助的生物科學研究開啟了新方向。
論文標題:ESM All-Atom:
Multi-Scale Protein Language Model for Unified Molecular ModelingGithub開源位址:
https://github.com/zhengkangange/ESM-AA ://arxiv.org/abs/2403.12995以上是清華AIR等提出ESM-AA,首個從胺基酸到原子尺度的蛋白質語言模型的詳細內容。更多資訊請關注PHP中文網其他相關文章!

熱AI工具

Undresser.AI Undress
人工智慧驅動的應用程序,用於創建逼真的裸體照片

AI Clothes Remover
用於從照片中去除衣服的線上人工智慧工具。

Undress AI Tool
免費脫衣圖片

Clothoff.io
AI脫衣器

Video Face Swap
使用我們完全免費的人工智慧換臉工具,輕鬆在任何影片中換臉!

熱門文章

熱工具

記事本++7.3.1
好用且免費的程式碼編輯器

SublimeText3漢化版
中文版,非常好用

禪工作室 13.0.1
強大的PHP整合開發環境

Dreamweaver CS6
視覺化網頁開發工具

SublimeText3 Mac版
神級程式碼編輯軟體(SublimeText3)
 突破傳統缺陷檢測的界限,\'Defect Spectrum\'首次實現超高精度豐富語意的工業缺陷檢測。
Jul 26, 2024 pm 05:38 PM
突破傳統缺陷檢測的界限,\'Defect Spectrum\'首次實現超高精度豐富語意的工業缺陷檢測。
Jul 26, 2024 pm 05:38 PM
在現代製造業中,精準的缺陷檢測不僅是確保產品品質的關鍵,更是提升生產效率的核心。然而,現有的缺陷檢測資料集常常缺乏實際應用所需的精確度和語意豐富性,導致模型無法辨識特定的缺陷類別或位置。為了解決這個難題,由香港科技大學廣州和思謀科技組成的頂尖研究團隊,創新地開發了「DefectSpectrum」資料集,為工業缺陷提供了詳盡、語義豐富的大規模標註。如表一所示,相較於其他工業資料集,「DefectSpectrum」資料集提供了最多的缺陷標註(5438張缺陷樣本),最細緻的缺陷分類(125個缺陷類別
 數百萬晶體資料訓練,解決晶體學相位問題,深度學習方法PhAI登Science
Aug 08, 2024 pm 09:22 PM
數百萬晶體資料訓練,解決晶體學相位問題,深度學習方法PhAI登Science
Aug 08, 2024 pm 09:22 PM
編輯|KX時至今日,晶體學所測定的結構細節和精度,從簡單的金屬到大型膜蛋白,是任何其他方法都無法比擬的。然而,最大的挑戰——所謂的相位問題,仍然是從實驗確定的振幅中檢索相位資訊。丹麥哥本哈根大學研究人員,開發了一種解決晶體相問題的深度學習方法PhAI,利用數百萬人工晶體結構及其相應的合成衍射數據訓練的深度學習神經網絡,可以產生準確的電子密度圖。研究表明,這種基於深度學習的從頭算結構解決方案方法,可以以僅2埃的分辨率解決相位問題,該分辨率僅相當於原子分辨率可用數據的10%到20%,而傳統的從頭算方
 英偉達對話模式ChatQA進化到2.0版本,上下文長度提到128K
Jul 26, 2024 am 08:40 AM
英偉達對話模式ChatQA進化到2.0版本,上下文長度提到128K
Jul 26, 2024 am 08:40 AM
開放LLM社群正是百花齊放、競相爭鳴的時代,你能看到Llama-3-70B-Instruct、QWen2-72B-Instruct、Nemotron-4-340B-Instruct、Mixtral-8x22BInstruct-v0.1等許多表現優良的模型。但是,相較於以GPT-4-Turbo為代表的專有大模型,開放模型在許多領域仍有明顯差距。在通用模型之外,也有一些專精關鍵領域的開放模型已被開發出來,例如用於程式設計和數學的DeepSeek-Coder-V2、用於視覺-語言任務的InternVL
 GoogleAI拿下IMO奧數銀牌,數學推理模型AlphaProof面世,強化學習 is so back
Jul 26, 2024 pm 02:40 PM
GoogleAI拿下IMO奧數銀牌,數學推理模型AlphaProof面世,強化學習 is so back
Jul 26, 2024 pm 02:40 PM
對AI來說,奧數不再是問題了。本週四,GoogleDeepMind的人工智慧完成了一項壯舉:用AI做出了今年國際數學奧林匹克競賽IMO的真題,並且距拿金牌僅一步之遙。上週剛結束的IMO競賽共有六道賽題,涉及代數、組合學、幾何和數論。谷歌提出的混合AI系統做對了四道,獲得28分,達到了銀牌水準。本月初,UCLA終身教授陶哲軒剛剛宣傳了百萬美元獎金的AI數學奧林匹克競賽(AIMO進步獎),沒想到7月還沒過,AI的做題水平就進步到了這種水平。 IMO上同步做題,做對了最難題IMO是歷史最悠久、規模最大、最負
 PRO | 為什麼基於 MoE 的大模型更值得關注?
Aug 07, 2024 pm 07:08 PM
PRO | 為什麼基於 MoE 的大模型更值得關注?
Aug 07, 2024 pm 07:08 PM
2023年,幾乎AI的每個領域都在以前所未有的速度進化,同時,AI也不斷地推動著具身智慧、自動駕駛等關鍵賽道的技術邊界。在多模態趨勢下,Transformer作為AI大模型主流架構的局面是否會撼動?為何探索基於MoE(專家混合)架構的大模型成為業界新趨勢?大型視覺模型(LVM)能否成為通用視覺的新突破? ……我們從過去的半年發布的2023年本站PRO會員通訊中,挑選了10份針對以上領域技術趨勢、產業變革進行深入剖析的專題解讀,助您在新的一年裡為大展宏圖做好準備。本篇解讀來自2023年Week50
 為大模型提供全新科學複雜問答基準與評估體系,UNSW、阿貢、芝加哥大學等多家機構共同推出SciQAG框架
Jul 25, 2024 am 06:42 AM
為大模型提供全新科學複雜問答基準與評估體系,UNSW、阿貢、芝加哥大學等多家機構共同推出SciQAG框架
Jul 25, 2024 am 06:42 AM
編輯|ScienceAI問答(QA)資料集在推動自然語言處理(NLP)研究中發揮著至關重要的作用。高品質QA資料集不僅可以用於微調模型,也可以有效評估大語言模型(LLM)的能力,尤其是針對科學知識的理解和推理能力。儘管目前已有許多科學QA數據集,涵蓋了醫學、化學、生物等領域,但這些數據集仍有一些不足之處。其一,資料形式較為單一,大多數為多項選擇題(multiple-choicequestions),它們易於進行評估,但限制了模型的答案選擇範圍,無法充分測試模型的科學問題解答能力。相比之下,開放式問答
 準確率達60.8%,浙大基於Transformer的化學逆合成預測模型,登Nature子刊
Aug 06, 2024 pm 07:34 PM
準確率達60.8%,浙大基於Transformer的化學逆合成預測模型,登Nature子刊
Aug 06, 2024 pm 07:34 PM
編輯|KX逆合成是藥物發現和有機合成中的關鍵任務,AI越來越多地用於加快這一過程。現有AI方法性能不盡人意,多樣性有限。在實踐中,化學反應通常會引起局部分子變化,反應物和產物之間存在很大重疊。受此啟發,浙江大學侯廷軍團隊提出將單步逆合成預測重新定義為分子串編輯任務,迭代細化目標分子串以產生前驅化合物。並提出了基於編輯的逆合成模型EditRetro,該模型可以實現高品質和多樣化的預測。大量實驗表明,模型在標準基準資料集USPTO-50 K上取得了出色的性能,top-1準確率達到60.8%。
 Nature觀點,人工智慧在醫學上的測試一片混亂,該怎麼做?
Aug 22, 2024 pm 04:37 PM
Nature觀點,人工智慧在醫學上的測試一片混亂,該怎麼做?
Aug 22, 2024 pm 04:37 PM
編輯|ScienceAI基於有限的臨床數據,數百種醫療演算法已被批准。科學家們正在討論由誰來測試這些工具,以及如何最好地進行測試。 DevinSingh在急診室目睹了一名兒科患者因長時間等待救治而心臟驟停,這促使他探索AI在縮短等待時間中的應用。 Singh利用了SickKids急診室的分診數據,與同事們建立了一系列AI模型,用於提供潛在診斷和推薦測試。一項研究表明,這些模型可以加快22.3%的就診速度,將每位需要進行醫學檢查的患者的結果處理速度加快近3小時。然而,人工智慧演算法在研究中的成功只是驗證此






