在藥物研發中,確定小分子配體對蛋白質的結合親和力和功能效應至關重要。目前的計算方法可以預測這些蛋白質-配體相互作用特性,但如果沒有高解析度的蛋白質結構,通常會失去準確性,並且無法預測功能效應。
莫納什大學(Monash University)和格里菲斯大學(Griffith University)的研究人員開發了PSICHIC(PhySIcoCHemICal 圖神經網絡),這是一個結合物理化學約束的框架,可直接從序列數據解碼交互作用指紋(fingerprints)。這使 PSICHIC 能夠解碼蛋白質-配體相互作用背後的機制,實現最先進的準確性和可解釋性。
在沒有結構數據的相同蛋白質-配體對上進行訓練後,PSICHIC 在結合親和力預測方面與領先的基於結構的方法性能相當,甚至超過了它們。
PSICHIC 的可解釋指紋識別了參與相互作用的蛋白質殘基和配體原子,並有助於揭示蛋白質-配體相互作用的選擇性決定因素。
本研究以「Physicochemical graph neural network for learning protein–ligand interaction fingerprints from sequence data」為題,於 2024 年 6 月 17 日發佈在《Nature MachinelienceNature Machinelience
Nature Machinegence Nature Machinegence
Nature Machinegence
在藥物發現過程中,了解小分子配體對蛋白質的結合親和力和功能效應至關重要,因為配體與特定蛋白質的選擇性相互作用決定了藥物的預期效果。
然而,目前的計算方法雖然能夠預測蛋白質-配體相互作用屬性,但在缺乏高分辨率蛋白質結構的情況下,預測準確性往往會下降,且在預測功能效應方面也存在困難。
基於序列的方法雖然在成本和資源上更具優勢(例如不需要昂貴的實驗結構確定過程),但這些方法常常面臨模式匹配中的過度自由度問題,容易導致過擬合和有限的泛化能力,從而造成與基於結構或複合物的方法之間的性能差距。
物理化學圖神經網絡莫納什大學和格里菲斯大學的研究團隊開發了PSICHIC(物理化學圖神經網絡),這是一種遵循物理化學原理從序列數據直接解碼蛋白質-配體相互作用指紋的方法。與以往基於序列的模型不同,PSICHIC 獨特地結合了物理化學約束,以實現最先進的準確性和可解釋性。
 作為一種基於二維序列的方法,PSICHIC 透過應用聚類演算法來產生並在二維圖上施加這些約束,從而使 PSICHIC 能夠主要適應訓練期間決定蛋白質-配體相互作用的合理基本模式。與最先進的基於結構和基於複合物的方法相媲美甚至超越了它們。
作為一種基於二維序列的方法,PSICHIC 透過應用聚類演算法來產生並在二維圖上施加這些約束,從而使 PSICHIC 能夠主要適應訓練期間決定蛋白質-配體相互作用的合理基本模式。與最先進的基於結構和基於複合物的方法相媲美甚至超越了它們。
PDBBind v2016 和 PDBBind v2020 基準測試中蛋白質-配體結合親和力預測的效能統計摘要。 (資料來源:論文)
具體而言: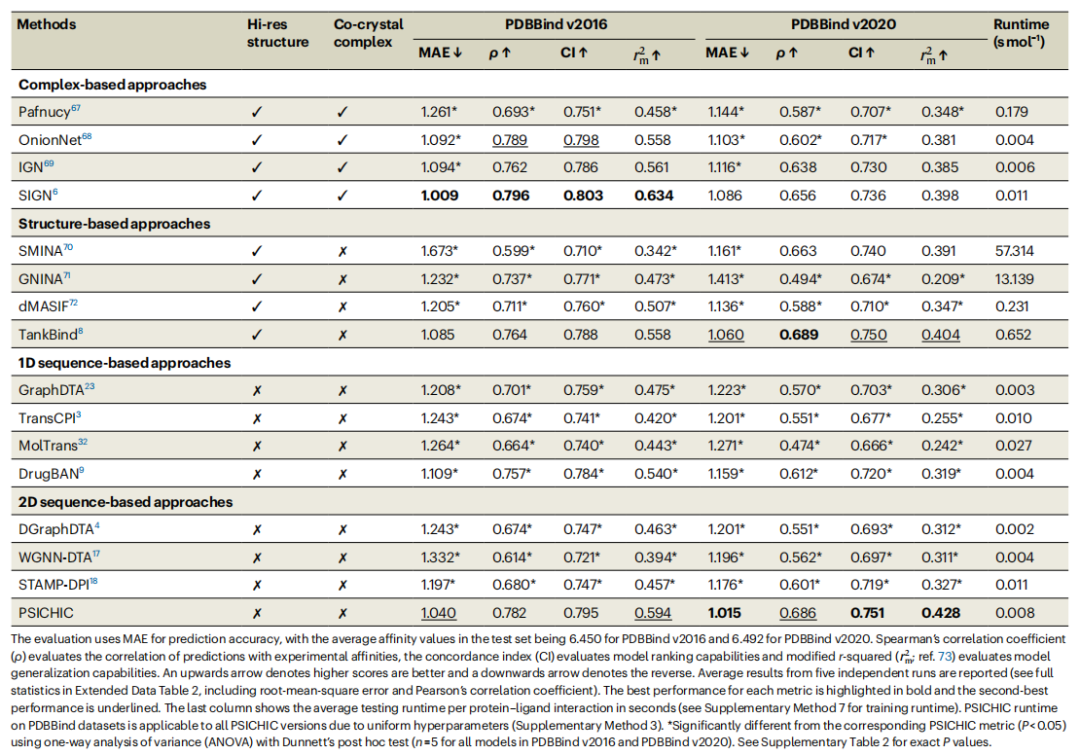
此外:
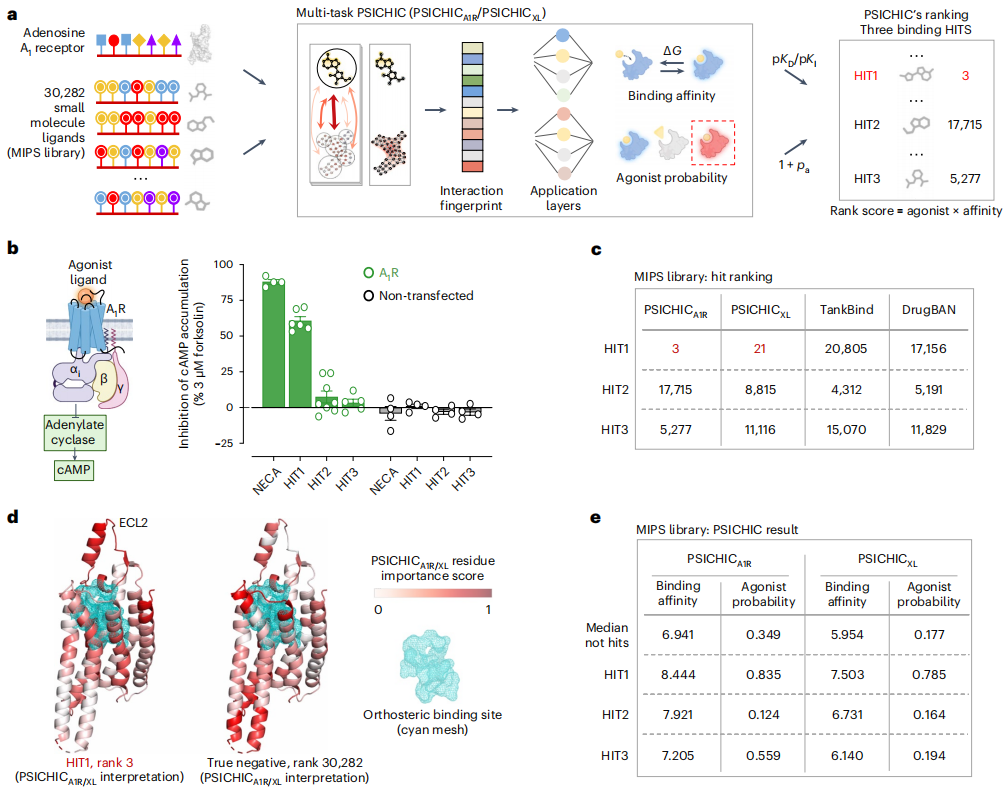
有趣的是,PSICHIC 的可解釋指紋表明,它獲得了僅從序列數據解碼蛋白質-配體相互作用的潛在機制、識別結合位點蛋白質殘基和所涉及的配體原子的能力,即使僅在具有結合親和力標籤而沒有相互作用資訊的序列資料上進行訓練也是如此。
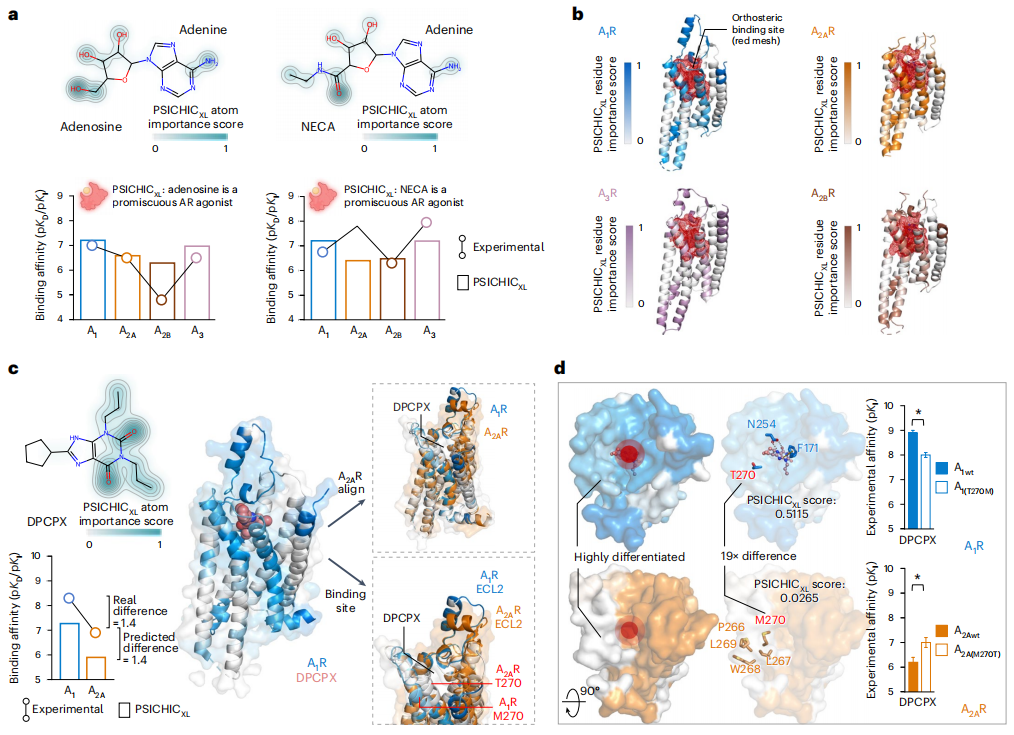
未來展望
以上是Nature子刊,準確率達96%,AI從序列預測蛋白-配體互作的詳細內容。更多資訊請關注PHP中文網其他相關文章!




