分子描述符的應用與挑戰
分子描述符廣泛應用於分子建模。然而,在 AI 輔助分子發現領域,缺乏自然適用、完整且原始的分子表徵,影響模型性能和可解釋性。
t-SMILES 框架的提出
基於片段的多尺度分子表徵框架 t-SMILES 解決分子表徵問題。此框架使用 SMILES 類型的字串描述分子,支援序列模型作為生成模型。
t-SMILES 的程式碼演算法
t-SMILES 有三種程式碼演算法:TSSA、TSDY 和 TSID。
實驗結果
實驗表明,t-SMILES 模型生成分子具有 100% 理論有效性和高新穎性,優於基於 SOTA SMILES 的模型。
此外,t-SMILES 模型避免過擬合,在標記的低資源資料集上保持相似性,同時實現更高新穎性。
發表訊息
研究以「t-SMILES: a fragment-based molecular representation framework for de novo ligand design」為題,於 6 月 11 日發表在《》上。
 論文連結:https://www.nature.com/articles/s41467-024-49388-6
論文連結:https://www.nature.com/articles/s41467-024-49388-6

新提出的 t-SMILES 框架
與 SMILES 相比
t-SMILES 僅引入了兩個新符號“&”和“^”,編碼多尺度和分層的分子拓撲。t-SMILES 演算法
提供了一個可擴展且適應性強的框架,理論上能夠支持廣泛的子結構方案。基於 t-SMILES 的模型
能夠在處理詳細子結構資訊的同時學習高階拓撲結構資訊。多程式碼系統
t-SMILES 演算法可以建立一個用於分子描述的多程式碼系統,其中: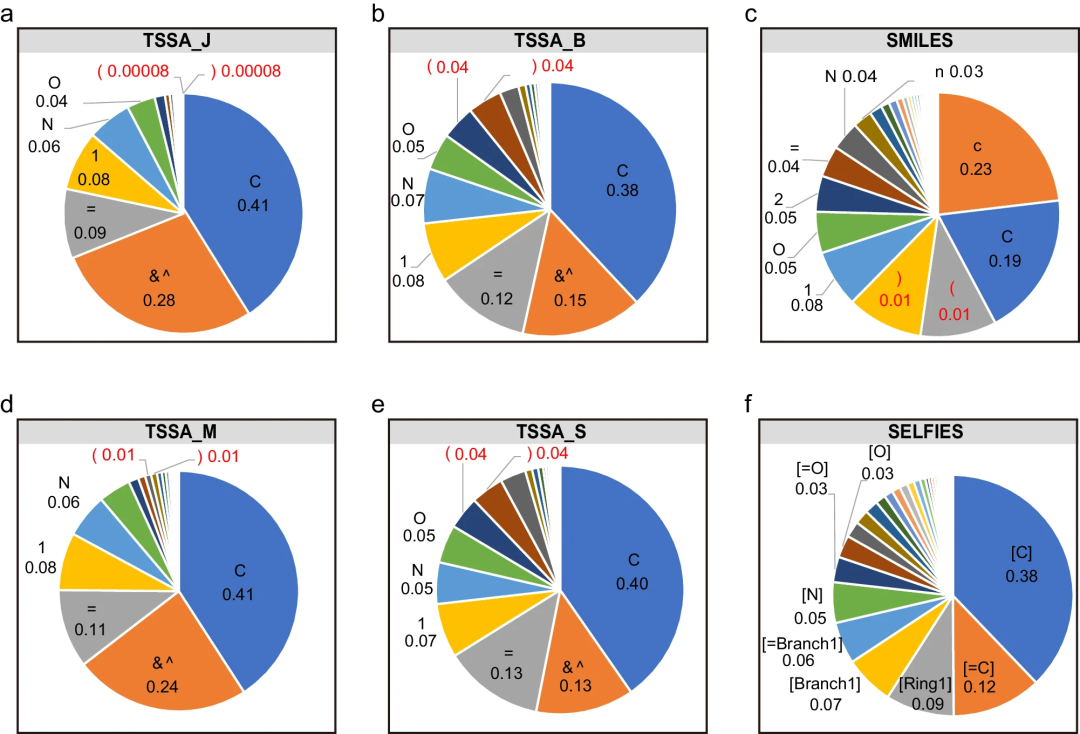 圖示:TSSA 代碼、SMILES 和 SELFIES 的 tokens 分佈。 (資料來源:論文)
圖示:TSSA 代碼、SMILES 和 SELFIES 的 tokens 分佈。 (資料來源:論文)
最後,進行了消融研究,以確認基於帶重建的 SMILES 的生成模型的有效性。為了評估 t-SMILES 演算法的適應性和靈活性,使用了四種先前發表的碎片演算法來分解分子,包括 JTVAE、BRICS、MMPA 和 Scaffold。不同實驗採用了三種指標:分佈學習基準、目標導向基準和物理化學性質的 Wasserstein 距離指標。
詳細的對比實驗表明,t-SMILES 模型產生的新分子 100% 理論有效,優於基於 SOTA SMILES 的模型。與 SMILES、DSMILES 和 SELFIES 相比,t-SMILES 的整體解決方案可以避免過擬合問題,並顯著提高低資源資料集上的平衡性能,無論是使用資料增強還是預訓練然後微調的模型。
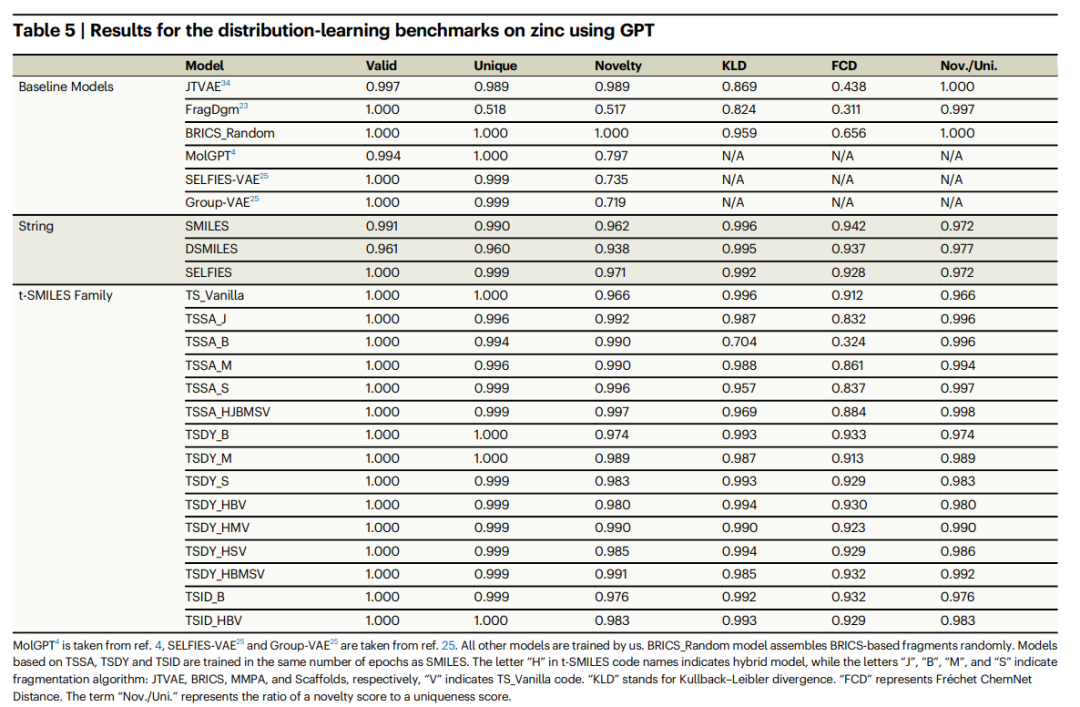
此外,t-SMILES 模型能夠熟練地捕捉分子的物理化學性質,確保產生的分子與訓練分子分佈保持相似性。與現有的基於片段和基於圖的基線模型相比,這顯著提高了效能。特別是,具有目標導向重建演算法的 t-SMILES 模型在目標導向的任務中比 SMILES、DSMILES、SELFIES 和 SOTA CReM 表現出明顯的優勢。
局限性和有待改進之處
註:封面來自網路
以上是分子100%有效,從頭設計配體,湖南大學提出以片段為基礎的分子表徵框架的詳細內容。更多資訊請關注PHP中文網其他相關文章!




